5 Créer des rapports dynamiques
“You say yourself it wasn’t reproducible. So it could have been anything that “crashed” your R, cosmic radiation, a bolt of lightning reversing a bit in your computer memory, …" — M. Maechler
Nous allons cette fois suivre directement ce module à partir du site de la formation. Vous allez faire une série de copier-collers pour préparer les rapports dynamiques. Ceux-ci sont formatés pour être utilisables à partir de la page web (et pas à partir du fichier source) en les sélectionnant à la souris, puis en les copiant dans vos rapports.
5.1 Vue d’ensemble
R n’est en fin de compte qu’un moteur de statistiques… Mais sa flexibilité est sans pareille, et permet des usages du moteur tout à fait inédits. Un des usages est de pouvoir générer des documents à partir de fichiers sources contenant du code R, exécuté au cours de la génération. Cet usage permet une reproductibilité totale du produit final — comme le dit si bien Lawrence Lessig : “Code is law.”
À la base de cette approche se trouve le langage Markdown, et son extension pour
R appelée tout simplement R Markdown. Le langage
Markdown est un langage de
balisage léger avec une syntaxe certe limitée, mais créée dans le but d’être
facile à utiliser. Le langage markdown est ultra simplifié, on ne peut pas tout
faire avec, mais il suffit dans la très grande majorité des cas : on peut
notamment mettre en forme (italique, gras, barré), ajouter des
sections, des listes (ordonnées ou non), des tableaux et des figures, etc. Avec
R Markdown, nous pouvons y intégrer des bouts
de code R, dont l’exécution fournira tout le matériel nécessaire à nos
documents.
C’est ce que nous allons voir dans ce module à l’aide du package rmarkdown,
afin de produire d’une part des documents imprimables statiques (au format PDF),
et d’autre part des documents web interactifs (au format HTML). Au fait, ne vous
laissez pas impressionner, vous utilisez des fichiers R Markdown depuis le début
de cette formation (fichiers .Rmd) ! En fait, le site web de la formation est
lui-même entièrement généré avec des fichiers RMarkdown.
Encore une fiche de synthèse indispensable, mais toutefois assez complexe, pour le package rmarkdown (elle présente entre autres la syntaxe markdown). Elle est disponible en cliquant sur la vignette ci-dessous (en anglais) :
Deux autres ressources notables sont à signaler, sous la forme de livres web (en anglais) :
Pour avoir un guide en français, on pourra se référer à ce tutorial.
5.2 Documents imprimables statiques : PDF
5.2.1 Premiers pas
Pour cette première étape, nous allons créer ittérativement un document PDF très
simple d’une page, en s’appuyant sur le jeu de données des iris disponible
sous R. Pour démarrer, il faut créer un nouveau document R Markdown à l’aide du
bouton + au tout début de la barre d’outils, puis sélectionner R Markdown.
Dans cette section, on remplira le titre du document (« Mon premier rapport PDF
»), l’auteur du document (vous-même), puis on choisira un format de sortie PDF
(Default Output Format: PDF).
RStudio nous mache un peu le travail en nous présentant un document R Markdown
minimal déjà tout prêt. On pourra tout de suite enregistrer ce nouveau document
dans rapport-pdf.Rmd. Avant de tout effacer pour y placer nos propres
informations et données, on peut prendre le temps d’y regarder d’un peu plus
près.
On peut y voir du texte, légèrement formaté, et des bouts de code R placés dans
des blocs de code ouverts et fermés par ``` : ce sont les bloc
(chunks). On y trouve à l’intérieur du code R tout à fait valide, celui-ci
sera entièrement exécuté pour générer le document. Voyons donc comment ce
faire : Il suffit d’appuyer sur le bouton Knit en-dessous de la barre des
onglets. Et voilà ! Un fichier PDF tout frais sorti de l’imprimerie.
La génération de documents PDF nécessite un environnement () fonctionnel. Si vous avez installé TinyTeX, vous pouvez vérifier l’installation avec la commande suivante (sous R), qui devrait retourner TRUE :
tinytex::is_tinytex()
Si malgré cela, vous avez toujours des problèmes (par exemple, l’erreur Error: pandoc document conversion failed with error 1, vous êtes peut-être sur une infrastructure réseau qui crée des soucis.)
XXX
5.2.2 Code R et sorties R
Maintenant que l’on sait générer un PDF, on va essayer de faire le notre, en
nettoyant un peu le document final. Première étape, on efface tout à partir de
la section ## R Markdown (y compris le titre de section), et on rajoute ceci :
## La taille des sépales d'iris
Ceci est document *R Markdown*. Il utilise donc la **syntaxe Markdown**, ce qui
permet quelques effets de style. On peut insérer du code R, qui sera exécuté au
moment de générer le rapport :
```{r iris-sepales}
library("dplyr")
iris %>%
group_by(Species) %>%
summarise(n = n(),
Sepal.Length.mean = mean(Sepal.Length),
Sepal.Length.sd = sd(Sepal.Length),
Sepal.Length.max = max(Sepal.Length),
) -> info
knitr::kable(info,
col.names = c("Espèce", "Effectif", "Moyenne", "Écart-type", "Maximum"),
align = "lcrrr",
digits = 3,
caption = "Résumé statistique des longueurs de sépales d'iris.")
```On obtient un PDF avec un joli tableau, mais couvrez donc ce code R, que je ne
saurais voir ! Rien de plus simple. On peut le faire individuellement, par
bloc, ou bien globalement, avec l’option echo = FALSE. Ici, on le fera de
manière globale dans la fonction knitr::opts_chunk$set du premier bloc. On en
profite pour ajouter message = FALSE pour enlever également les messages de R,
notamment au chargement des packages. On sauvegarde et on génère le PDF à
nouveau.
Le package knitr, qui s’occupe de l’exécution du code R en sous-main, permet
de formater joliment les résultats tabulaires de R avec la fonction
kable(). Parmi les options, on trouve :
col.namespour renommer les noms de colonnes ;alignpour ajuster l’alignement au sein de chaque colonne,lpour un alignement à gauche (left),rà droite (right) etcpour centrer (center) ;digitspour spécifier le nombre maximal de chiffres après la virgule ;captionpour donner un titre au tableau (et le numéroter).
On commence à y voir plus clair. Notez que l’on peut aussi ajouter des sorties R
au sein du text à l’aide de fragments de code de la forme
`r <du code>`. Ajoutons ceci à la fin du document :
On peut faire des maths et ajouter des fragments de code R : $\sqrt{2} \simeq
`r round(sqrt(2), 4)`$.On enregistre et génère le document à nouveau. On vient de voir que l’on peut
même faire des maths avec de jolies équations et autres symboles mathématiques !
Tout ce qui se trouve entre deux signes dollar est interprété selon la syntaxe
mathématique de \(\LaTeX\), comme par exemple ici la racine de 2 grâce à
$\sqrt{2}$.
5.2.3 Et des graphiques ggplot2 ?
Bien sûr ! Les graphiques de base ne sont évidemment pas les seuls à pouvoir être intégrés, et la marche-à-suivre est exactement la même. On rajoute maintenant ceci à la fin du document :
## Oui mais les pétales ?
On peut évidemment ajouter des graphiques, par exemple des sorties `ggplot2` :
```{r iris-ggplot, out.width = "70%", fig.align = "center", fig.cap = "Longueur des pétales en fonction de la longueur des sépales."}
library("ggplot2")
ggplot(iris, aes(Sepal.Length, Petal.Length, color = Species)) +
geom_point() +
theme_classic()
```On sauvegarde, puis on génère le PDF. Et voilà, tout y est ! Parmi les options du bloc, on en retrouve plusieurs liées aux graphiques :
out.widthdéfinit la largeur du graphique (en fonction de la largeur disponible dans la page ou bien selon une unité de mesure telle que le cm) ;fig.alignpermet de centrer le graphique (aligné à gauche par défaut) ;fig.cap, à l’instar decaptionpour les tableaux, permet de donner un titre au graphique (et le numéroter).
5.2.4 Pour aller plus loin
Les possibilités du format PDF sont multiples : on peut par exemple avoir une mise en page avancée, inclure une table des matières, travailler sur la base de modèles (templates), etc. En toile de fond, c’est \(\LaTeX\) qui fait le sale boulot : à peu près tout ce qui est possible avec \(\LaTeX\) est accessible d’une manière ou d’une autre via R Markdown au format PDF.
Note : Pour les aventureux, on peut aussi exporter en document Word (word_document dans l’en-tête) ! Ce format n’est pas parfaitement pris en charge, et certaines options et formatages ne seront pas correctement appliqués.
Il existe toutefois un package additionnel, officedown, qui ajoute tout un tas de fonctionnalités pour exporter en document Word, notamment dans la mise-en-forme. Un avant-goût des possibilités est visible sur le site du package.
5.3 Documents web interactifs : HTML
La démarche pour obtenir une page web est fondamentalement la même que ce qu’on
a vu jusqu’ici, mais elle permet en revanche de bénéficier des fonctionnalités
web modernes, notamment au niveau de l’interactivité. Pour commencer, on crée un
nouveau document R Markdown que l’on appelera « Document archi-confidentiel »,
avec cette fois un format de sortie en HTML, sauvegardé dans
rapport-html.Rmd. Comme précédemment, RStudio nous prend par la main et nous
propose le même exemple, mais cette fois en HTML. Différence notable :
html_document dans l’en-tête.
Pour voir de quoi il en retourner, on génère la page web, encore avec le bouton
Knit en-dessous de la barre des onglets. Le résultat est une page web propre
avec du code R, et des sorties tabulaires et graphiques. Allons un peu plus
loin.
Note : On peut ensuite publier le fichier HTML résultant sur n’importe quel serveur. Si vous n’avez pas accès à votre propre serveur (ou celui de l’université), vous pouvez utiliser le service RPubs pour publier des rapports HTML. La marche à suivre :
- Une fois le rapport généré, cliquer sur le bouton « Publish » dans la barre d’outils du Viewer ;
-
Accepter l’installation des packages (
packrat,rsconnect) ; - Dans la fenêtre qui s’ouvre (« Publish to »), choisir « RPubs », puis « Publish » ;
- Si besoin, créer un nouveau compte (« Create an account ») ou bien se connecter à un compte existant ;
- Ajouter un titre, une description (optionnel), et spécifier l’URL (optionnel).
Attention ! Toute publication dans RPubs est entièrement publique et accessible par tout le monde ! À utiliser avec précaution.
5.3.1 L’en-tête YAML et les options knitr
Sous ce barbarisme se cache le morceau tout en haut du fichier, entre les tirets
---. Celui-ci permet de définir plusieurs options globales concernant le
document. On va modifier l’output ainsi :
output:
html_document:
toc: true
toc_float: true
code_download: true
self_contained: true
theme: flatlyVoici ces options dans le détail :
toc: truepour rajouter une table des matières (table of contents) ;toc_float: truepour avoir cette table des matières en menu flottant toujours en haut de la page (très pratique, mais ne fonctionne pas dans le Viewer de RStudio) ;code_download: truepour ajouter un bouton permettant de télécharger le fichier R Markdown (à ne pas utiliser si l’on ne veut pas partager son code !) ;self_contained: truepour que le fichier HTML résultant contiennt toute l’information nécessaire (très pratique aussi pour partager ; nous allons faire rapidement appel à de nombreuses libraires qui seraient autrement placées dans des sous-dossiers) ;theme: flatlypour utiliser le thèmeflatly, qui est plutôt réussi. Plusieurs thèmes existent, et tout est évidemment entièrement ajustable si on le souhaite vraiment.
Juste en-dessous de l’en-tête, on trouve le premier bloc qui permet de
spécifier les options de knitr :
```{r setup, include = FALSE}On peut en replacer le contenu avec les options suivantes et regarder le résultat :
knitr::opts_chunk$set(
cache = FALSE, # Gère l'utilisation du cache pour l'exécution du code (TRUE/FALSE)
error = FALSE, # Doit-on arrêter l'exécution dès la première erreur ? (TRUE/FALSE)
echo = FALSE, # Gère l'affichage du code (TRUE/FALSE)
warning = FALSE, # Gère l'affichage des warnings de R (TRUE/FALSE)
message = FALSE # Gère l'affichage des messages de R (TRUE/FALSE)
)La première option en particulier est intéressante, puisqu’elle permet de mettre
les blocs de code R en cache (cache = TRUE), et donc ne pas les réexécuter
s’ils n’ont pas été modifiés. Quand on a des étapes assez lourdes, le gain de
temps est significatif. (à tester à la maison !)
5.3.2 Retour à nos données : des étudiants et des donuts
Comme précédemment, on efface tout à partir de la section ## R Markdown pour
recommencer avec nos propres informations, et on charge le fichier .RData dans
le premier bloc :
## Un donut d'étudiants
```{r load}
load("data/um18.RData")
```En plus de la fonction kable() du package knitr, rmarkdown propose une
deuxième façon de visualiser des données tabulaires, cette fois en permettant
d’afficher le tableau en entier (plus précisément toutes les colonnes et les
1000 premières lignes par défaut), via la fonction paged_table(). On peut
notamment y régler le nombre de lignes à conserver, que je fixe ici à 100 pour
gagner en légèreté :
On travaille sur les données d'inscriptions suivantes :
```{r data}
rmarkdown::paged_table(um18, options = list(max.print = 100))
```Exercice 1 : Dans cet exercice, vous avez trois tâches à accomplir :
- Ajouter un peu de texte, avec un minimum de mise-en-forme, expliquant ce que l’on fait ;
- Préparer sous R les effectifs de chaque type de bac, et les inclure dans un joli tableau ;
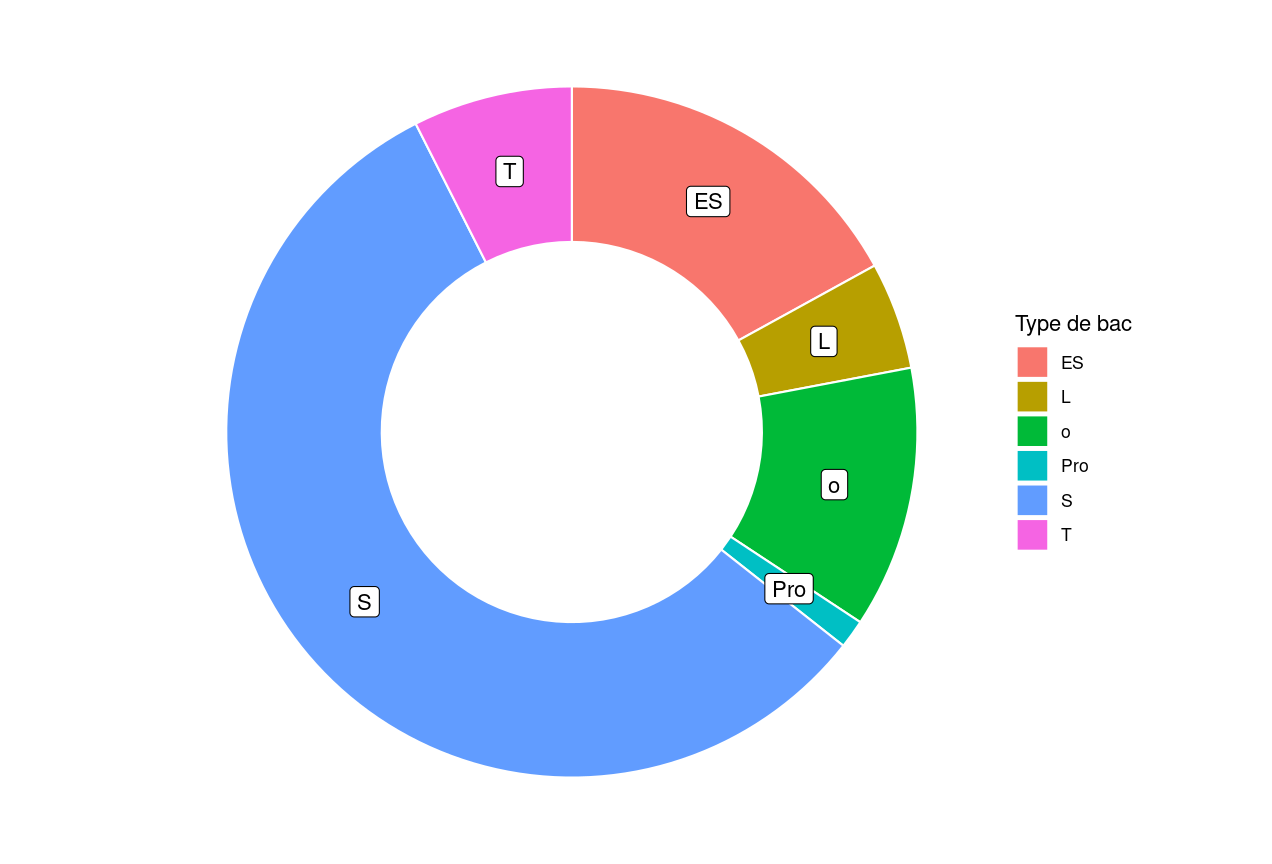
- Inclure le donut des types de bac, tel que préparé dans le module de dataviz :
5.3.3 Des diagrammes programmatiques avec nomnoml
La librairie nomnoml (et le package R du même nom) permettent de créer des
diagrammes de manière programmatique. En clair, une fois le package chargé
(library("nomnoml")), on peut créer des diagrammes par le biais de nouveaux
blocs de type "nomnoml" :
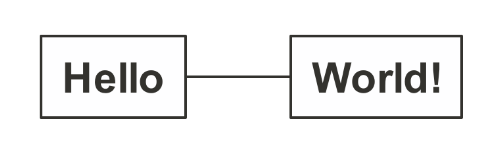
```{nomnoml}
[Hello]-[World!]
```Qui produira le diagramme suivant :
Mais pour que ça colle un peu mieux à nos données, nous intégrerons le diagramme suivant (à ajouter à la fin du document R Markdown avant de générer le document web à nouveau) :
## Des effectifs ventilés par domaine et mention
### Un diagramme explicatif
```{r nomnoml}
library("nomnoml")
```
```{nomnoml}
#stroke: blue
#.box: fill=#a8caff dashed visual=ellipse
#.circ: fill=#3b547a stroke=white visual=ellipse
[Effectif]-[<box>Domaine]
[Domaine]-[<circ>Mention]
```Une option sympatique est l’export du diagramme en SVG plutôt qu’en PNG. Le SVG
est format web natif, et à l’instar du PDF, propose une image vectorielle de
qualité parfaite. Le SVG est disponible via l’option de bloc nomnoml svg = TRUE.
Pour aller plus loin, on pourra jeter un œil sur le site du package ou bien directement sur celui de la libraire sous-jacente.
À noter : L’intégration avec R Markdown est parfois un peu capricieuse. On pourrait avoir besoin de la libraire additionnelle Phantom.JS… Pour cela, on l’installe via R :
install.packages("webshot")
webshot::install_phantomjs()Il peut également y avoir des problèmes liés au cache, qu’il faudra parfois supprimer avant de relancer la génération de la page web. Et on croise les doigts pour que ça fonctionne.
5.3.4 Des graphiques interactifs avec plotly
Plotly.js est une fantastique librairie JavaScript pour créer des graphiques
et tableaux de bord interactifs. Comme il se doit, il existe un package R pour
pouvoir l’utiliser : plotly. Celle-ci permet à la fois de récupérer des
graphiques R déjà préparés, ou bien d’en créer d’autres avec (malheureusement)
une syntaxe qui lui est propre.
On commence donc par intégrer un graphique ggplot2. Pour cela, il suffit
d’enregistrer la sortie de ggplot() dans un objet R, puis de l’utiliser
directement comme paramètre dans la fonction ggplotly(). Par exemple :
### Un graphique `ggplot2` dynamique
```{r plotly-ggplot}
library("plotly")
ggplot(um18, aes(x = bac_regroup, y = prem_insc_univ_fr, color = bac_regroup)) +
geom_boxplot() +
ylim(1980, 2020) +
theme_classic() +
labs(title = "Quelle est l'année de première inscription à l'université, selon la filière du bac ?",
subtitle = "(données pour les inscriptions à l'Université de Montpellier 2018–2019)",
x = "Type de baccalauréat",
y = "Année (tronquée à partir de 1980") +
theme(legend.position = "none",
axis.text.x = element_text(angle = -45, vjust = 0.1, hjust = 0.1)) -> gg
ggplotly(gg)
```Notez que j’ai volontairement enlevé le geom_jitter() utilisé précédemment…
sans quoi la sortie serait excessivement lourde pour présenter interactivement
chaque point correspondant à un étudiant !
5.3.5 Le soleil explose…
Les diagrammes sunburst permettent de visualiser des données hiérarchiques
organisées dans des cercles concentriques, comme un donut sur plusieurs niveaux.
Il est très utile pour voir des effectifs rangés en classes et sous-classes par
exemple. Nous allons donc inclure un diagramme sunburst dans le rapport via le
package plotly (avec sa syntaxe propre). Voici un exemple :
### Le soleil explose…
```{r plotly-sunburst-test}
plot_ly(
type = "sunburst",
labels = c("A", "B", "C", "A1", "A2", "A3", "B1", "B2", "C1"),
parents = c("Par", "Par", "Par", "A", "A", "A", "B", "B", "C"),
values = c(30, 20, 10, 10, 6, 4, 10, 4, 3),
branchvalues = "total"
)
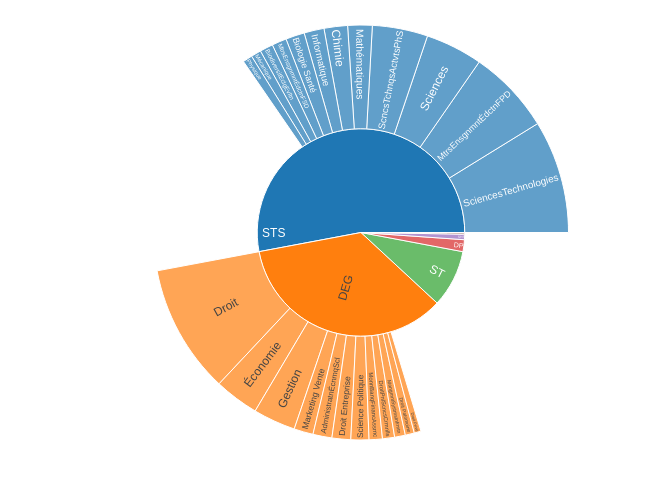
```Exercice 2 : Dans cet exercice, on va rajouter un diagramme sunburst présentant les effectifs par domaine disciplinaire et par mention, comme ci-dessous :
Quelques indications :
- Il faut retrouver la syntaxe « étiquettes–parents » présentée dans l’exemple, en l’appliquant aux domaines disciplinaires et aux mentions. Il faudra préparer les données en amont, en comptant d’abord les domaines, puis les mentions. Pour plus de facilité, on préparera les données séparément pour chaque niveau (“cercle”) de données : domaines disciplinaires puis mentions ;
- On pourra limiter l’affichage aux mentions avec au moins 200 inscrits ;
- On utilisera les domaines et mentions abrégés ;
-
On peut avoir un parent vide
""(qui ne s’affiche alors pas), mais il faut néanmoins toujours spécifier le parent ; -
On rajoutera l’argument
insidetextorientation = “radial”pour optimiser l’orientation du texte dans les quartiers.
5.3.6 … et les étudiants coulent !
Les diagrammes de Sankey (ou diagrammes alluviaux) sont une manière efficace de
présenter des flux entre classes à différentes étapes (par exemple au cours du
temps). Encore une fois, un graphique vaut mieux qu’un long discours, on teste
donc un diagramme de Sankey avec le package plotly (encore une fois avec sa
syntaxe propre) :
### … et les étudiants coulent !
```{r plotly-sankey-test}
plot_ly(
type = "sankey",
node = list( # Liste des nœuds
# Index 0 1 2 3 4 5 6 7
label = c("A1", "A2", "B1", "B2", "B1", "B2", "A1", "A2") # Étiquettes
),
link = list( # Liste des flux (nombre différent des nœuds)
source = c(0, 0, 1,1,2,2,3,3,4,5), # Index des nœuds sources (à partir de 0)
target = c(2, 3, 4,5,6,7,6,7,6,6), # Index des nœuds cibles (à partir de 0)
value = c(12,10,8,3,5,5,7,1,1,3) # Valeur du flux
)
)
```La clé ici est de considérer le diagramme comme des flux (liens, en anglais links) qui connectent des nœuds (nodes). La valeur des flux en précise la largeur, et chaque flux relie un nœud source (source) à un nœud cible (target). À noter que l’index des nœuds commence à 0. La liste des nœuds permet de préciser les étiquettes collées à chacun d’entre eux.
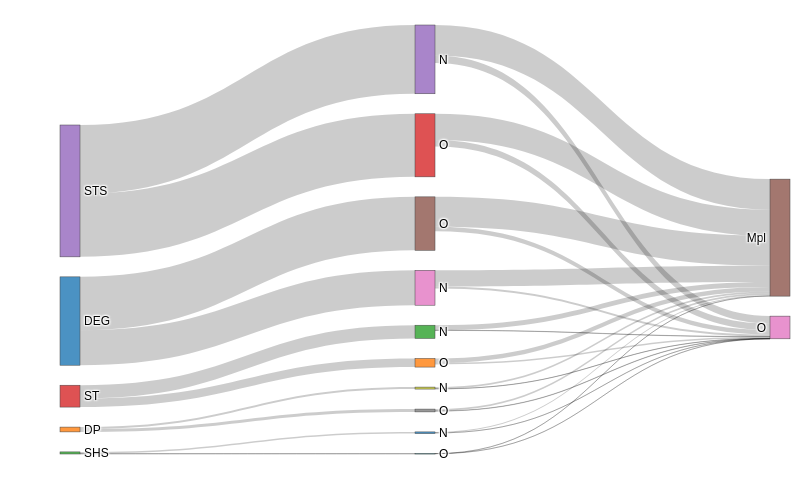
Exercice 3 : Dans cet exercice, on va rajouter un diagramme de Sankey présentant la réussite des étudiants par domaine disciplinaire, puis de savoir s’ils sont restés sur l’académie de Montpellier ou non, comme ci-dessous :
Quelques indications :
-
Il faut dans un premier temps créer une nouvelle variable
$montpellierqui prendra la valeurTRUEsi l’académie de 2019 (ins19_acaeta) est d’une valeur de 11,FALSEdans tous les autrescas. Regarder pour cela la fonctionifelse(); -
Dans les opérations qui suivent, on excluera le domaine
“ALL”qui comporte des effectifs trop faibles ; -
On comptera alors la réussite (
temoin_dip) par domaine disciplinaire (abrégé), avant de recompter les étudiants partis ou restés (montpellier) selon le couple domaine disciplinaire/réussite ; - On reliera ces valeurs de comptage selon la logique de flux déterminée sur nos 3 variables. Bien numéroter les nœuds permet d’y voir vraiment plus clair.
