2 Érable sycomore (Acer pseudoplatanus L.)
2.1 Taxonomie
On vérifie le statut taxonomique de l’érable sycomore sur l’ossature de taxonomie du GBIF :
[1] "Acer pseudoplatanus"# A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Acer pseudoplatanus L. ACCEPTED FALSE Acer pseudoplatanus 3189870 97 EXACT Pour l’érable sycomore, rien à signaler, c’est directement le nom scientifique accepté.
2.2 Données d’occurrences GBIF
Le Système mondial d’informations sur la biodiversité GBIF (Global Biodiversity Information Facility) est un projet scientifique international qui a pour but de mettre à disposition toute l’information connue sur la biodiversité, notamment les données d’observations ou de collections sur les animaux, plantes, champignons, bactéries et archaea. Ces données sont accessibles en ligne via le portail https://www.gbif.org/ depuis 2013.
Aparté sur les licences des données : Les données disponibles sur la plateforme du GBIF le sont selon 3 licences :
- CC0 : équivalent aussi proche que possible du domaine public, sans restriction ;
- CC BY : les données sont disponibles pour toute utilisation du moment que les sources sont créditées ;
- CC BY-NC : la même chose, mais ne permet pas d’utilisation commerciale des données.
La clause NC (non commerciale) est la plus problématique, et est largement
ambiguë, notamment sur les produits dérivés (comme il sera le cas dans cette
étude). On trouve notamment toute une discussion sur le sujet sur le Wiki
Creative
Commons. L’interprétation
la plus communément admise implique que la clause commerciale couvre également
les produits dérivés (Reusers may make NonCommercial uses only, even when
reusing NC material with other works.), même si ce n’est pas forcément
l’intention première des propriétaires des données.
Pour les données issues du GBIF, il sera donc obligatoire de n’utiliser que les données sous CC0 et CC BY, et d’enlever les données sous CC BY-NC.
Pour information, le contrat d’utilisation des données se trouve ici ; en cas d’utilisation des données, on citera le DOI du jeu de données téléchargés, ainsi que les différents jeux de données utilisés.
Les informations pour la citation sont disponibles ici.
La collecte de données du GBIF se fait en plusieurs étapes :
Préparer le jeu de données sur le serveur du GBIF. Note : il faut avoir spécifié au préalable ses identifiants GBIF pour la session R.
La préparation peut prendre du temps, et une fonction fait en sorte que R attende que le jeu de données soit prêt. Télécharger alors le jeu de données en local au format ZIP (on s’assurera de bien ranger tout ça avec un dossier par espèce, le nom de fichier ne le spécifiant pas) puis importer le jeu de données sous R à partir du fichier ZIP.
<<gbif download metadata>>
Status: SUCCEEDED
DOI: 10.15468/dl.6pzs3y
Format: SIMPLE_CSV
Download key: 0104676-230224095556074
Created: 2023-03-20T14:47:30.199+00:00
Modified: 2023-03-20T14:55:45.151+00:00
Download link: https://api.gbif.org/v1/occurrence/download/request/0104676-230224095556074.zip
Total records: 486584On peut dès lors explorer rapidement les données (classe du jeu de données et premières lignes du tableau) :
# A tibble: 486,584 × 50
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
2 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
3 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
4 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
5 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
6 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
7 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
8 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
9 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
10 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
# ℹ 486,574 more rows
# ℹ 40 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …Puis les transformer en objets géographiques (format Simple feature de sf) :
Simple feature collection with 486584 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -176.232734 ymin: -46.899969 xmax: 176.939614 ymax: 69.707067
Geodetic CRS: WGS 84
# A tibble: 486,584 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
2 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
3 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
4 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
5 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
6 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
7 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
8 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
9 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
10 1.88e-314 e707e6da-e143-445d-b41d-529c4… WA000005000… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
# ℹ 486,574 more rows
# ℹ 41 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
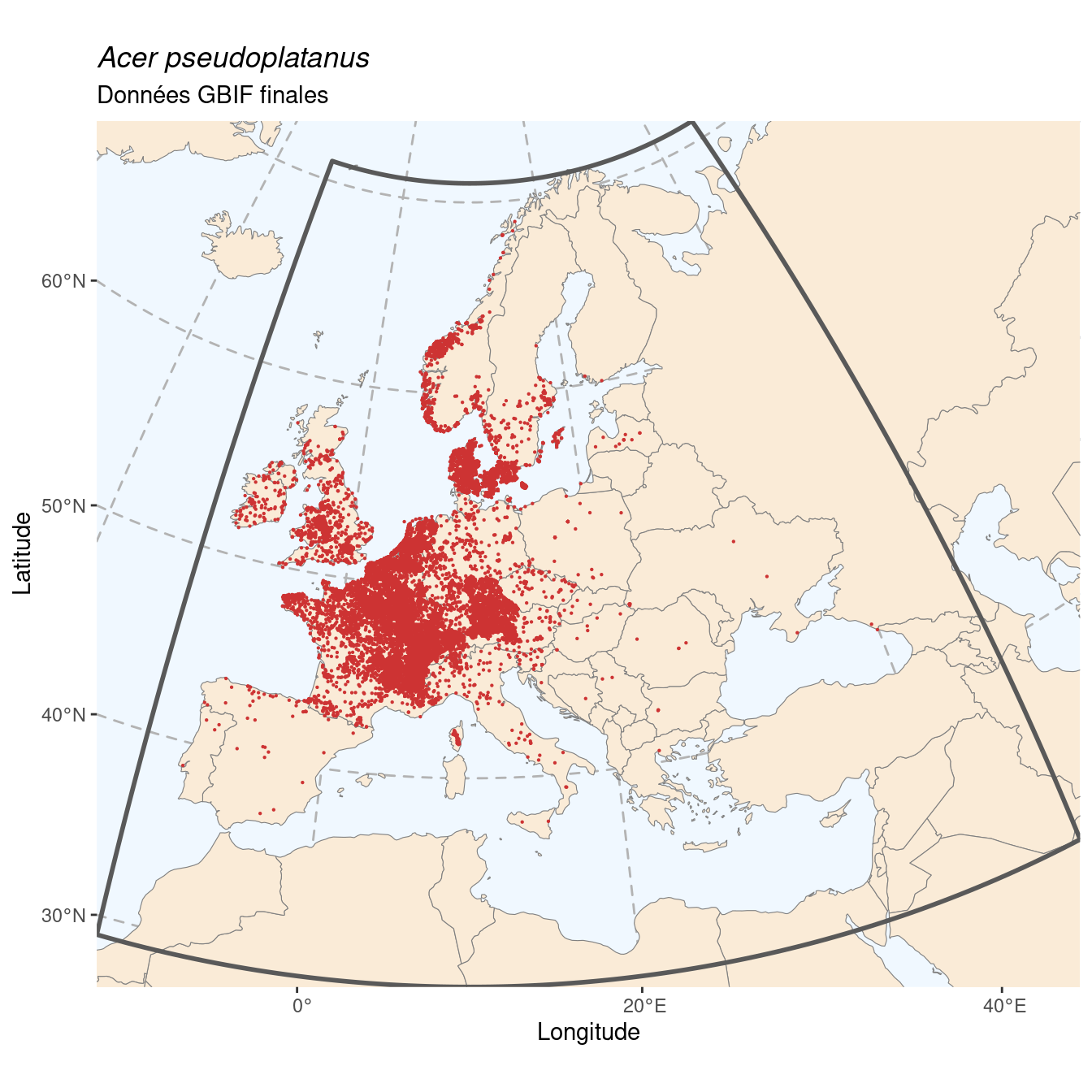
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …Il y a 486 584 occurrences dans le jeu de données. On les affiche sur la carte du monde, avec les 3 masques créés précédemment :
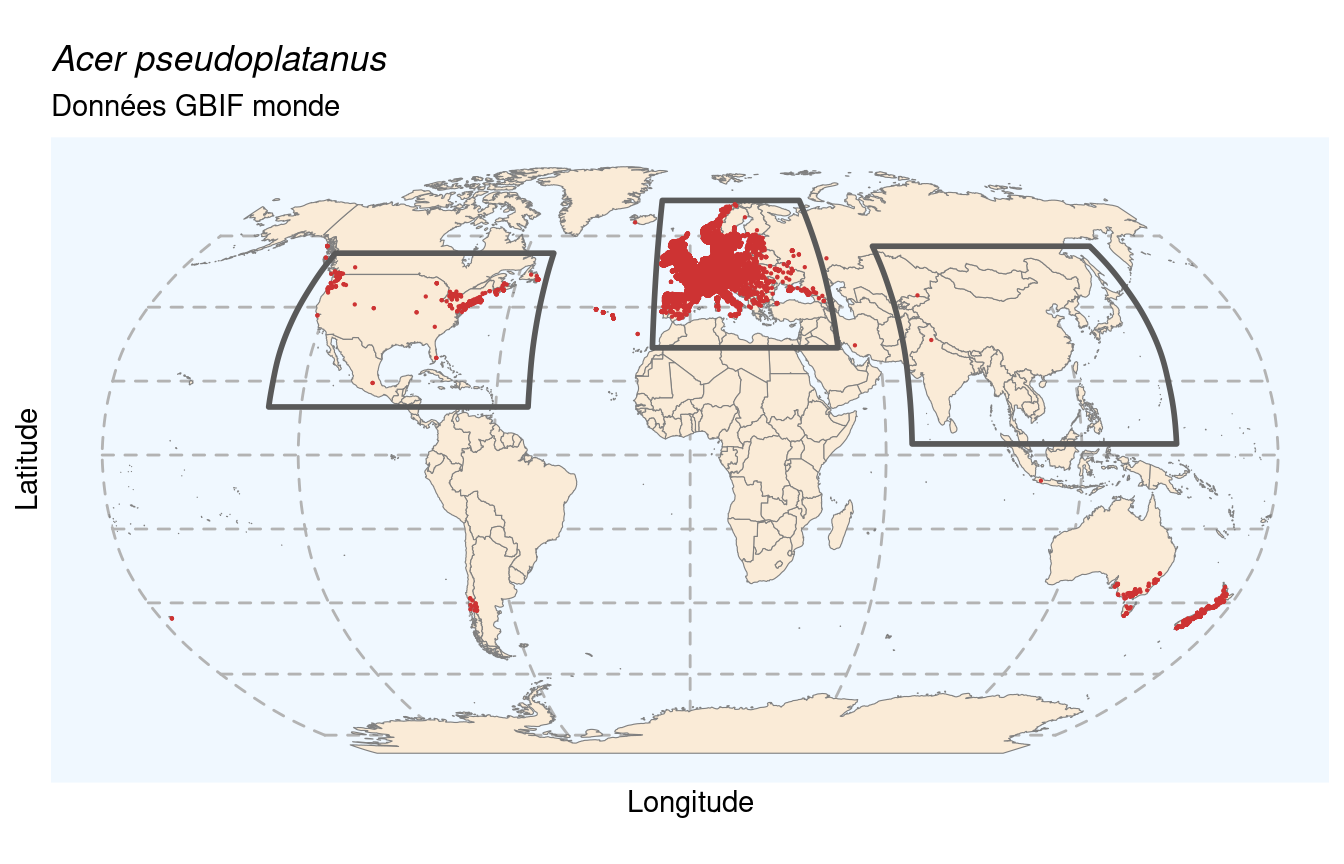
Figure 2.1: Occurrences de Acer pseudoplatanus dans le monde.
2.3 Données par région
On évalue la proportion d’occurrences dans chaque région, en utilisant les 3 masques créés précédemment. Si on a au moins 50 % des occurrences dans une région, on peut continuer en travaillant uniquement sur ces données. Sinon, on vérifie qu’au moins 67 % des données tombent dans l’ensemble des régions, et on prend la région la plus représentée. Au final, s’il s’agit d’une espèce endémique d’Europe, on n’a pas besoin de manipulation supplémentaire.
On calcule alors la proportion d’occurrences en Europe, en Amérique du Nord et en Asie pour déterminer l’endémisme de l’essence (défini si une région regroupe plus de la moitié des données).
[1] 0.9664744422[1] 0.02992083587[1] 4.110287227e-06On obtient 96.65 % d’occurrences en Europe, on est bien sur une espèce endémique de la région. On peut récupérer les données d’Europe, et les cartographier :
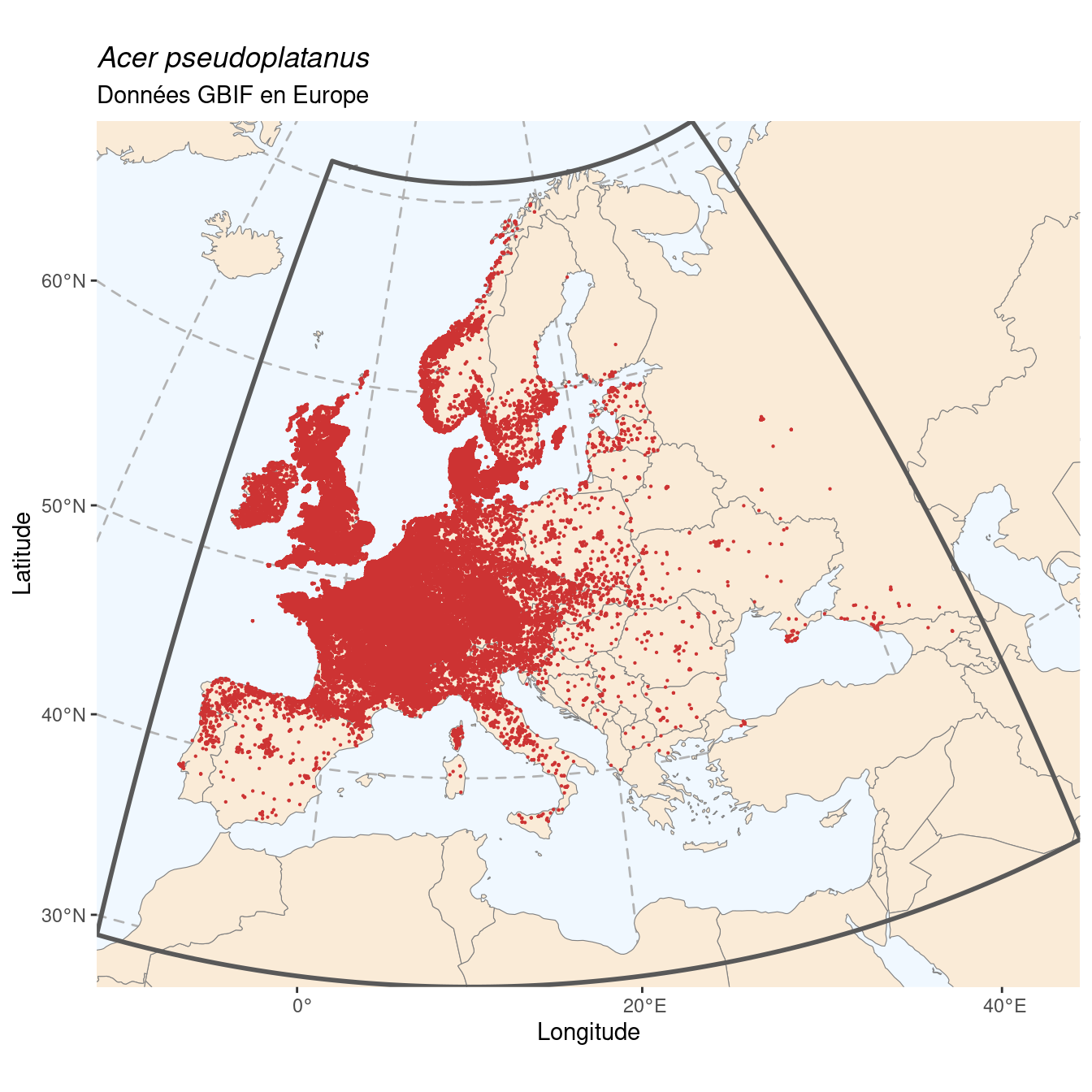
Figure 2.2: Occurrence de Acer pseudoplatanus en Europe.
2.3.1 Licences
On s’intéresse ici au problème particulier des licences :
CC_BY_4_0 CC_BY_NC_4_0 CC0_1_0
333579 107465 29227 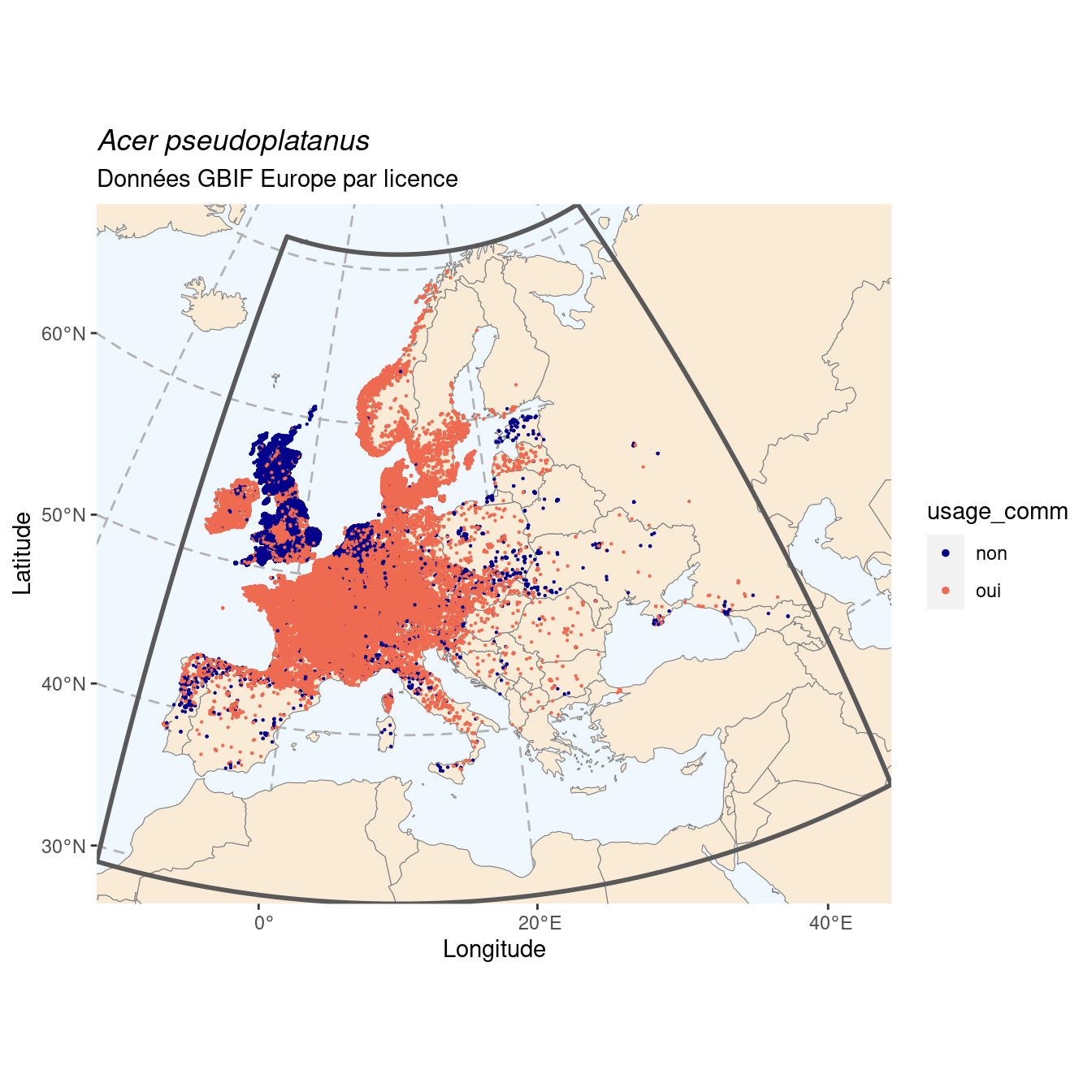
Figure 2.3: Occurrence de Acer pseudoplatanus en Europe, selon la licence : CC BY-NC en bleu ; CC0 et CC BY en orange.
Dans ce cas précis, même si le nombre d’occurrences en CC BY-NC reste très élevé, il est surtout limité à quelques jeux de données en Angleterre. Les omettre n’invalidera pas l’approche. On peut donc supprimer ces données sans risque.
2.3.2 Sous-échantillonnage
Sur les localisations en Europe, on sous-échantillonne les points pour descendre
à un nombre raisonnable (\(< 25k\) occurrences ; on ne sous-échantillonnera donc
pas si le jeu de données est déjà plus petit). Deux approches possibles : soit
on tire aléatoirement le nombre d’occurrences voulu, soit on procède à un
écrémage des données de manière stratifiée (fonction sample.grid() du package
GSIF que l’on importe ici ; celui-ci nécessite de convertir les données
d’occurrences au format SpatialPoints du package sp). La première approche a
l’avantage d’une reproductibilité complète et permet de préparer un jeu de
données d’évaluation indépendant dans la même étape ; la deuxième permet de
s’affranchir de biais d’échantillonnage trop criant en limitant le nombre
d’observations par unité de surface, mais en éliminant par la même occasion les
différences réelles d’abondance.
Simple feature collection with 25000 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -9.980495 ymin: 37.182608 xmax: 39.931355 ymax: 68.889571
Geodetic CRS: WGS 84
# A tibble: 25,000 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
<int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 5.44e-315 75956ee6-1a2b-4fa3-b3e8-ccda6… http://pifh… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
2 1.54e-314 83fdfd3d-3a25-4705-9fbe-3db1d… INFOFLORA-T… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
3 9.29e-315 64dabd3c-4f34-4520-b9dd-d227a… http://id.s… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
4 9.49e-315 8ea4250e-0ff0-44f8-812e-bffc3… http://id.s… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
5 1.42e-314 83fdfd3d-3a25-4705-9fbe-3db1d… INFOFLORA-T… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
6 1.95e-314 14d5676a-2c54-4f94-9023-1e8dc… q-102611580… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
7 1.94e-314 e13c8785-ca23-4aab-a30a-78807… f3389b5e-3d… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
8 1.24e-314 dd238f50-f594-4e51-82af-03322… 11b08a24-6d… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
9 5.48e-315 75956ee6-1a2b-4fa3-b3e8-ccda6… http://cbnb… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
10 9.51e-315 8ea4250e-0ff0-44f8-812e-bffc3… http://id.s… Plantae Trach… Magn… Sapi… Sapin… Acer Acer p…
# ℹ 24,990 more rows
# ℹ 41 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …class : SpatialPointsDataFrame
features : 20287
extent : -10.364399, 42.721557, 37.06, 68.508508 (xmin, xmax, ymin, ymax)
crs : +proj=longlat +datum=WGS84 +no_defs
variables : 50
names : gbifID, datasetKey, occurrenceID, kingdom, phylum, class, order, family, genus, species, infraspecificEpithet, taxonRank, scientificName, verbatimScientificName, verbatimScientificNameAuthorship, ...
min values : 1.9814403913334e-318, 014f3d14-a409-4090-b690-a10bd18e6e62, , Plantae, Tracheophyta, Magnoliopsida, Sapindales, Sapindaceae, Acer, Acer pseudoplatanus, , FORM, Acer pseudoplatanus f. pseudoplatanus, Acer psedoplatanus L., , ...
max values : 2.00594481220301e-314, fef99f8e-e00e-447a-9896-1ce15538e4fb, zlrgh6v5, Plantae, Tracheophyta, Magnoliopsida, Sapindales, Sapindaceae, Acer, Acer pseudoplatanus, villosum, VARIETY, SH1272204.09FU, SH1272204.09FU, L., ... 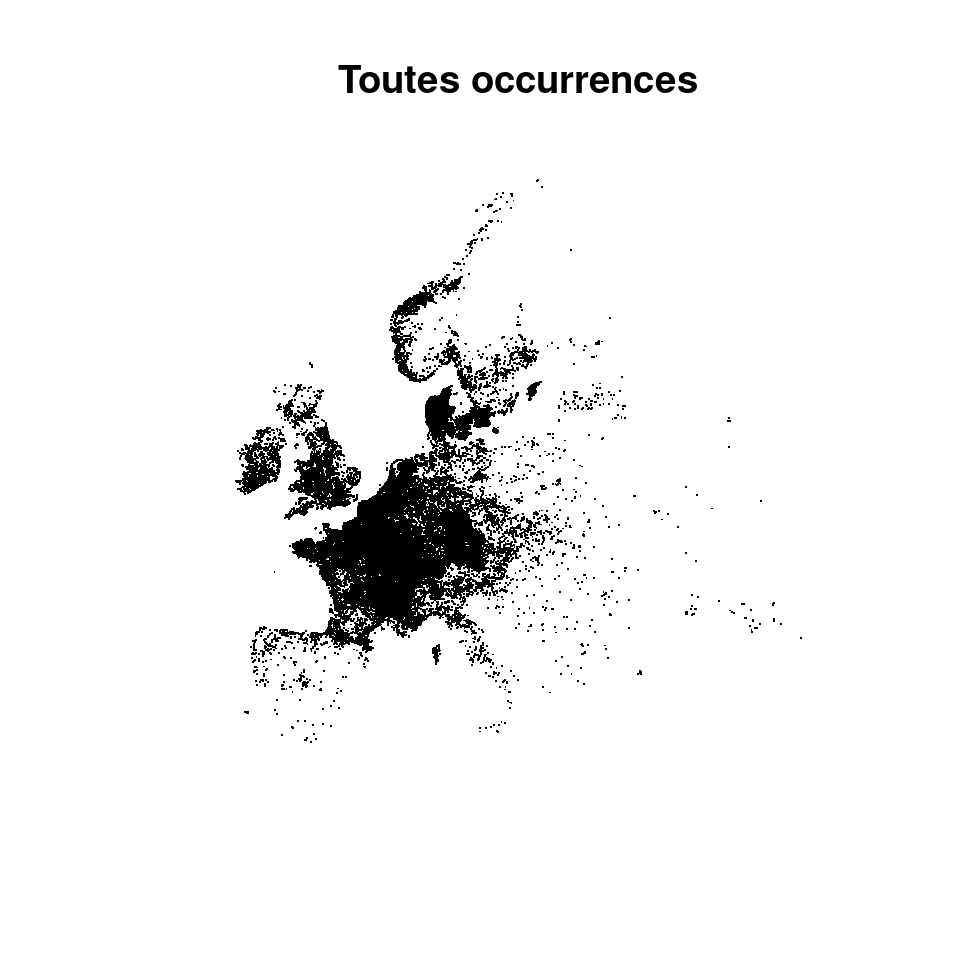
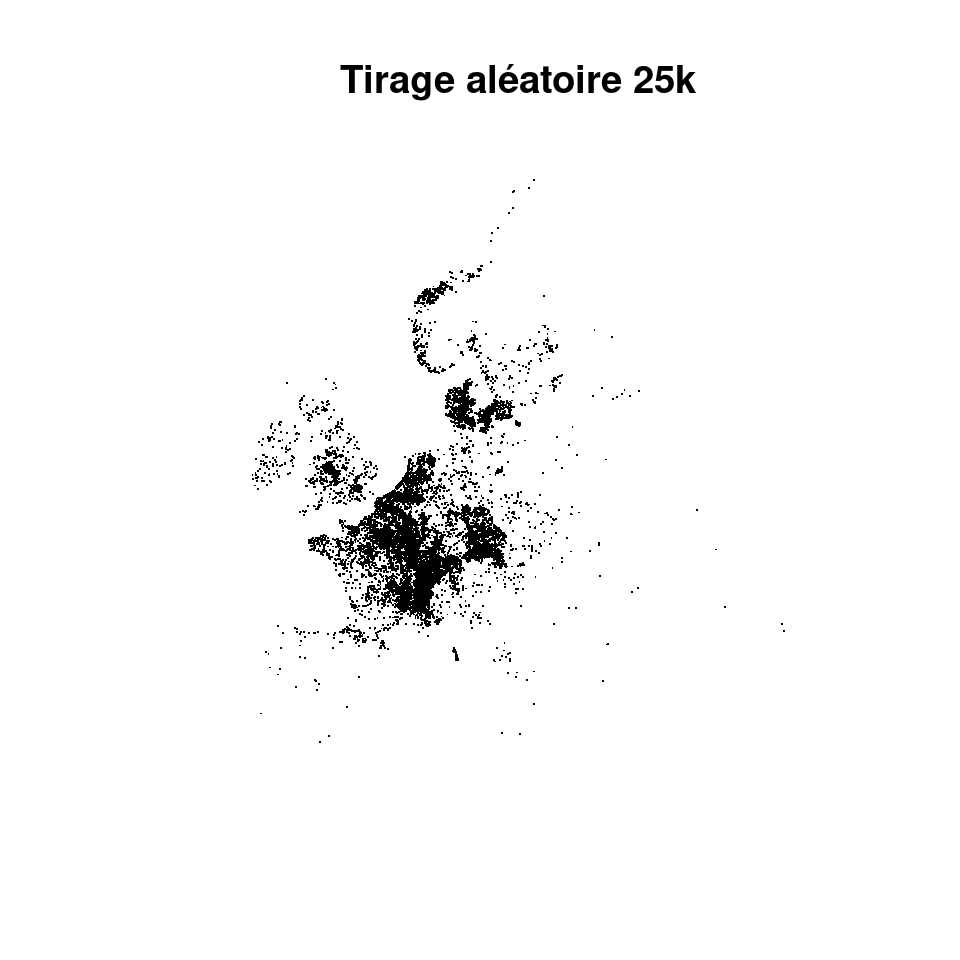
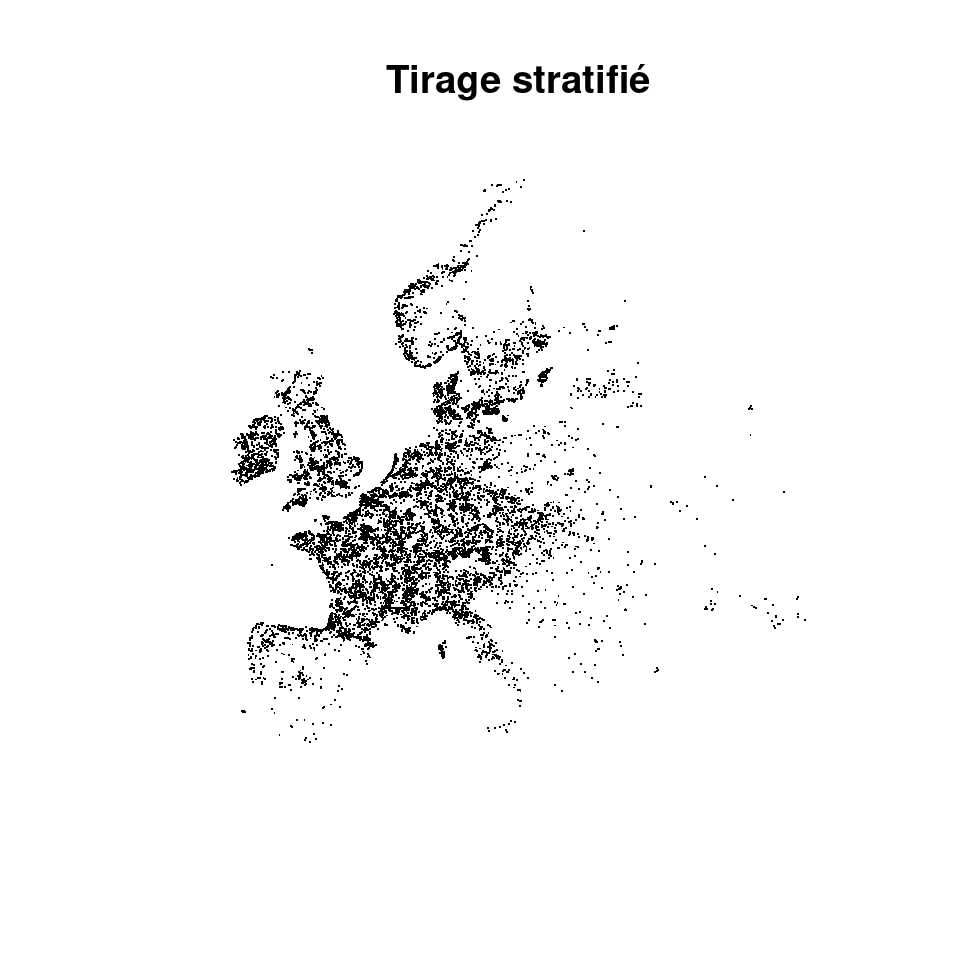
Figure 2.4: Échantillonnage aléatoire : A) Toutes occurrences ; B) Tirage aléatoire de 25000 occurrences ; C) Tirage stratifié sur l’étendue de distribution.
On obtient effectivement une meilleure représentativité des zones faiblement échantillonnées avec l’approche stratifiée, mais le coût est trop fort en terme d’abondance locale. On préférera pour cette raison l’approche de tirage aléatoire.
2.4 Modélisation de la niche climatique
Tout le travail de modélisation de la niche climatique se fait avec l’aide du
package biomod2. Ce package permet de
gérer toutes les étapes de modélisation :
- génération des pseudo-absences ;
- modèles individuels de niche ;
- modèles d’ensemble ;
- projections.
Note : On travaille avec la version 4 de développement de biomod2.
2.4.1 Préparer les données
La première étape est de préparer les données de travail, c’est-à-dire organiser
les données d’entrées (occurrences et données environnementales) et y associer
des données produites de pseudo-absences. Les résultats sont stockés sur le
disque, dans un sous-dossier par espèce. On commence par convertir les données
d’occurrences (sous-échantillonnées) au format SpatVector du package terra
qui sera utilisé pour la modélisation.
class : SpatVector
geometry : points
dimensions : 25000, 0 (geometries, attributes)
extent : -9.980495, 39.931355, 37.182608, 68.889571 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326) Il existe plusieurs approches pour les pseudo-absences. Nous utiliserons les paramètres suivants :
- On utilisera un tirage aléatoire des pseudo-absences. C’est une approche préférable quand la présence de l’espèce n’est pas complète ou biaisée, et quand la spécificité (taux de faux négatifs) est préférable à la sensitivité (taux de faux positifs) ;
- Chaque jeu de pseudo-absences aura un nombre de pseudo-absences égal au nombre d’occurrences ;
- On tirera pour chaque espèces 3 jeux indépendants de pseudo-absences pour obtenir des résultats plus robustes.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= acps Data Formating -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
! Response variable is considered as only presences... Is it really what you want?
! No data has been set aside for modeling evaluation
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
Checking Pseudo-absence selection arguments...
> random pseudo absences selection
> Pseudo absences are selected in explanatory variables
! Some NAs have been automatically removed from your data
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.formated.data -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
dir.name = Output
sp.name = acps
24979 presences, 0 true absences and 72904 undefined points in dataset
6 explanatory variables
temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart
Min. : 0.22400 Min. :-22.0480003 Min. :-14.856667 Min. : 0.0000 Min. : 3.0000
1st Qu.:19.98800 1st Qu.: -8.7840004 1st Qu.: 7.981333 1st Qu.: 603.8567 1st Qu.: 175.0000
Median :23.04800 Median : -3.2560000 Median : 12.525333 Median : 720.0280 Median : 217.0000
Mean :24.51093 Mean : -4.1916097 Mean : 11.869645 Mean : 748.3040 Mean : 228.8275
3rd Qu.:27.87600 3rd Qu.: 0.8666667 3rd Qu.: 16.103333 3rd Qu.: 897.2839 3rd Qu.: 262.0000
Max. :45.97600 Max. : 12.3000002 Max. : 26.380001 Max. :1380.1462 Max. :1254.0000
prec_season
Min. : 4.719104
1st Qu.: 20.915611
Median : 30.756525
Mean : 34.548040
3rd Qu.: 39.135733
Max. :123.759880
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=On visualise les pseudo-absences :
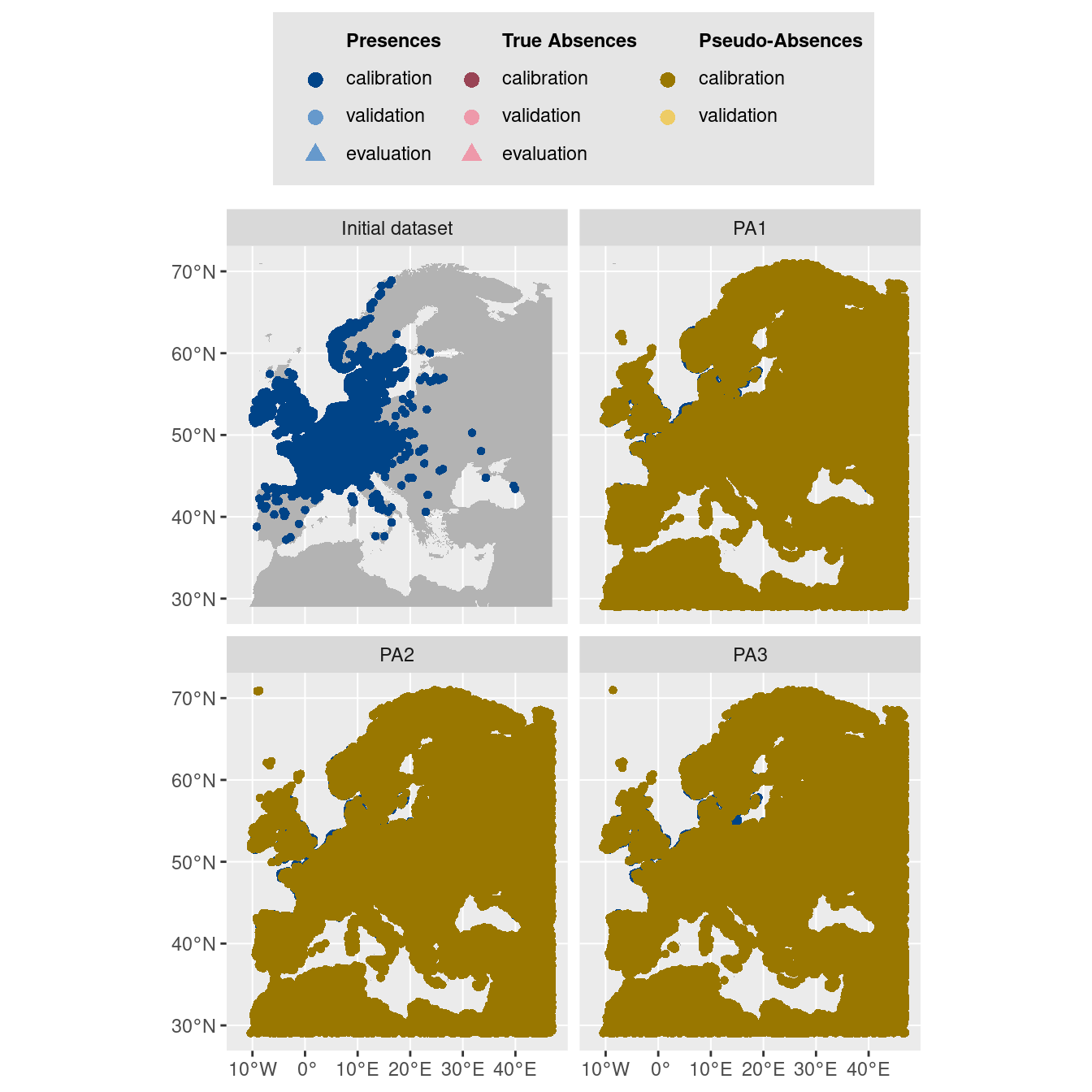
Figure 2.6: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
$data.vect
class : SpatVector
geometry : points
dimensions : 174916, 2 (geometries, attributes)
extent : -10.52083333, 46.97916667, 29.02083333, 70.97916667 (xmin, xmax, ymin, ymax)
coord. ref. :
names : resp dataset
type : <num> <chr>
values : 10 Initial dataset
10 Initial dataset
10 Initial dataset
$data.label
9 10 11
"**Presences**" "Presences (calibration)" "Presences (validation)"
12 19 20
"Presences (evaluation)" "**True Absences**" "True Absences (calibration)"
21 22 29
"True Absences (validation)" "True Absences (evaluation)" "**Pseudo-Absences**"
30 31 1
"Pseudo-Absences (calibration)" "Pseudo-Absences (validation)" "Background"
$data.plot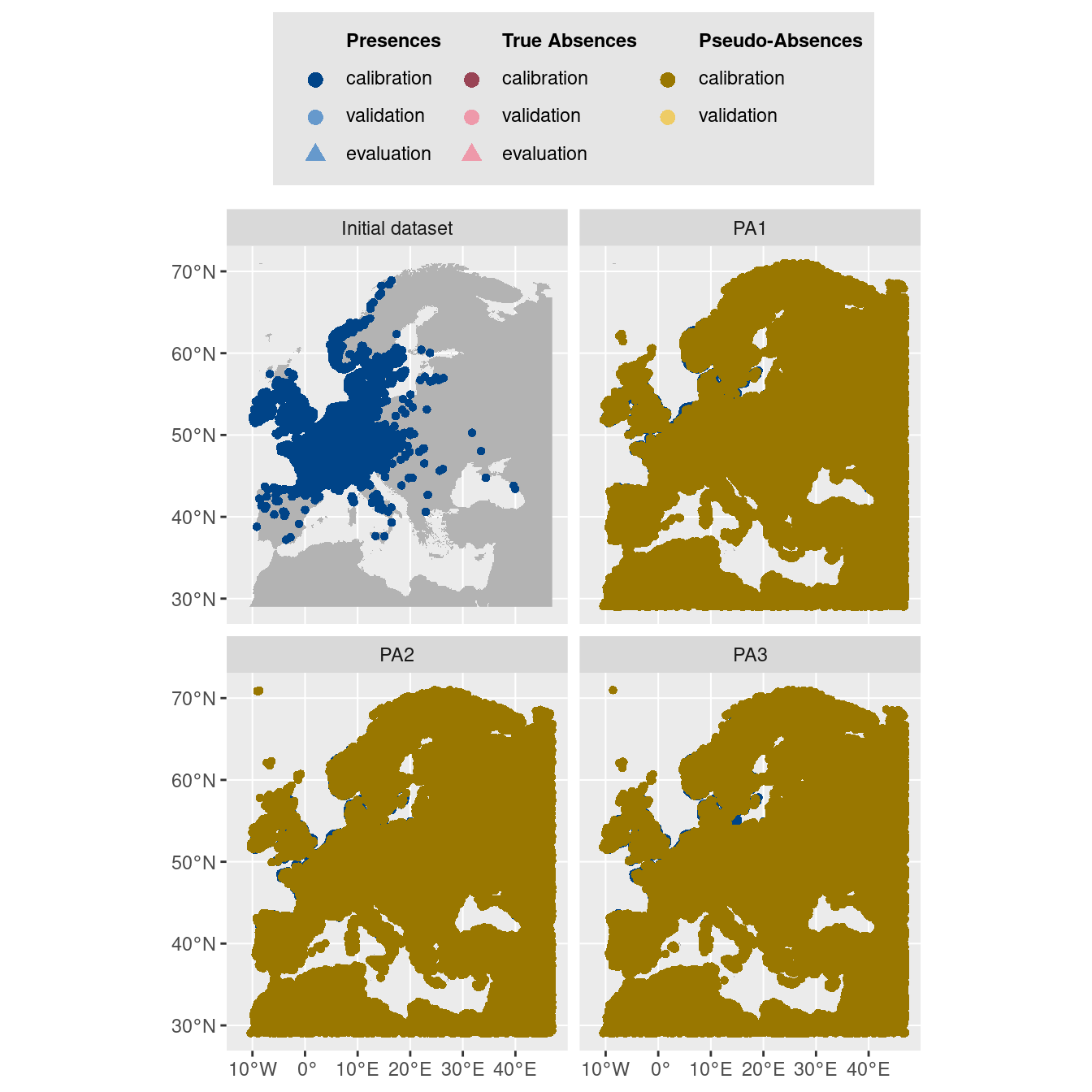
Figure 2.7: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
2.4.2 Modèles individuels de niche
à partir des occurrences + données environnementales
prendre en compte jeu de données validation
8 algorithmes :
- Méthodes de régression :
- Modèle additif généralisé (Generalized additive model GAM)
- Splines de régression multivariée adaptative (Multivariate adaptive regression splines MARS)
- MaxEnt ou sa nouvelle implémentation MaxNet
- Mélange de méthodes de classification et d’apprentissage automatique (machine
learning)
- Arbres de régression boostés (Generalized boosting model GBM, habituellement appelé Boosted regression trees)
- Réseaux artificiels de neurones (Artificial neural networks ANN)
- Forêts aléatoires (Random forest RF)
- on change le dossier de travail spécifiquement pour
biomod2(enregistrement des résultats dans Output/) - metric.eval = c(“KAPPA”, “TSS”, “ROC”) par défaut → on regarde TSS/ROC
- jeu de données découpé en 80/20 pour ajustement/évaluation croisée
- on éliminera si besoin les modèles qui ne donnent pas de bons résultats manuellement
On prépare une colonne dans le tableau des espèces qui permettra de spécifier quels modèles ne sont pas pris en compte (par défaut, tous sont inclus) :
→ options par défaut pour chaque algorithme (par exemple utilisation de la méthode “MGCV” pour le GAM, 500 arbres pour les forêts aléatoires, 200 itérations maximum pour MaxEnt, etc.)
prendre en compte jeu de données validation
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Build Single Models -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Checking Models arguments...
! 'MAXENT.Phillips.2' model name is deprecated, please use 'MAXNET' instead.
Creating suitable Workdir...
! Weights where automatically defined for acps_PA1 to rise a 0.5 prevalence !
! Weights where automatically defined for acps_PA2 to rise a 0.5 prevalence !
! Weights where automatically defined for acps_PA3 to rise a 0.5 prevalence !
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= acps Modeling Summary -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
6 environmental variables ( temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season )
Number of evaluation repetitions : 1
Models selected : GAM MARS MAXNET GBM ANN RF
Total number of model runs: 18
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=- Run : acps_PA1
-=-=-=--=-=-=- acps_PA1_RUN1
-=-=-=- Run : acps_PA2
-=-=-=--=-=-=- acps_PA2_RUN1
-=-=-=- Run : acps_PA3
-=-=-=--=-=-=- acps_PA3_RUN1
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=2.4.2.1 Évaluation de la performance
L’évaluation de la performance se fait par deux statistiques complémentaires, basées sur la sensibilités/sélectivité, c’est-à-dire le taux de vrais positifs, et la spécificité, c’est-à-dire le taux de vrais négatifs :
- TSS : True skill statistic, qui évalue la capacité de discrimination d’une vraie présence par rapport à une absence, en utilisant un seuil de classification de la probabilité du modèle dans les deux catégories (0,5 par défaut). La TSS va de -1 à 1, avec un score parfait quand la TSS vaut 1 (0 dénotant une performance équivalente à un classement aléatoire). ;
- AUC : Aire sous la courbe de ROC (Receiver operating characteristic), qui s’affranchit de l’effet de seuil en testant plusieurs valeurs de 0 à 1. L’AUC va de 0 à 1, avec un score parfait quand l’AUC vaut 1 (0,5 dénotant une performance équivalente à un classement aléatoire).
On utilise la fonction get_evaluations() pour récupérer ces deux statistiques
d’intérêt, avant de les visualiser selon l’algorithme. On s’appuie sur des
seuils standards de 0,9 pour l’AUC et 0,8 pour la TSS :
full.name PA run algo metric.eval cutoff sensitivity specificity calibration
1 acps_PA1_RUN1_GAM PA1 RUN1 GAM TSS 687.0 95.106 64.825 0.600
2 acps_PA1_RUN1_GAM PA1 RUN1 GAM ROC 692.5 95.001 64.990 0.755
3 acps_PA1_RUN1_MARS PA1 RUN1 MARS TSS 488.0 96.727 85.220 0.820
4 acps_PA1_RUN1_MARS PA1 RUN1 MARS ROC 488.5 96.727 85.230 0.949
5 acps_PA1_RUN1_MAXNET PA1 RUN1 MAXNET TSS 341.0 97.108 84.850 0.820
6 acps_PA1_RUN1_MAXNET PA1 RUN1 MAXNET ROC 341.5 97.098 84.900 0.950
validation evaluation
1 0.599 NA
2 0.754 NA
3 0.823 NA
4 0.949 NA
5 0.820 NA
6 0.949 NA
Figure 2.8: Évaluation de la performance des modèles individuels : True Skill Statistic & aire sous la courbe de ROC.
Les GAMs, et une partie des ANNs ont une performance médiocre, et seront automatiquement exclus pour les modèles d’ensemble.
2.4.2.2 Importance des variables environnementales
L’importance de chaque variable est estimée par randomisation de la variable
d’intérêt (moyenne/variance sur tous les algorithmes). On utilise la fonction
get_variables_importance() pour récupérer l’importance des variables, que l’on
peut visualiser par algorithme ou par variable :
full.name PA run algo expl.var rand var.imp
1 acps_PA1_RUN1_GAM PA1 RUN1 GAM temp_max_august 1 0.276205
2 acps_PA1_RUN1_GAM PA1 RUN1 GAM temp_min 1 0.194088
3 acps_PA1_RUN1_GAM PA1 RUN1 GAM temp_wet_quart 1 0.008312
4 acps_PA1_RUN1_GAM PA1 RUN1 GAM temp_season 1 0.378992
5 acps_PA1_RUN1_GAM PA1 RUN1 GAM prec_wet_quart 1 0.065938
6 acps_PA1_RUN1_GAM PA1 RUN1 GAM prec_season 1 0.166128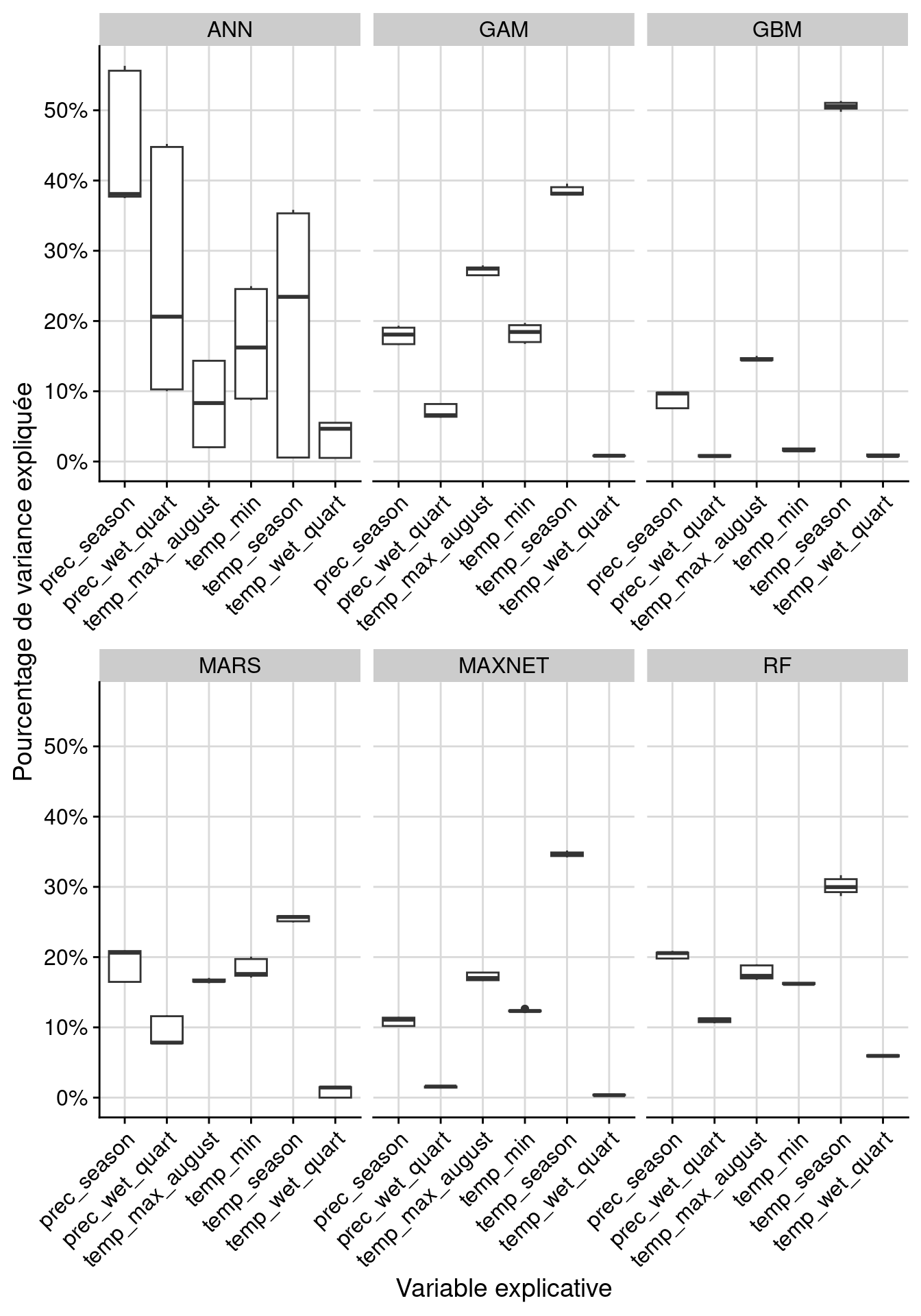
Figure 2.9: Pourcentage de variance expliquée pour chaque variable, décomposé par algorithme.
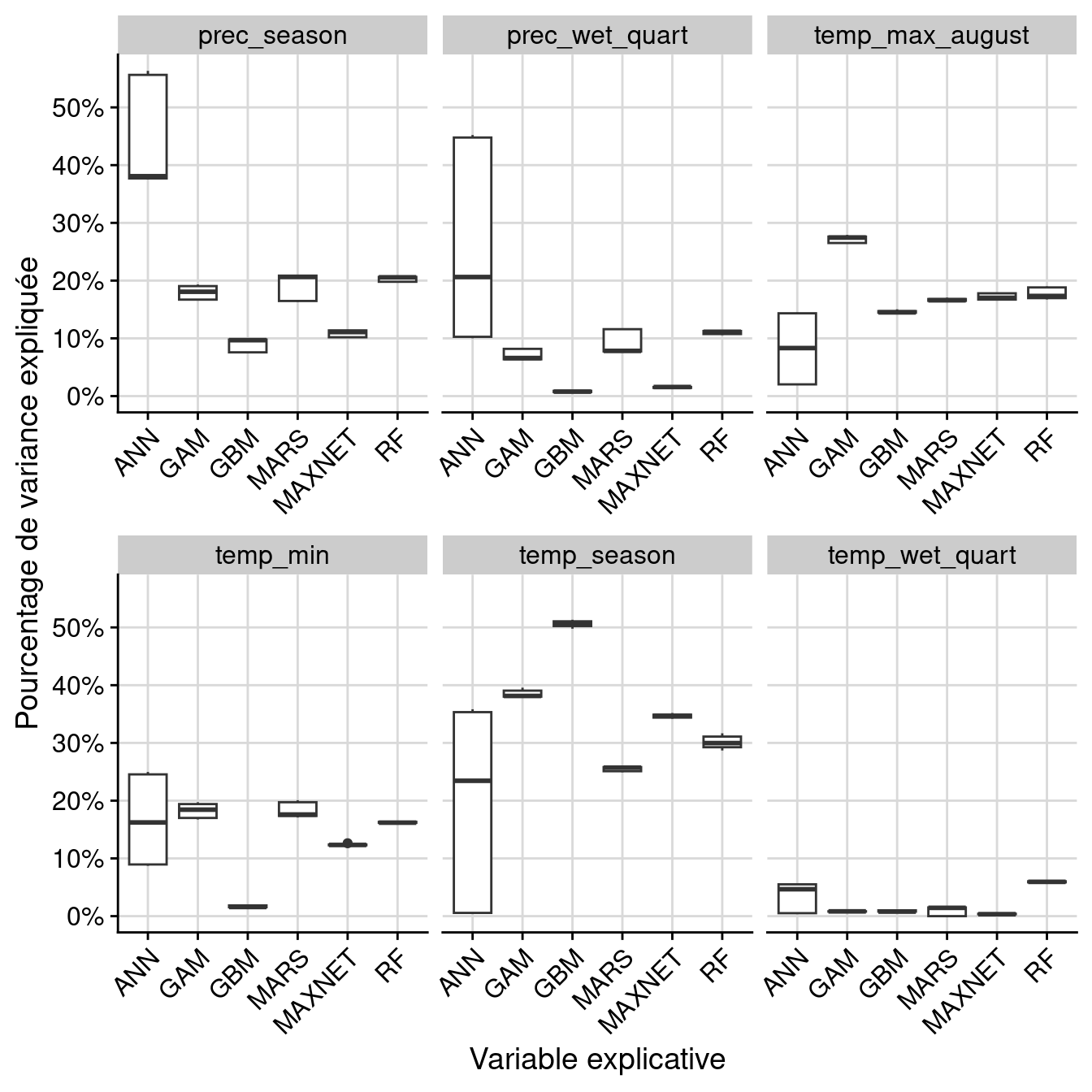
Figure 2.10: Pourcentage de variance expliquée pour chaque variable, décomposé par variable.
2.4.2.3 Courbes de réponse
Finalement, le modèle permet d’établir des courbes de réponse, c’est-à-dire la
variation de la probabilité d’occurrence selon chaque variable
environnementale. On utilise pour cela la fonction bm_PlotResponseCurves() qui
permet d’extraire ces valeurs et de les afficher :
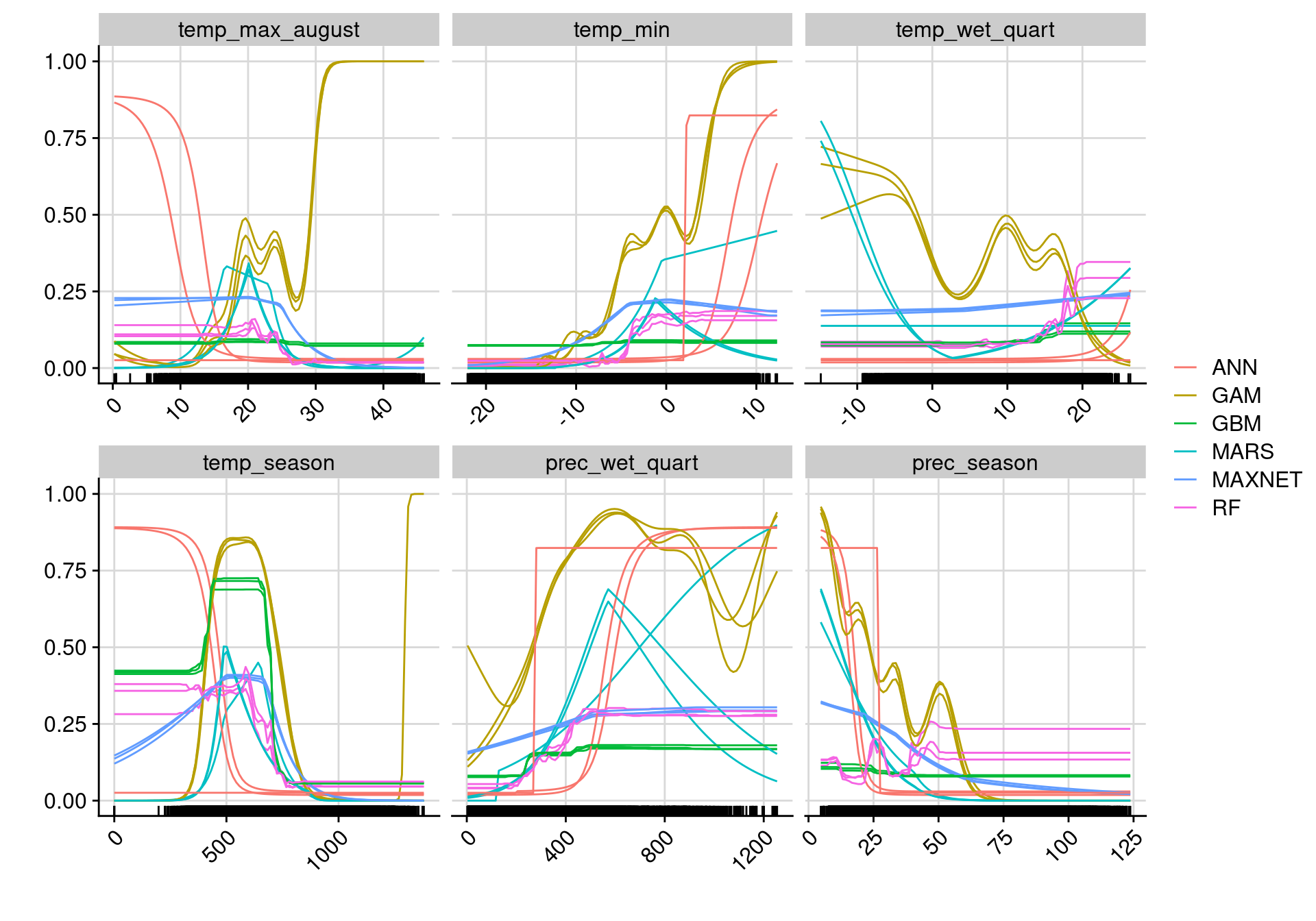
Figure 2.11: Courbes de réponse de chaque variable climatique selon l’algorithme (en couleurs).
2.4.3 Modèles d’ensemble de niche
Seulement modèles avec TSS > 0.8
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.ensemble.models.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
sp.name : acps
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
models computed:
acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo, acps_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
models failed: none
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=→ prend les modèles individuels en entrée → on décide sur quel critère les modèles sont gardés
même idée que l’interférence multi-modèles, mais basé sur métrique de performance (e.g. TSS > 0.75 et AUC/ROC > 0.9)
→ on peut calculer moyenne ou médiane → retourne des coefficients de variation → retourne des probabilités pondérées par la métrique choisie
→ grouping level stratifié jusqu’à “all”
2.4.3.1 Évaluation de la qualité
get_evaluations() pour récupérer les stats d’intérêt
full.name merged.by.PA merged.by.run merged.by.algo
1 acps_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
2 acps_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
filtered.by algo metric.eval cutoff sensitivity specificity calibration validation evaluation
1 TSS EMwmean TSS 605.0 95.648 90.974 0.866 NA NA
2 TSS EMwmean ROC 606.5 95.608 91.042 0.984 NA NA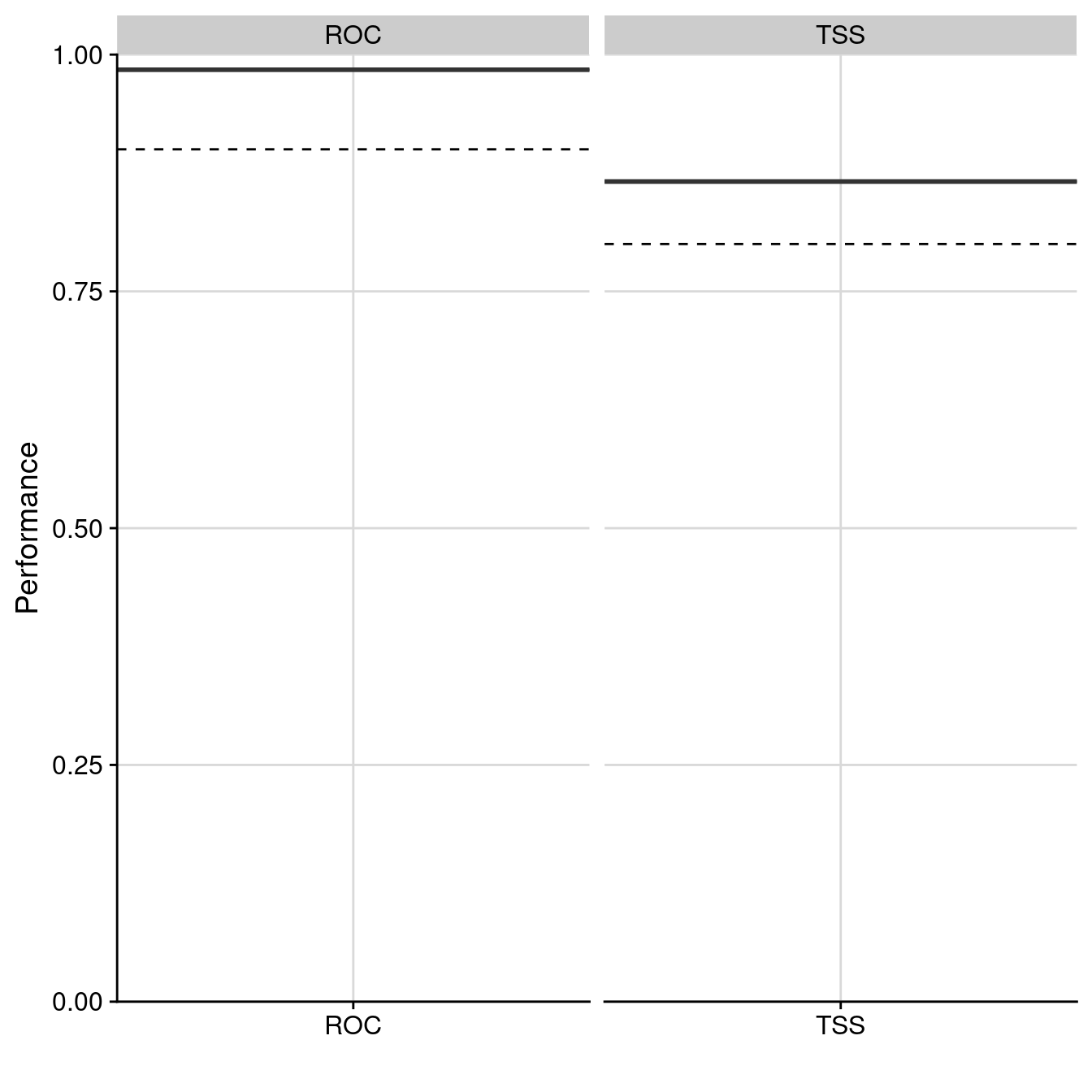
Figure 2.12: Évaluation de la qualité du modèle d’ensemble : Aire sous la courbe de ROC & True Skill Statistic.
full.name merged.by.PA merged.by.run merged.by.algo filtered.by
1 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
2 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
3 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
4 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
5 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
6 acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
algo expl.var rand var.imp
1 EMcv temp_max_august 1 0.157191
2 EMcv temp_min 1 0.145130
3 EMcv temp_wet_quart 1 0.021735
4 EMcv temp_season 1 0.378181
5 EMcv prec_wet_quart 1 0.048336
6 EMcv prec_season 1 0.136979Par variable :
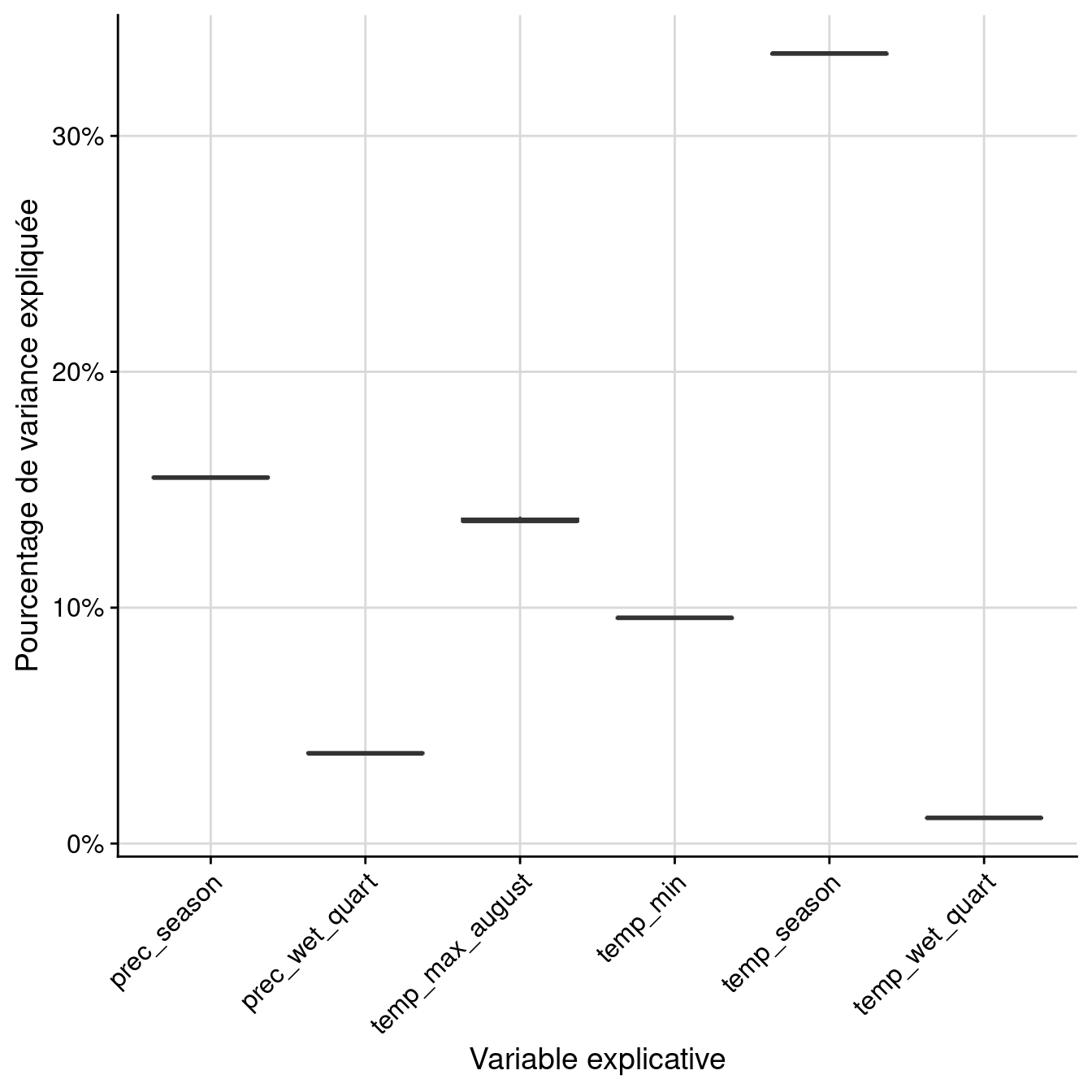
Figure 2.13: Pourcentage de variance expliquée par variable dans le modèle d’ensemble.
bm_PlotResponseCurves() pour forme de la réponse sur chaque variable
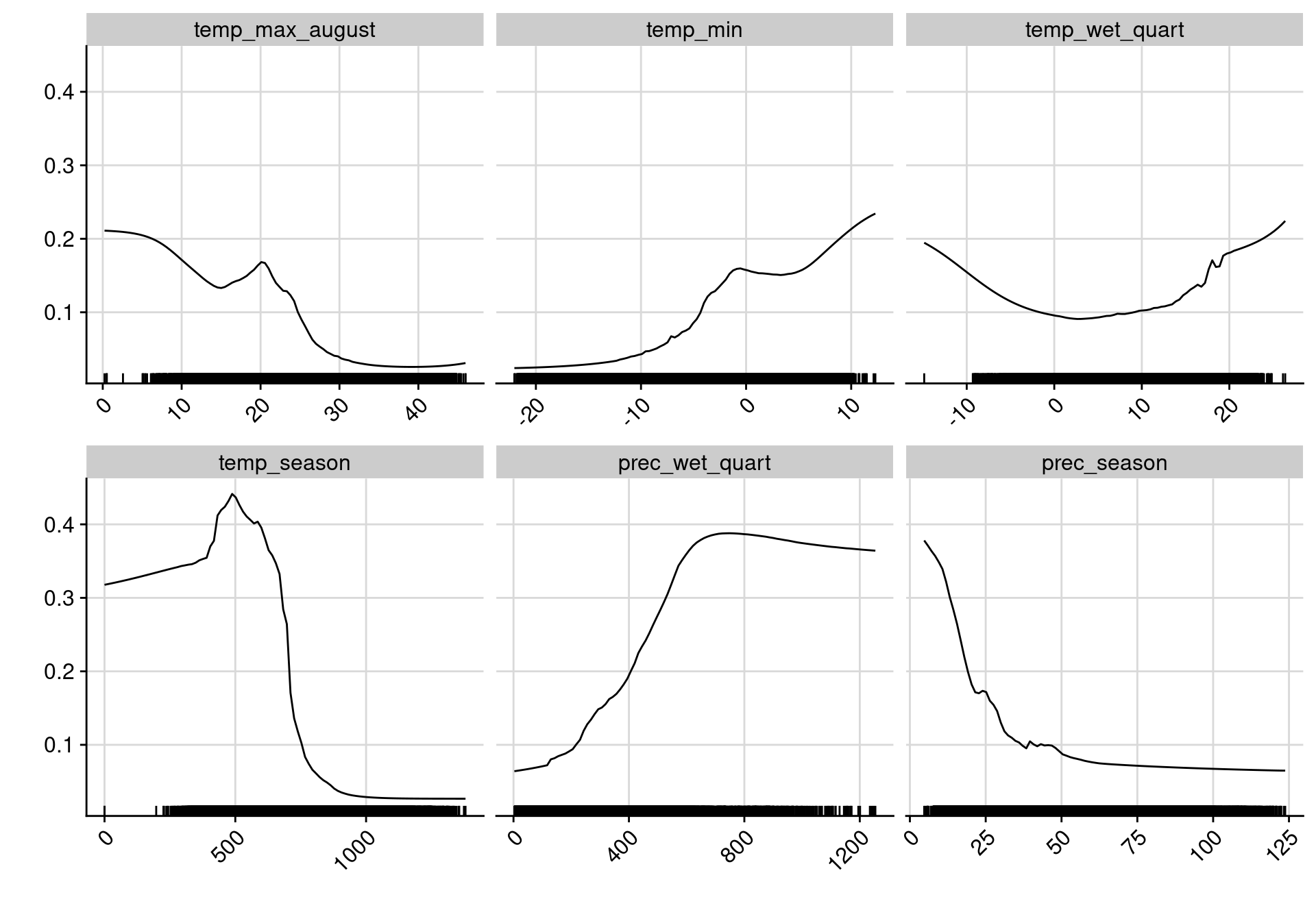
Figure 2.14: Courbes de réponse de chaque variable climatique dans le modèle d’ensemble.
2.5 Projections
2.5.1 Distribution potentielle contemporaine
Contemporaine : Cartographie des observations sur couches climatiques Extraction des conditions climatiques favorables
BIOMOD_Projection() un seul modèle
BIOMOD_EnsembleForecasting() modèle d’ensemble
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/acps/current
sp.name : acps
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/acps/acps.AllModels.models.out )
models.projected :
acps_PA1_RUN1_GAM, acps_PA1_RUN1_MARS, acps_PA1_RUN1_MAXNET, acps_PA1_RUN1_GBM, acps_PA1_RUN1_ANN, acps_PA1_RUN1_RF, acps_PA2_RUN1_GAM, acps_PA2_RUN1_MARS, acps_PA2_RUN1_MAXNET, acps_PA2_RUN1_GBM, acps_PA2_RUN1_ANN, acps_PA2_RUN1_RF, acps_PA3_RUN1_GAM, acps_PA3_RUN1_MARS, acps_PA3_RUN1_MAXNET, acps_PA3_RUN1_GBM, acps_PA3_RUN1_ANN, acps_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Figure 2.15: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.

Figure 2.16: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
Fonction get_proj() pour extraire plus facilement des sorties de projection.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/acps/current
sp.name : acps
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/acps/acps.AllModels.ensemble.models.out )
models.projected :
acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo, acps_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=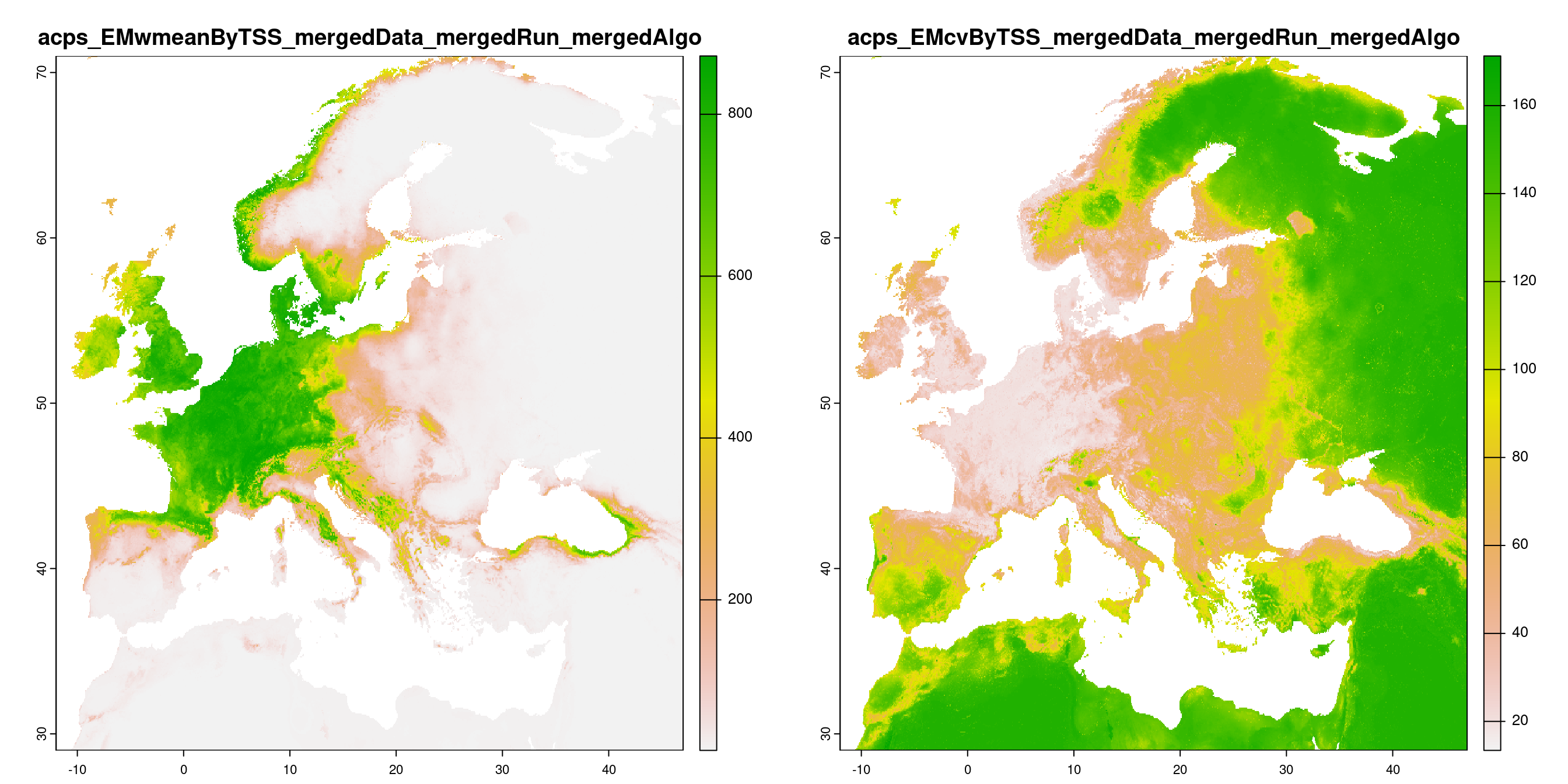
Figure 2.17: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) ; B) Incertitude associée à la projection.
2.5.2 Interpolation à l’échelle de la métropole
- → conditions climatiques favorables à fine échelle
On projette la niche climatique sur ce raster à fine échelle.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/acps/cont_gre
sp.name : acps
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/acps/acps.AllModels.models.out )
models.projected :
acps_PA1_RUN1_GAM, acps_PA1_RUN1_MARS, acps_PA1_RUN1_MAXNET, acps_PA1_RUN1_GBM, acps_PA1_RUN1_ANN, acps_PA1_RUN1_RF, acps_PA2_RUN1_GAM, acps_PA2_RUN1_MARS, acps_PA2_RUN1_MAXNET, acps_PA2_RUN1_GBM, acps_PA2_RUN1_ANN, acps_PA2_RUN1_RF, acps_PA3_RUN1_GAM, acps_PA3_RUN1_MARS, acps_PA3_RUN1_MAXNET, acps_PA3_RUN1_GBM, acps_PA3_RUN1_ANN, acps_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=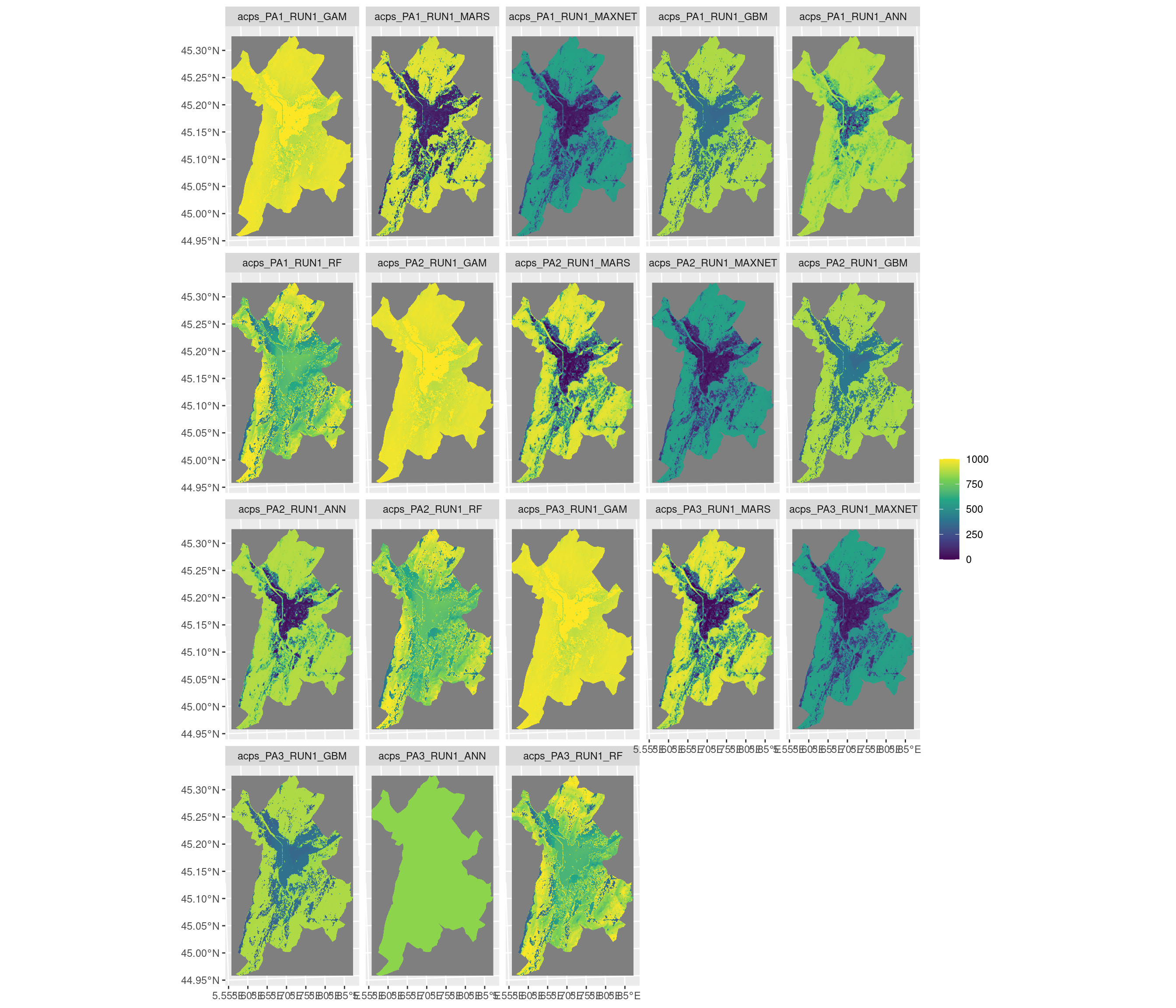
Figure 2.18: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
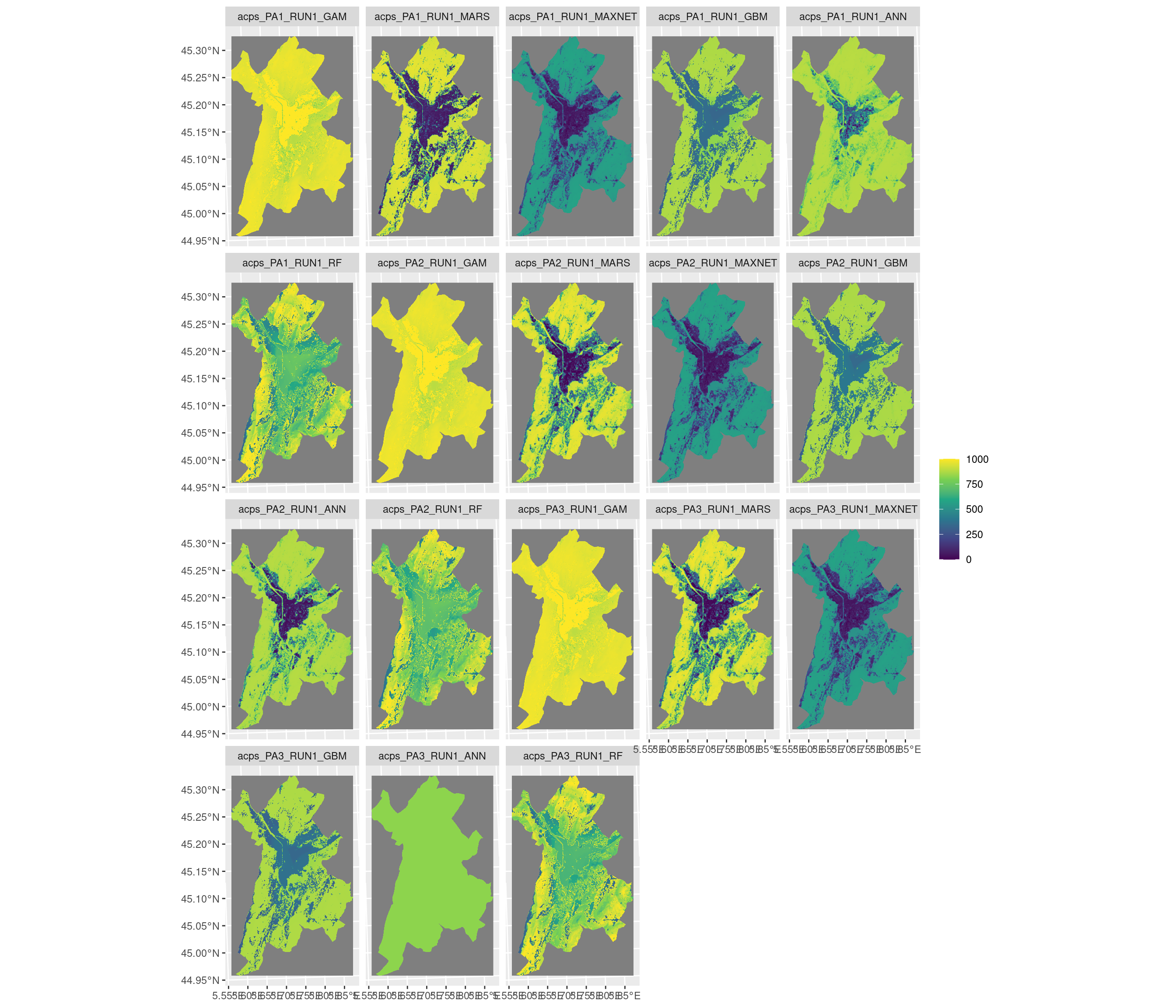
Figure 2.19: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/acps/cont_gre
sp.name : acps
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/acps/acps.AllModels.ensemble.models.out )
models.projected :
acps_EMcvByTSS_mergedData_mergedRun_mergedAlgo, acps_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=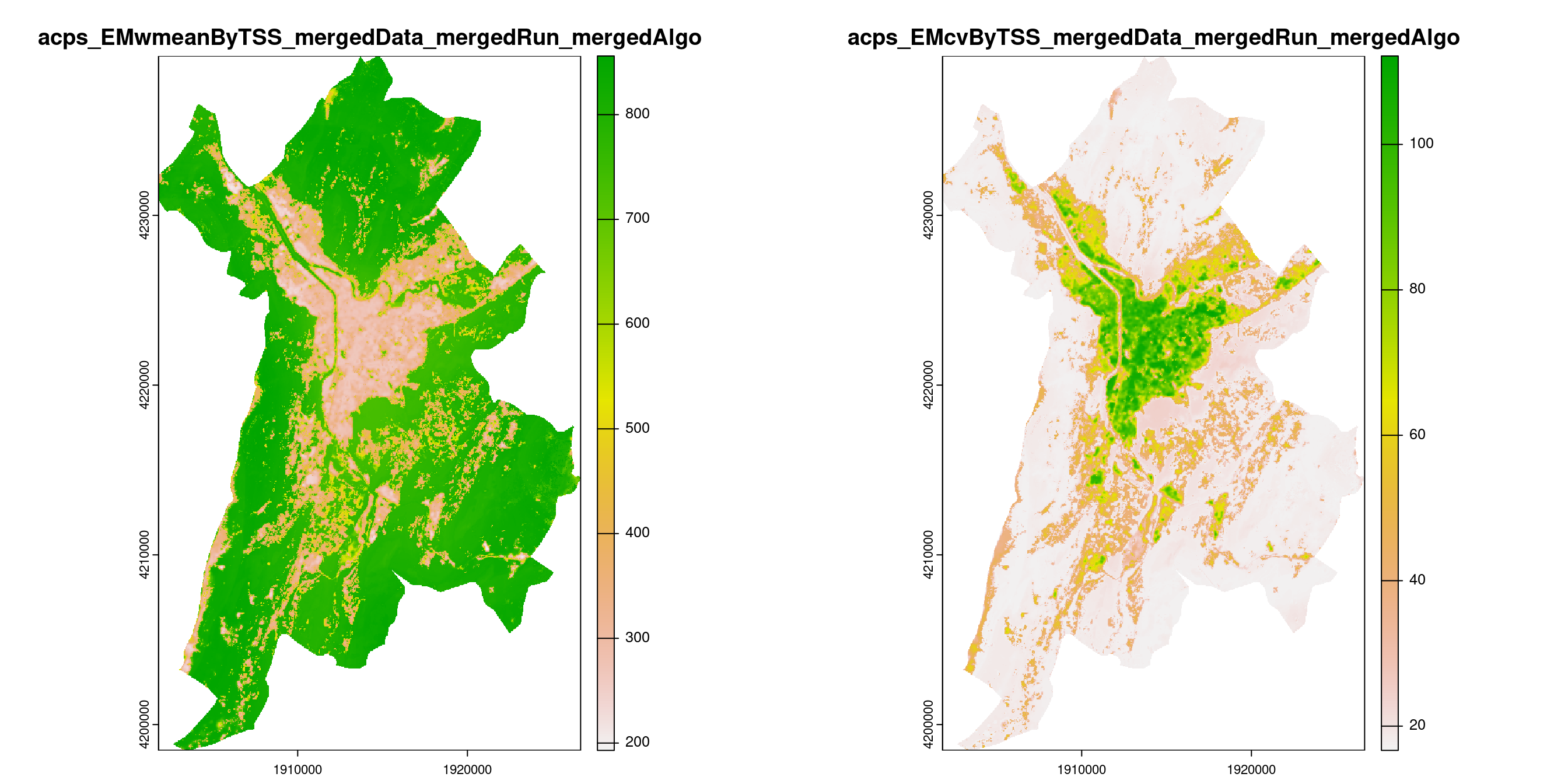
Figure 2.20: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) sur la métropole de Grenoble ; B) Incertitude associée à la projection.
2.5.3 Validation avec une référence française (IFN)
extraction des pixels par polygone (écorégion) et moyenne arithmétique pour chacun :
acps_density acps_current_proj
1 0.13838120104 640.2310757
2 0.20800000000 680.0717703
3 0.14847161572 769.3596112
4 0.03896103896 634.7235772
5 0.06145251397 723.5579515
6 0.00000000000 634.7446809IFN pour ACPS, densité + Carte de projection actuelle France
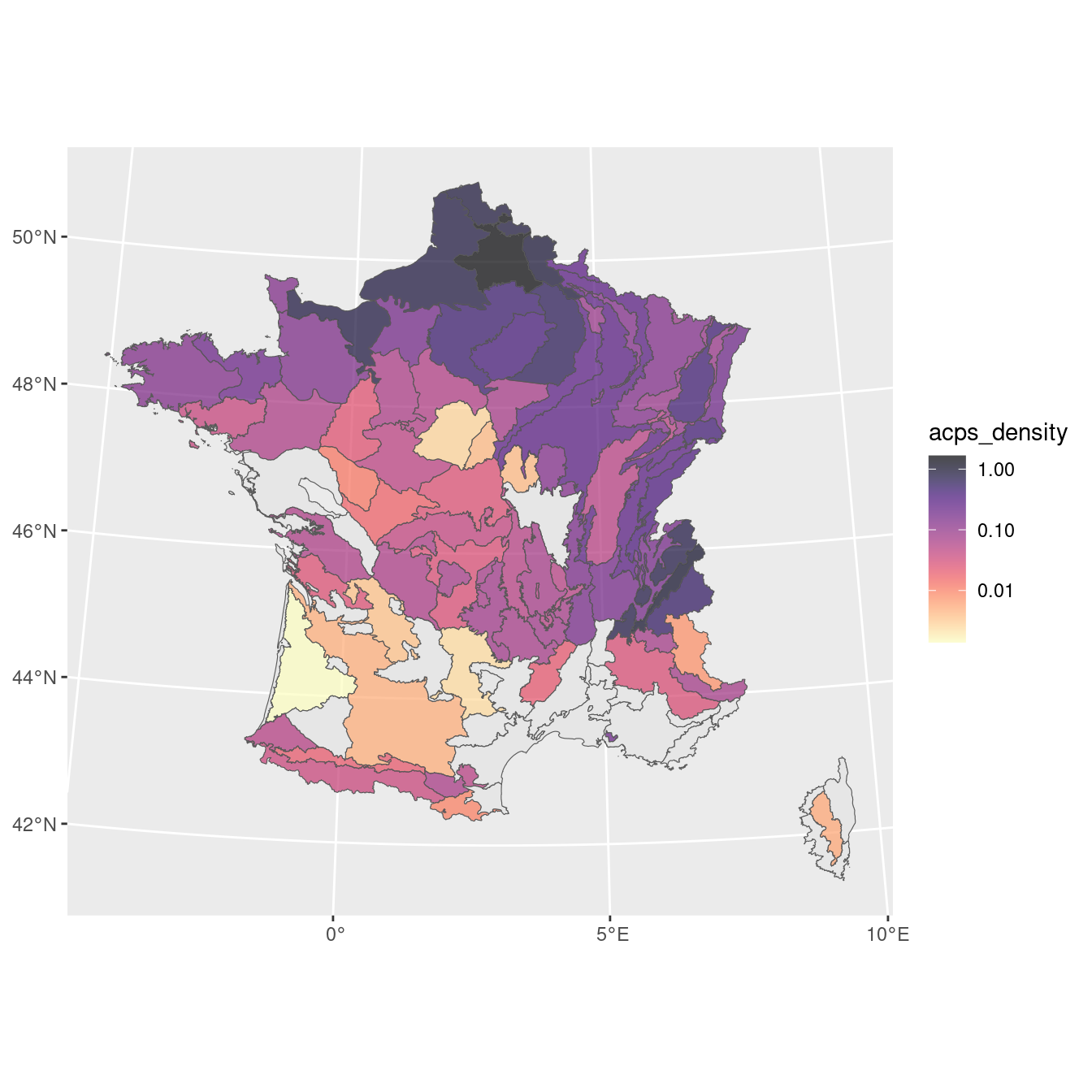
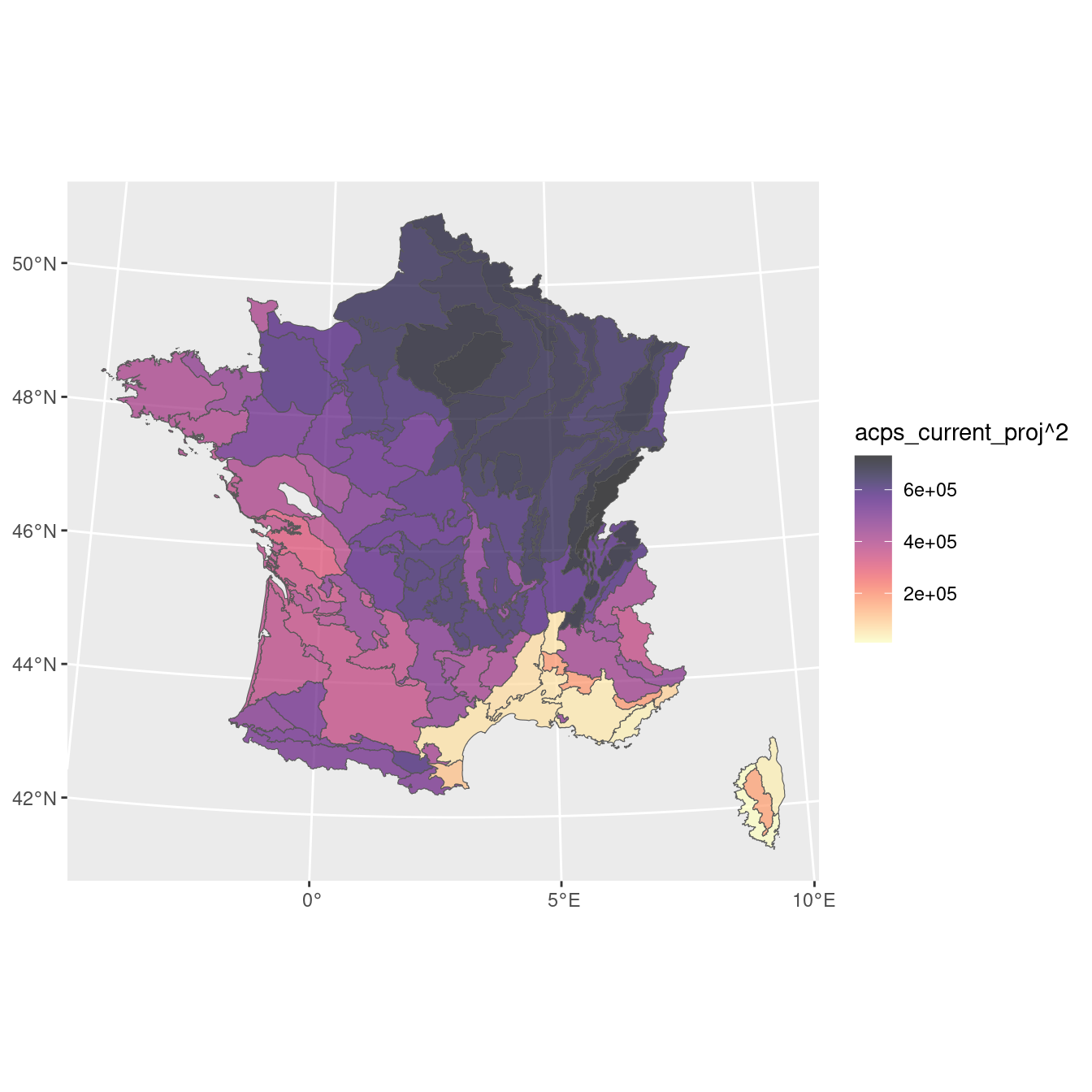
Figure 2.21: A) Carte de densité selon l’IFN ; B) Probabilité d’occurrence selon le modèle d’ensemble.
Les deux distributions à comparer sont très différentes (biaisées chacune d’un côté) On procède donc à une corrélation non paramétrique de Spearman (par les rangs) :
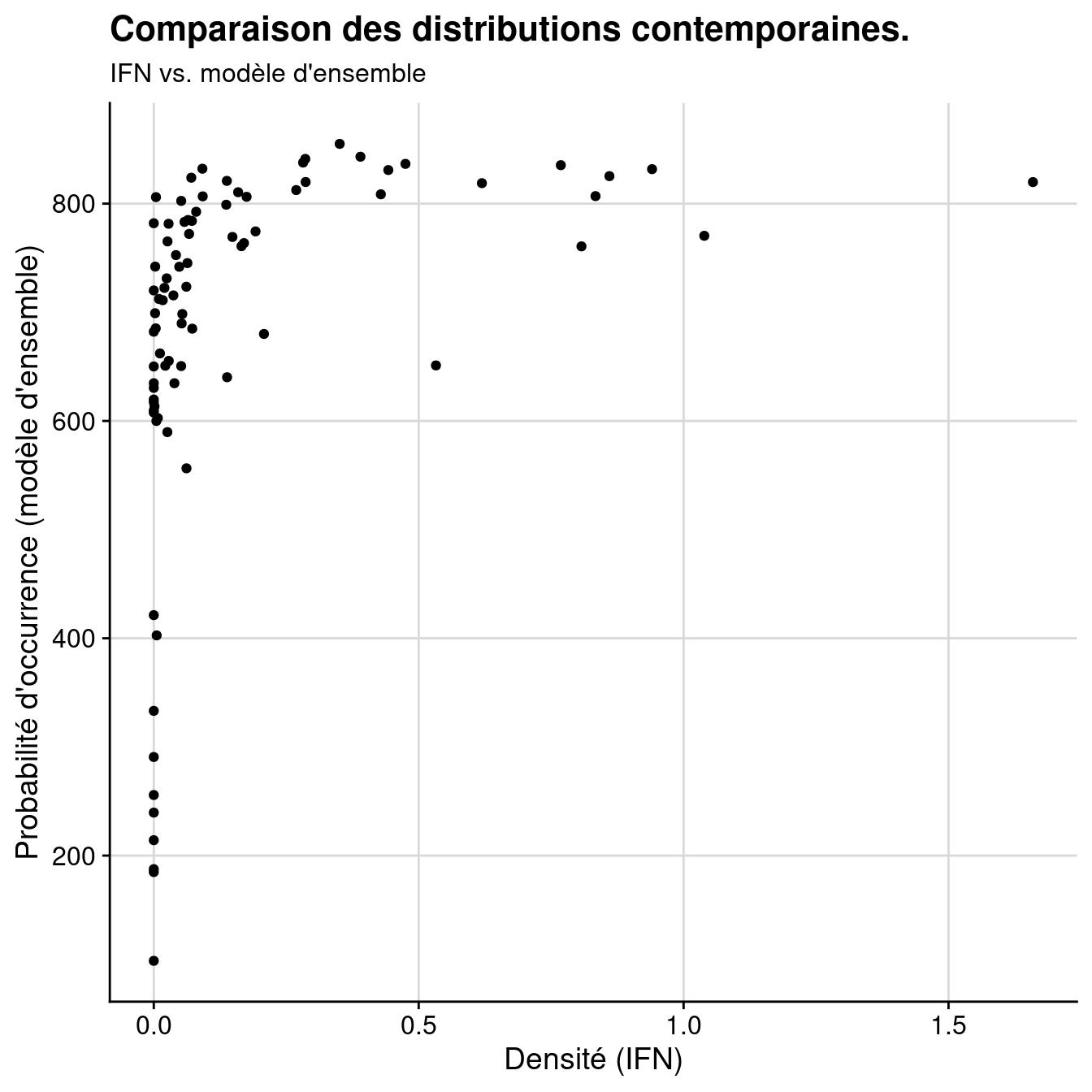
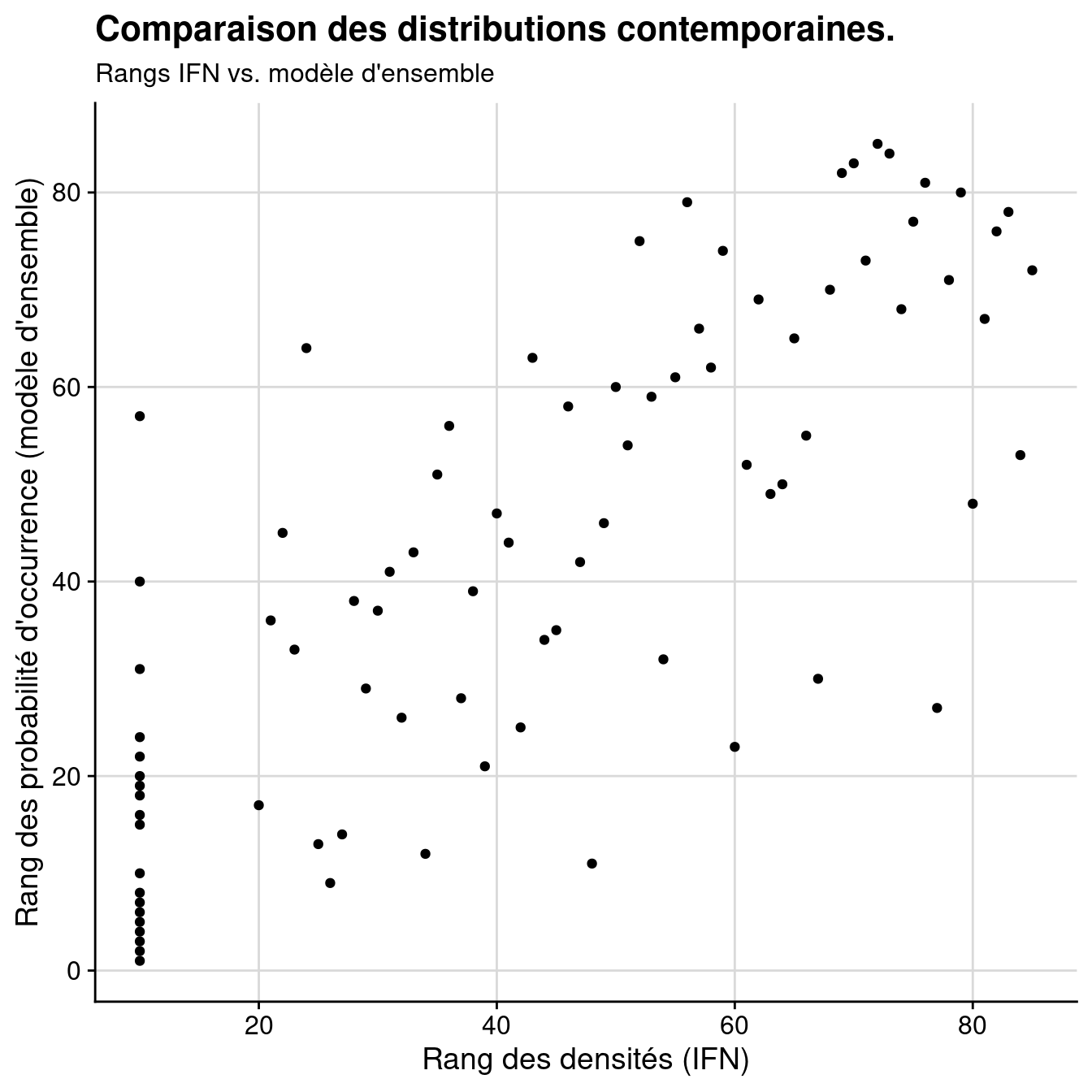
Figure 2.22: Comparaison des distributions contemporaines données par l’IFN vs. modèle d’ensemble : A) Données brutes ; B) Rangs.
[1] 0.77344241542.5.4 Distributions potentielles futures
Future : Projection à l’échelle de l’Europe jusqu’à l’horizon 2100
Toutes les fenêtres temporelles × scénarios pour le GCM « IPSL-CM6A-LR »
- SSP1-2.6
On peut évaluer directement la sortie du modèle d’ensemble.
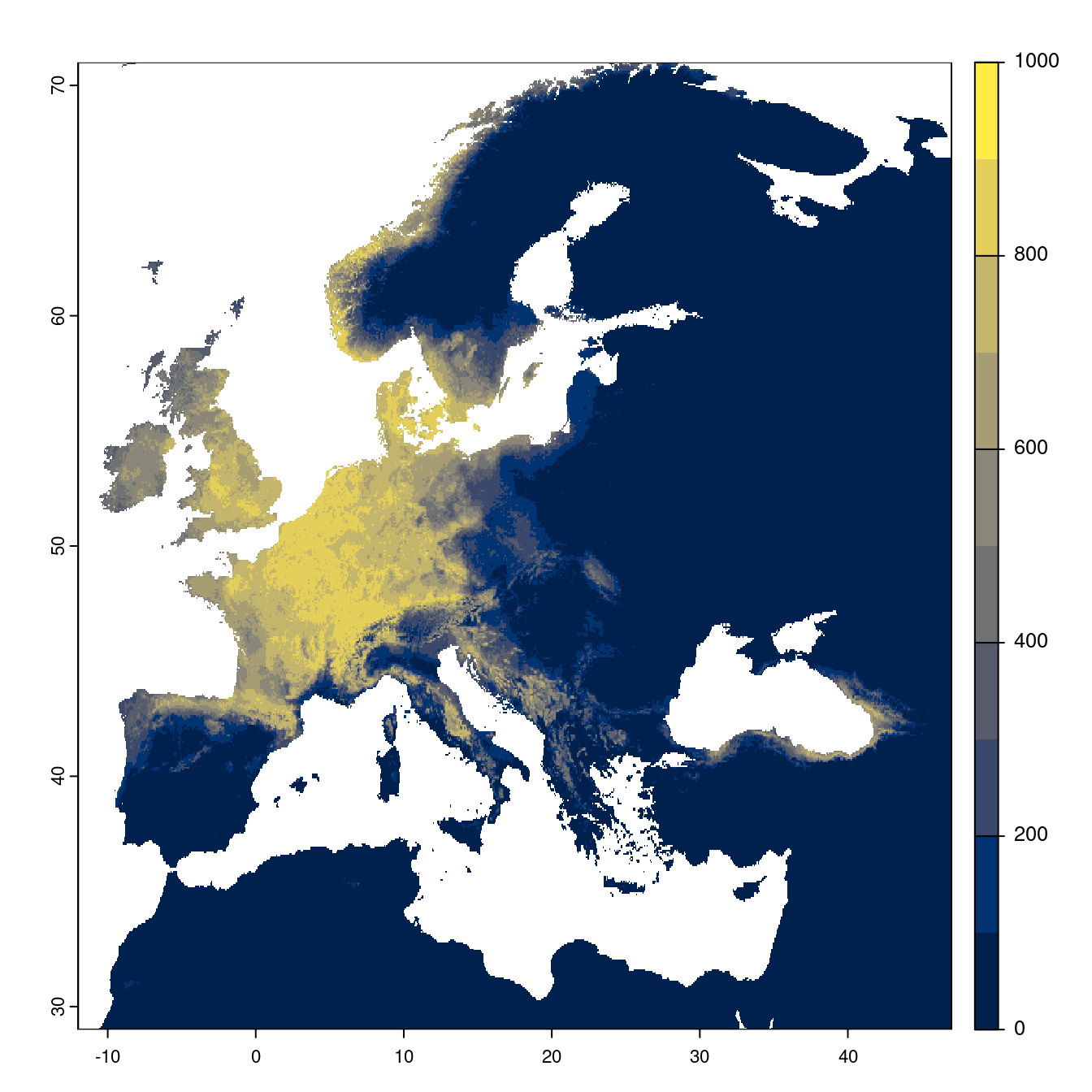
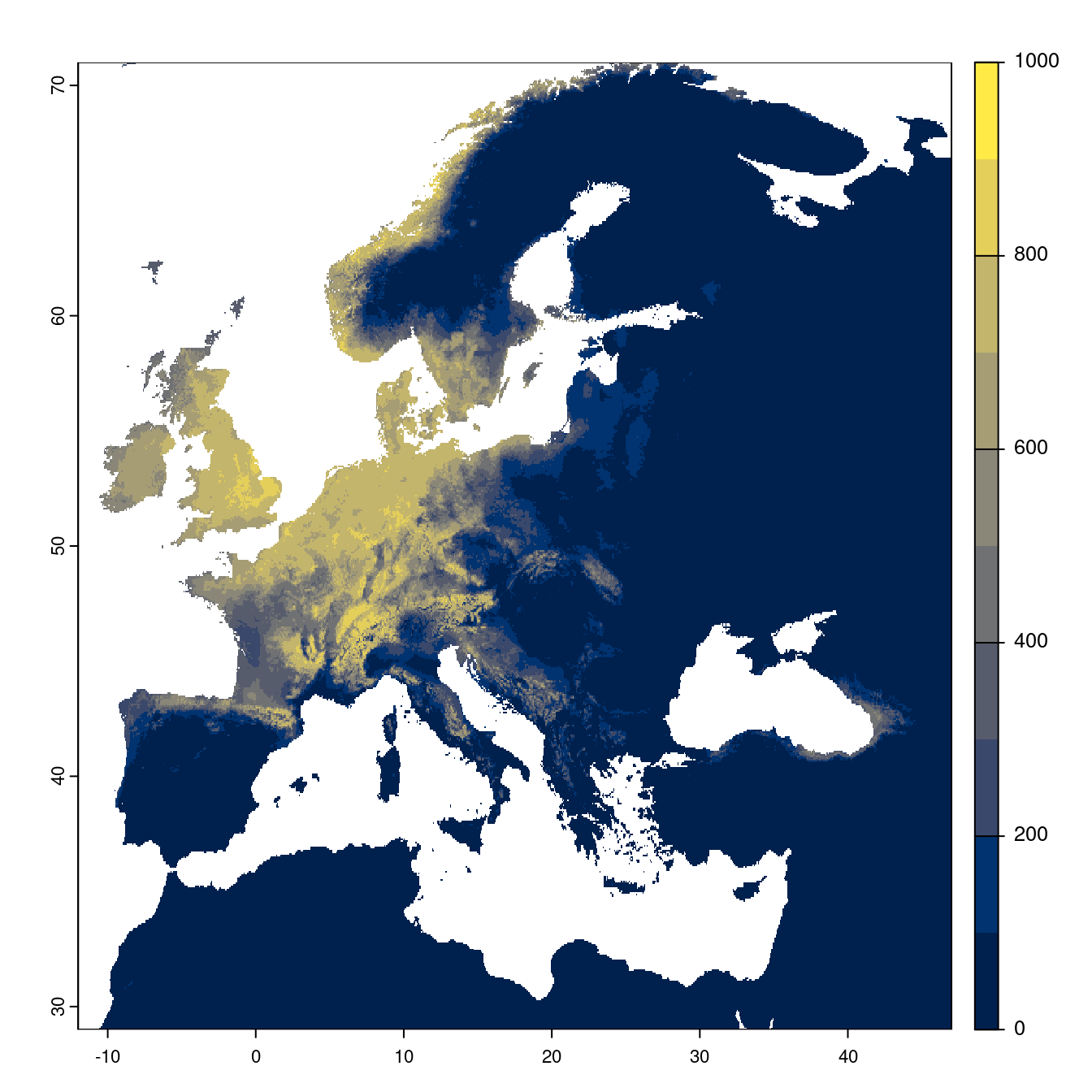
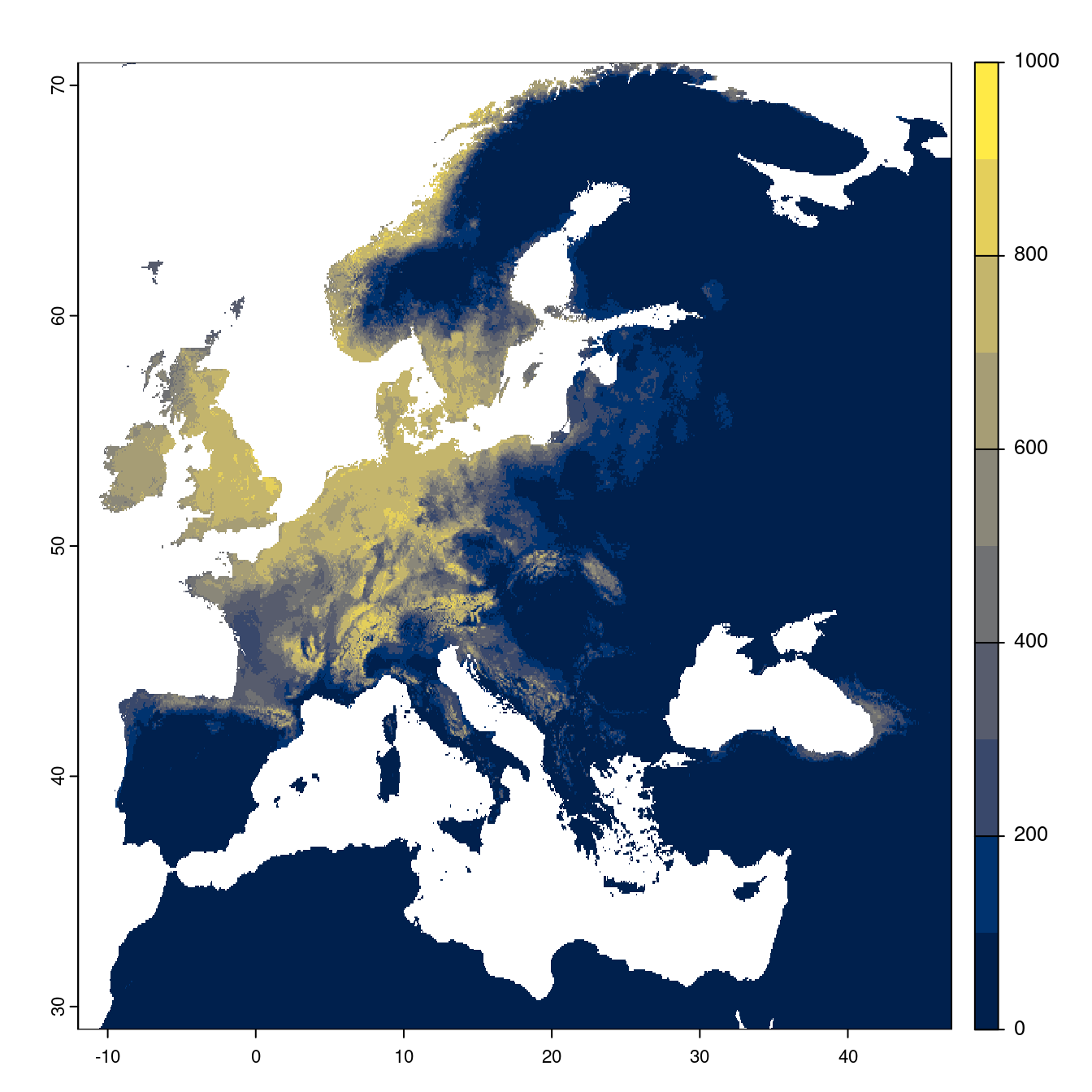
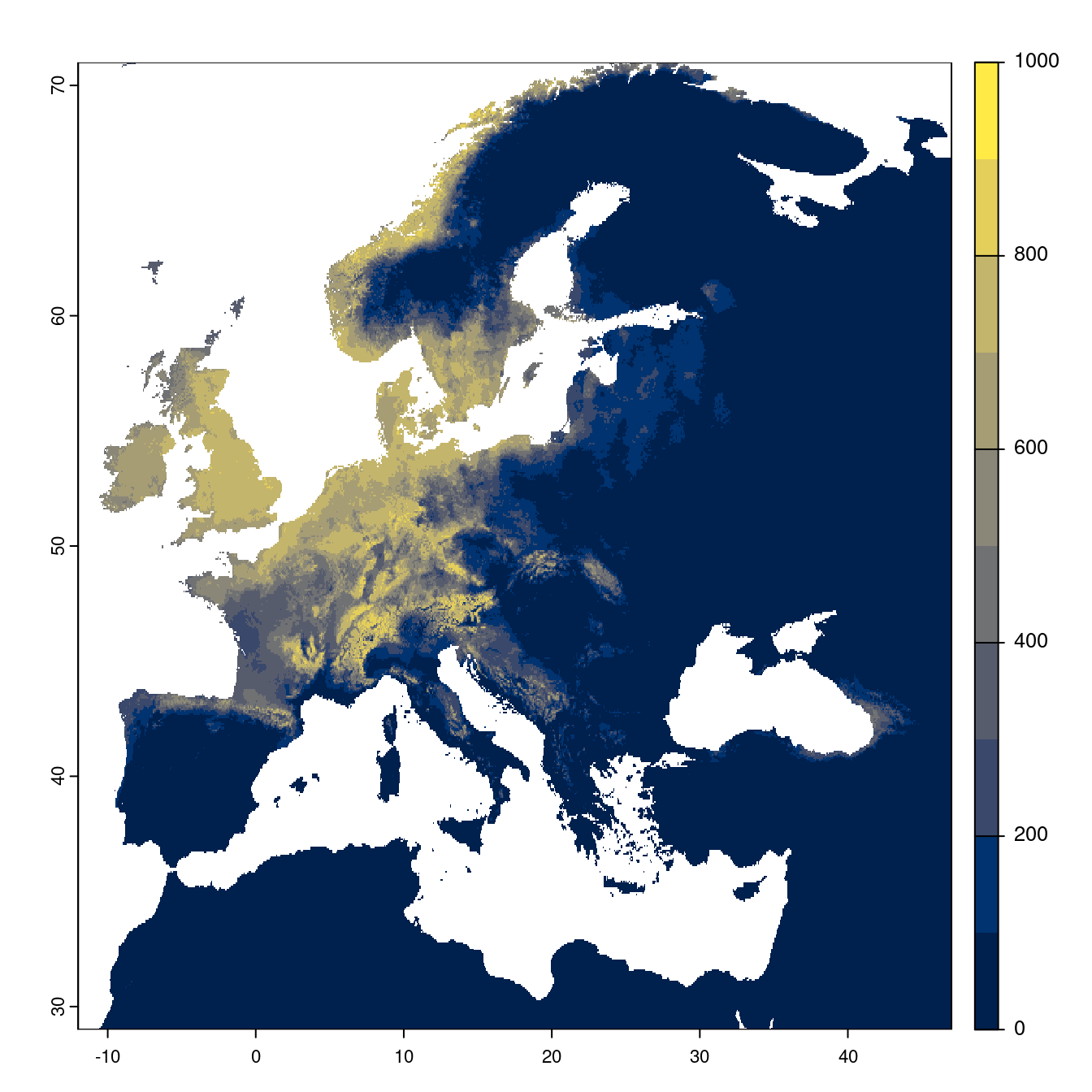

Figure 2.23: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP1-2.6 (modèle d’ensemble).
On obtient pour la métrique TSS une carte unique de moyenne pondérée sur les algorithmes et sur les jeux de pseudo-absences.
- SSP2-4.5
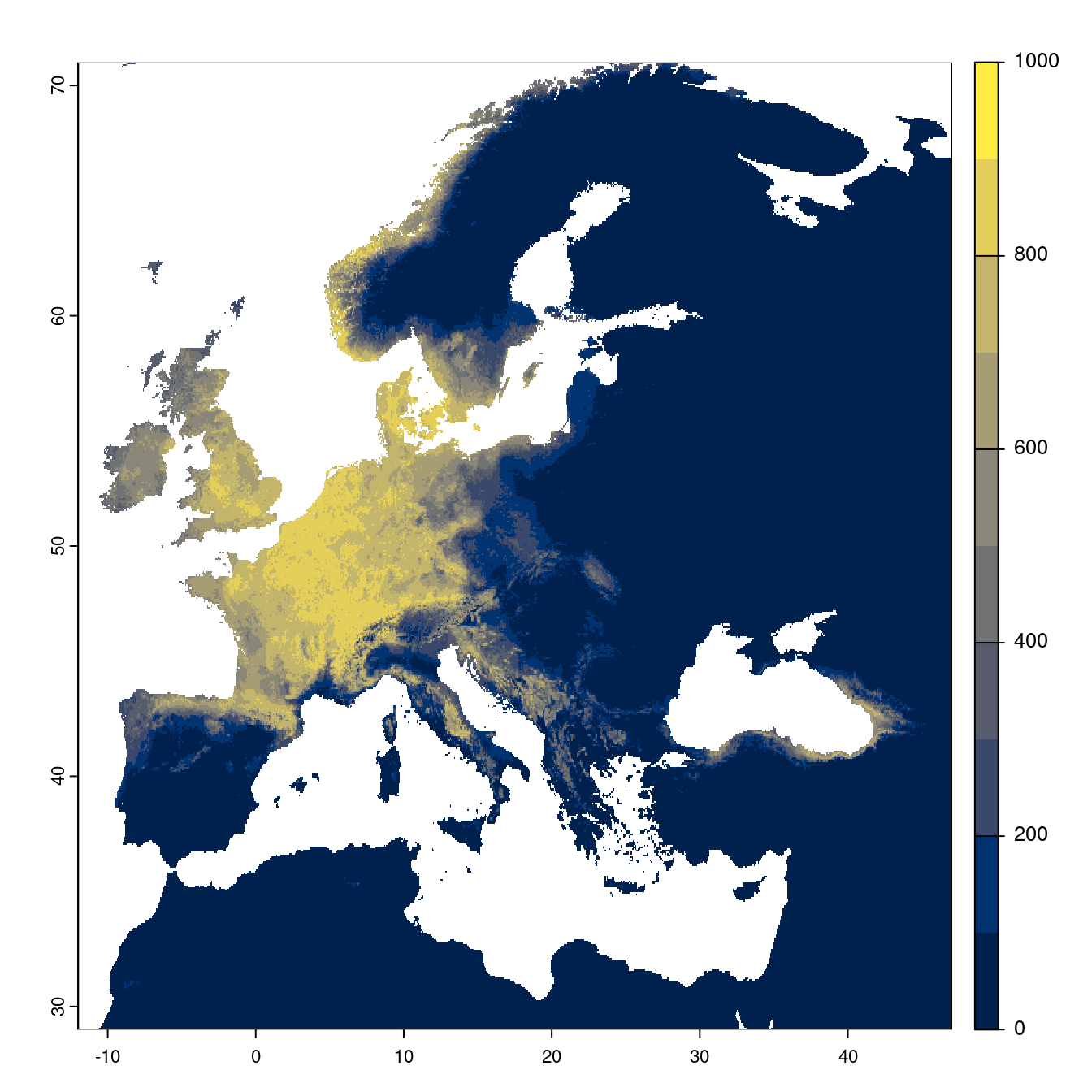
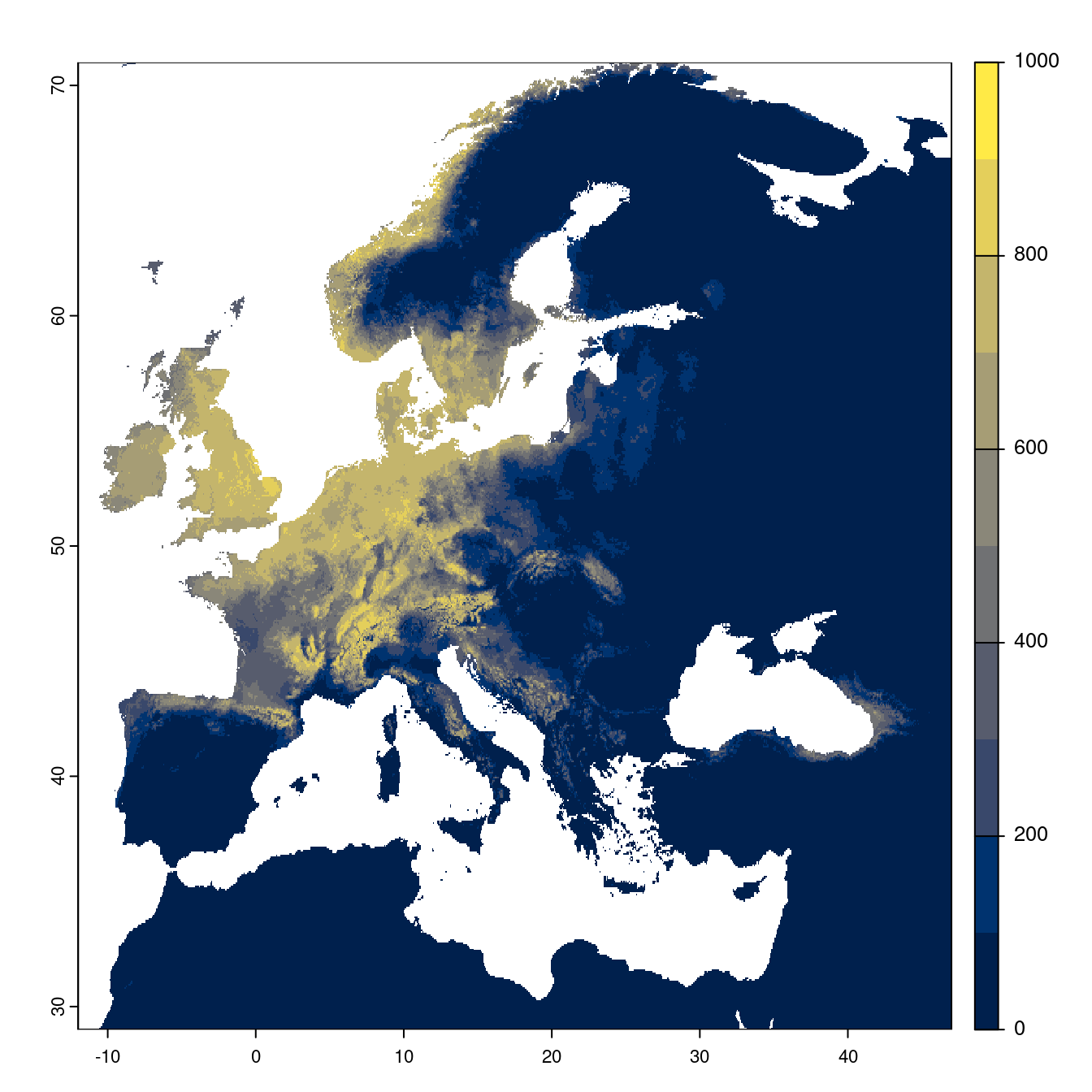
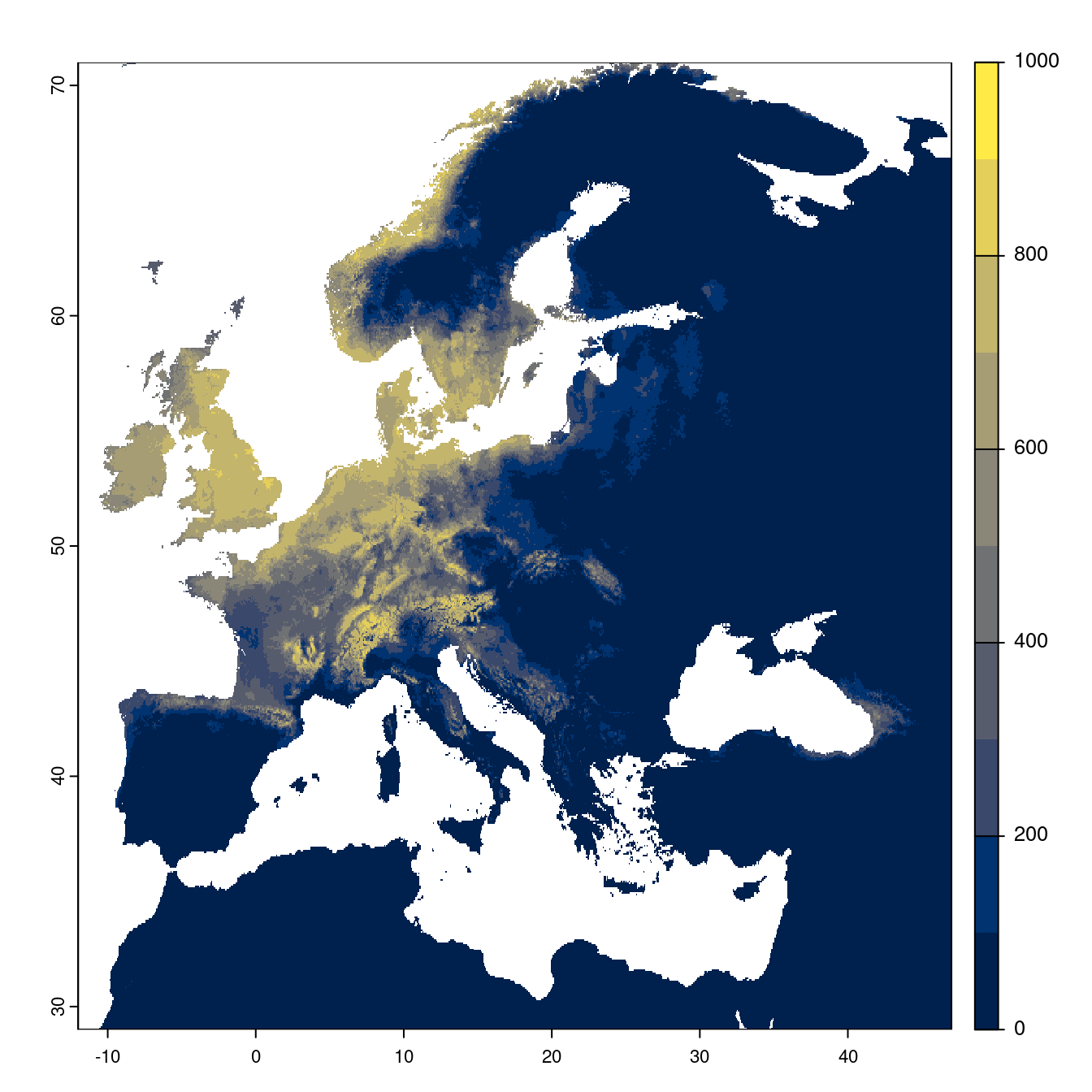
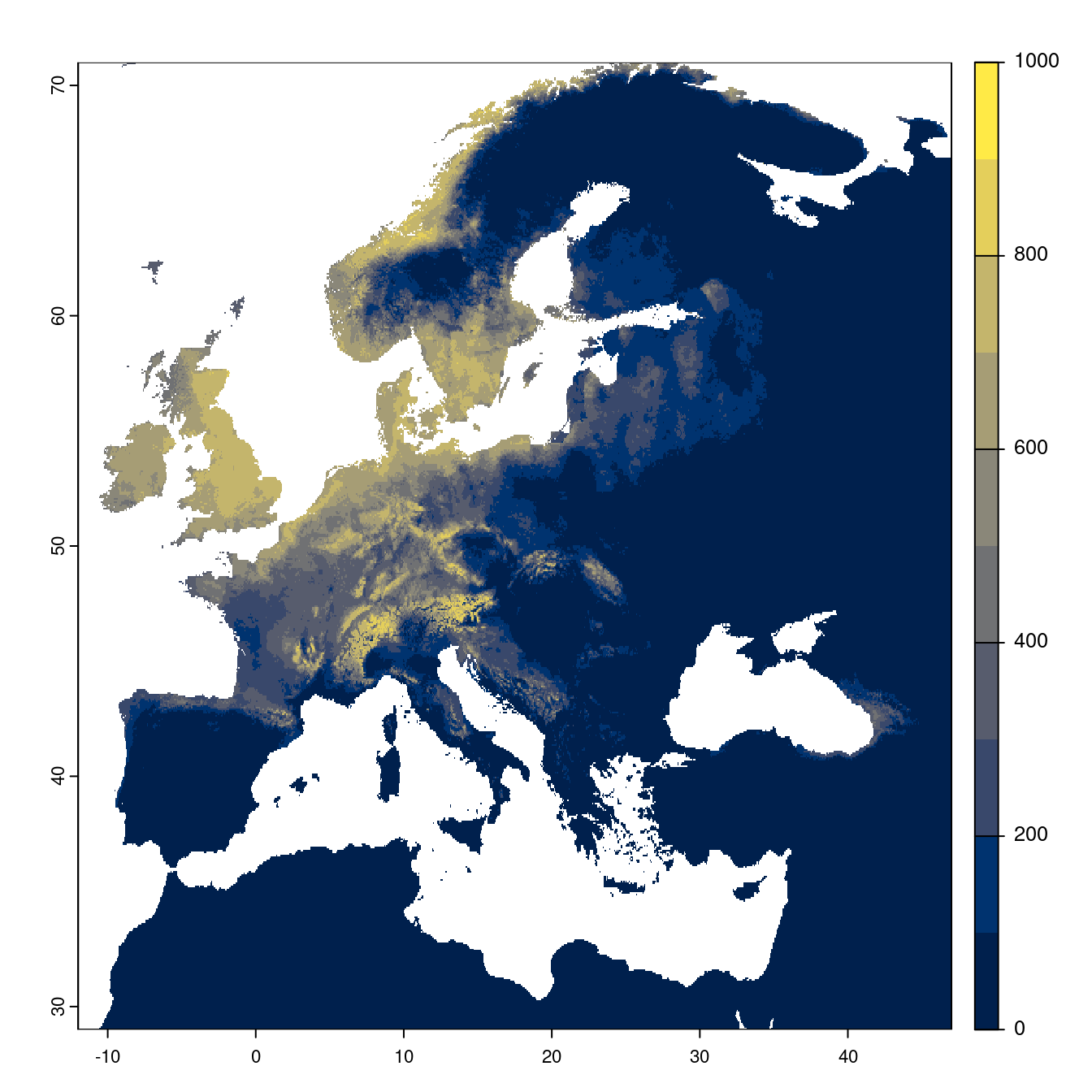
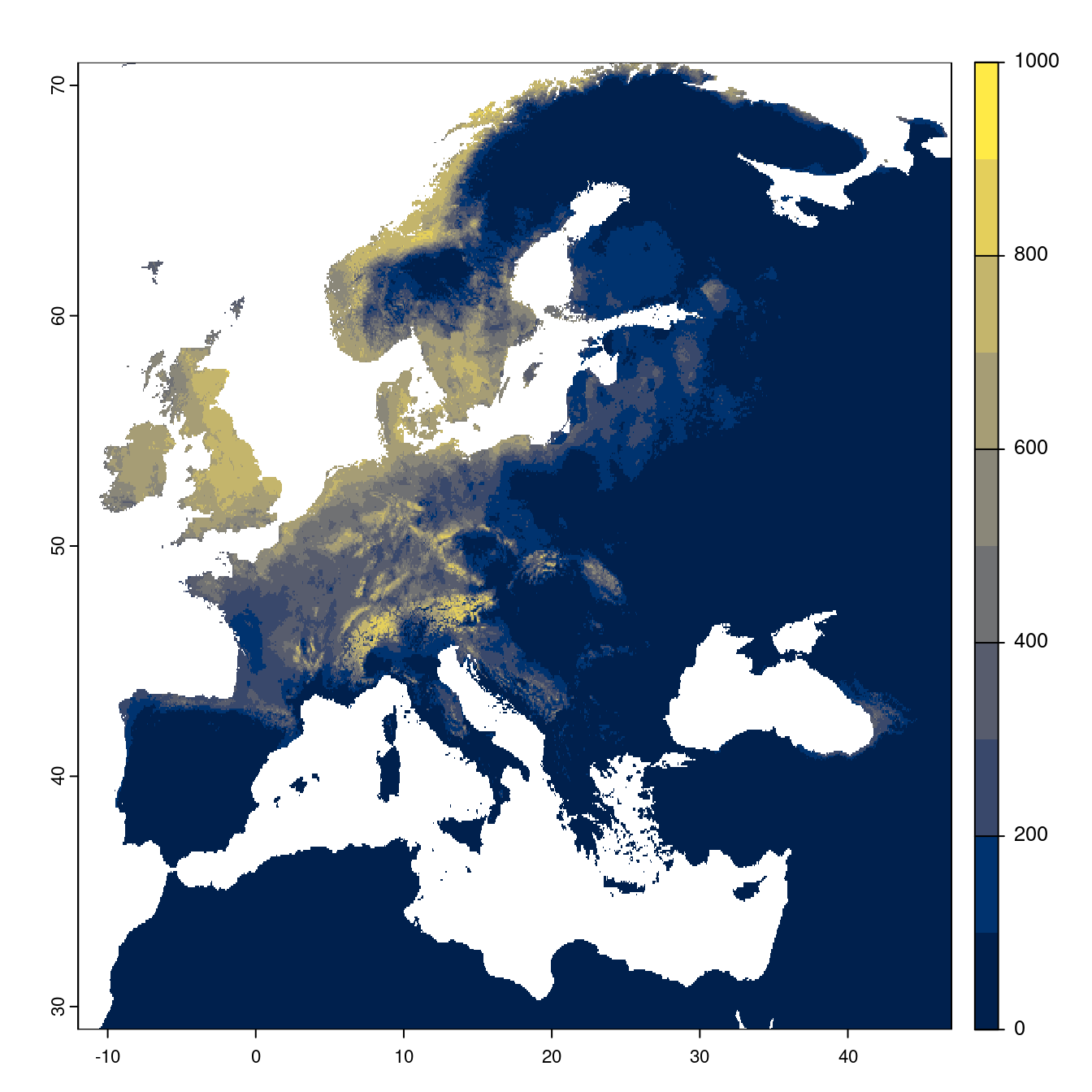
Figure 2.24: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP2-4.5 (modèle d’ensemble).
- SSP3-7.0
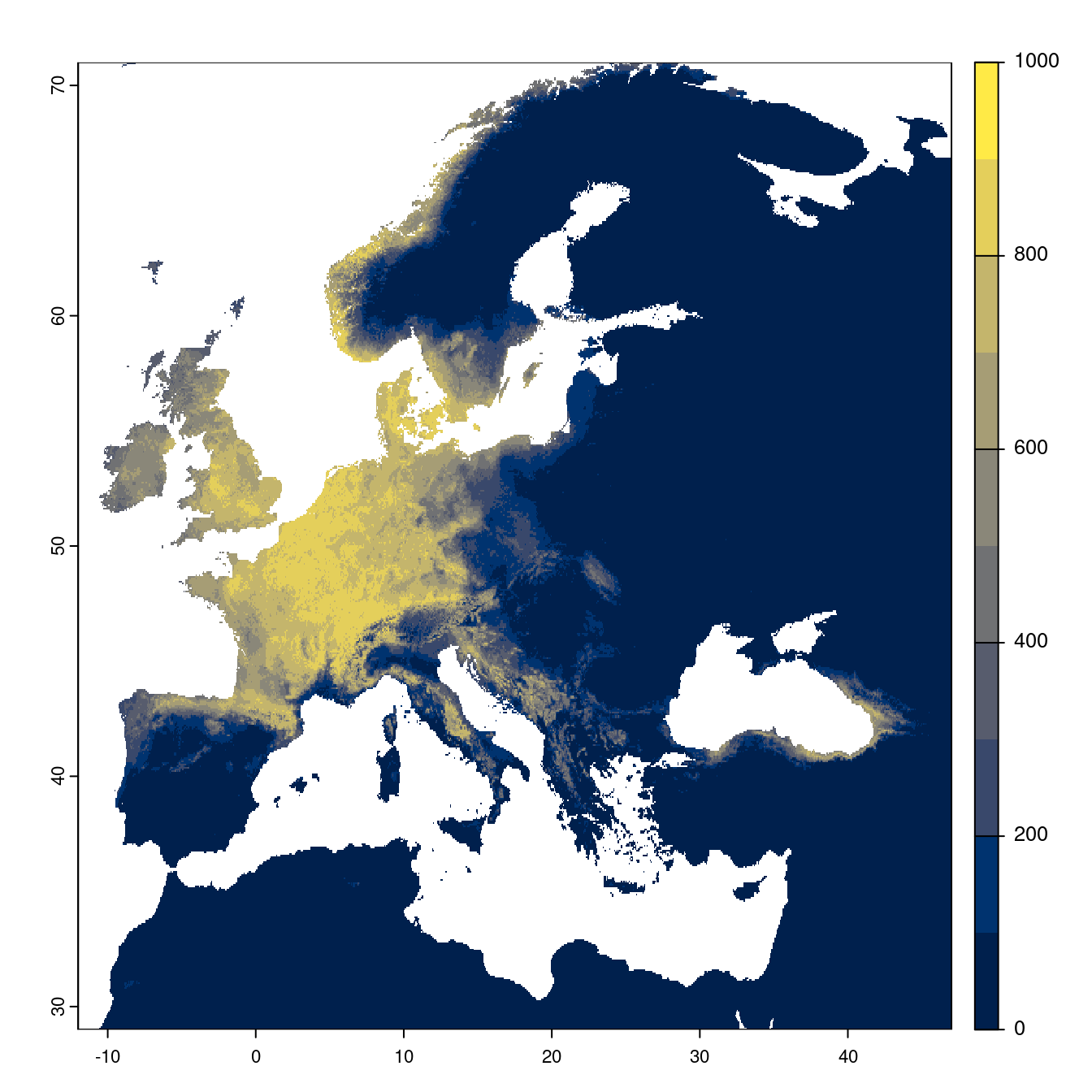
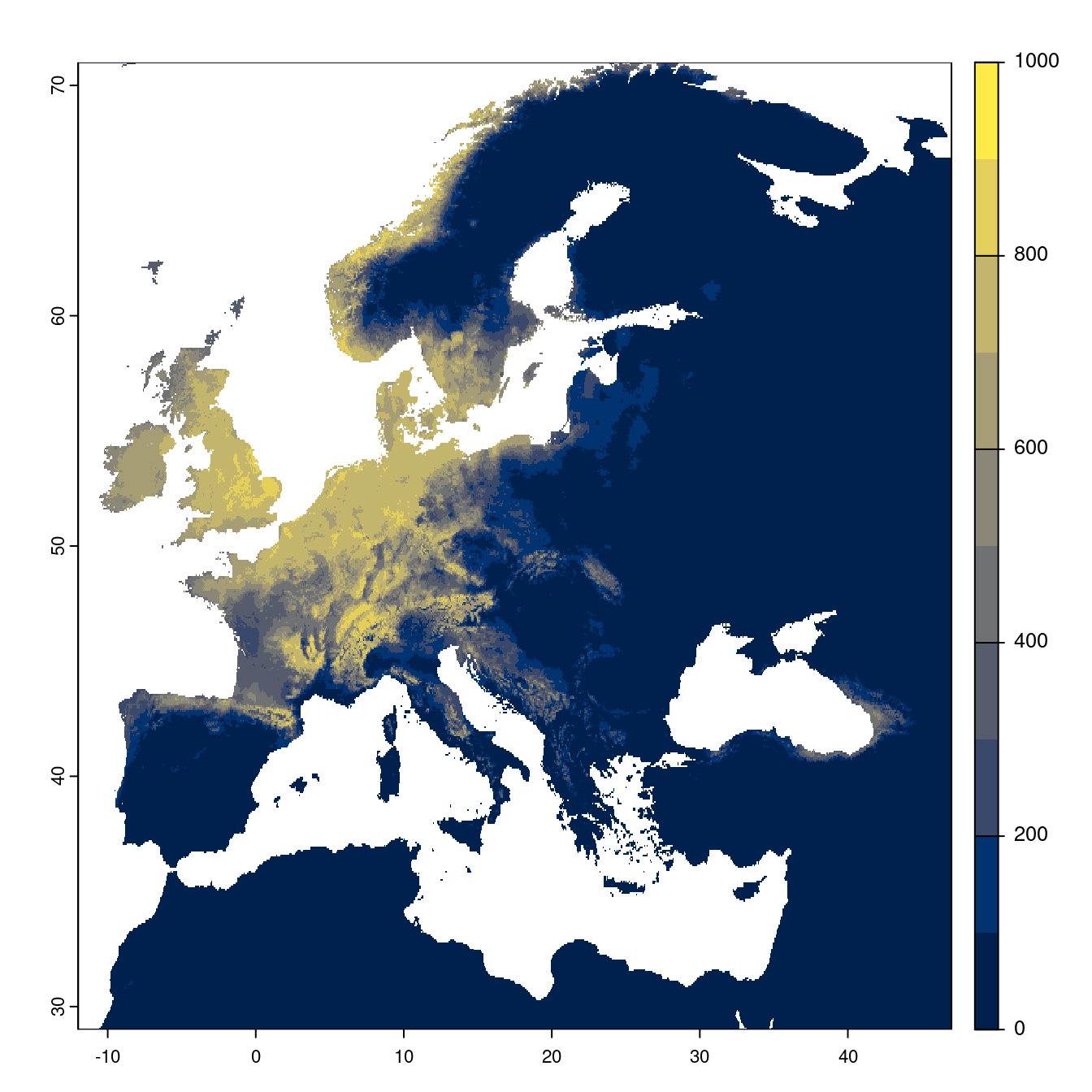
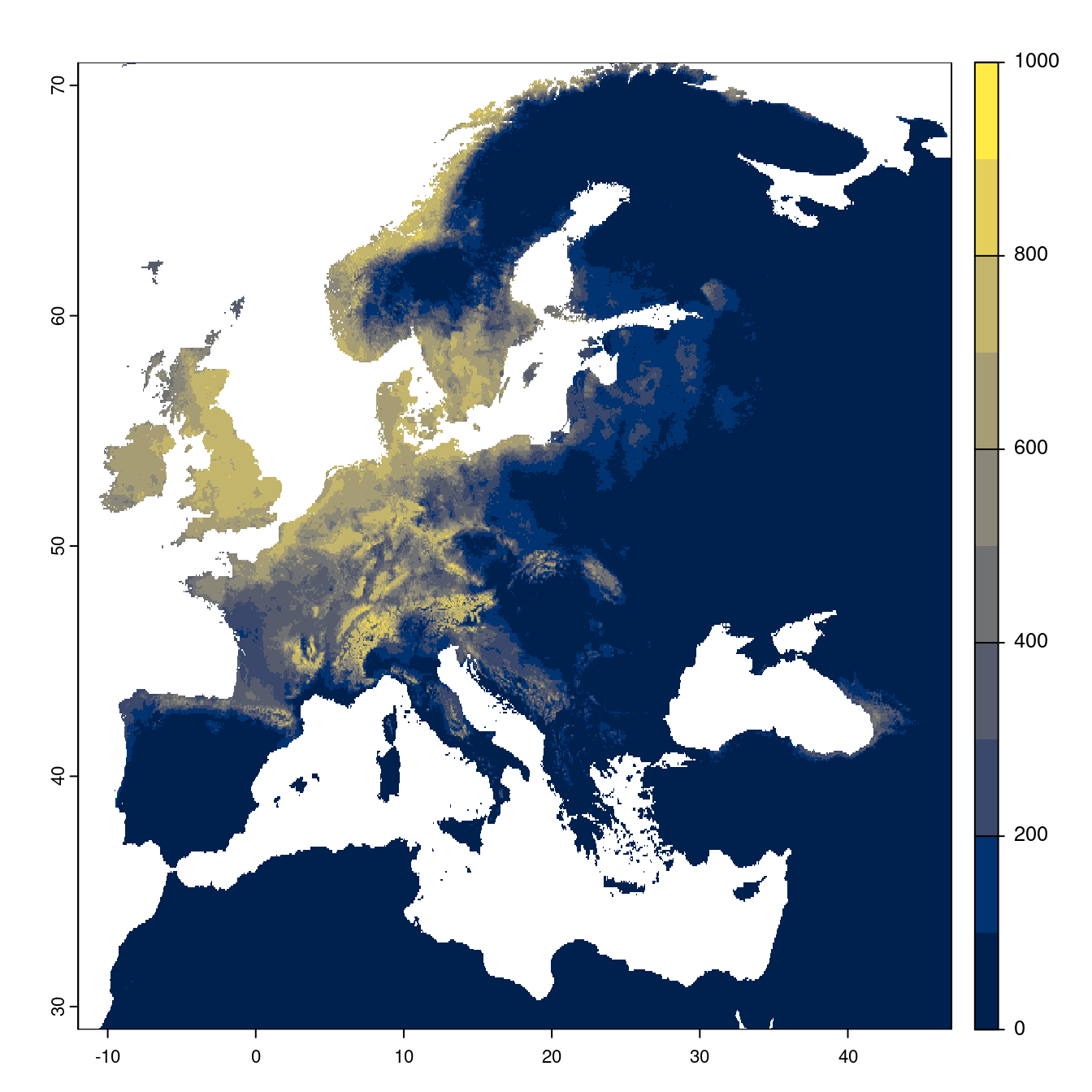
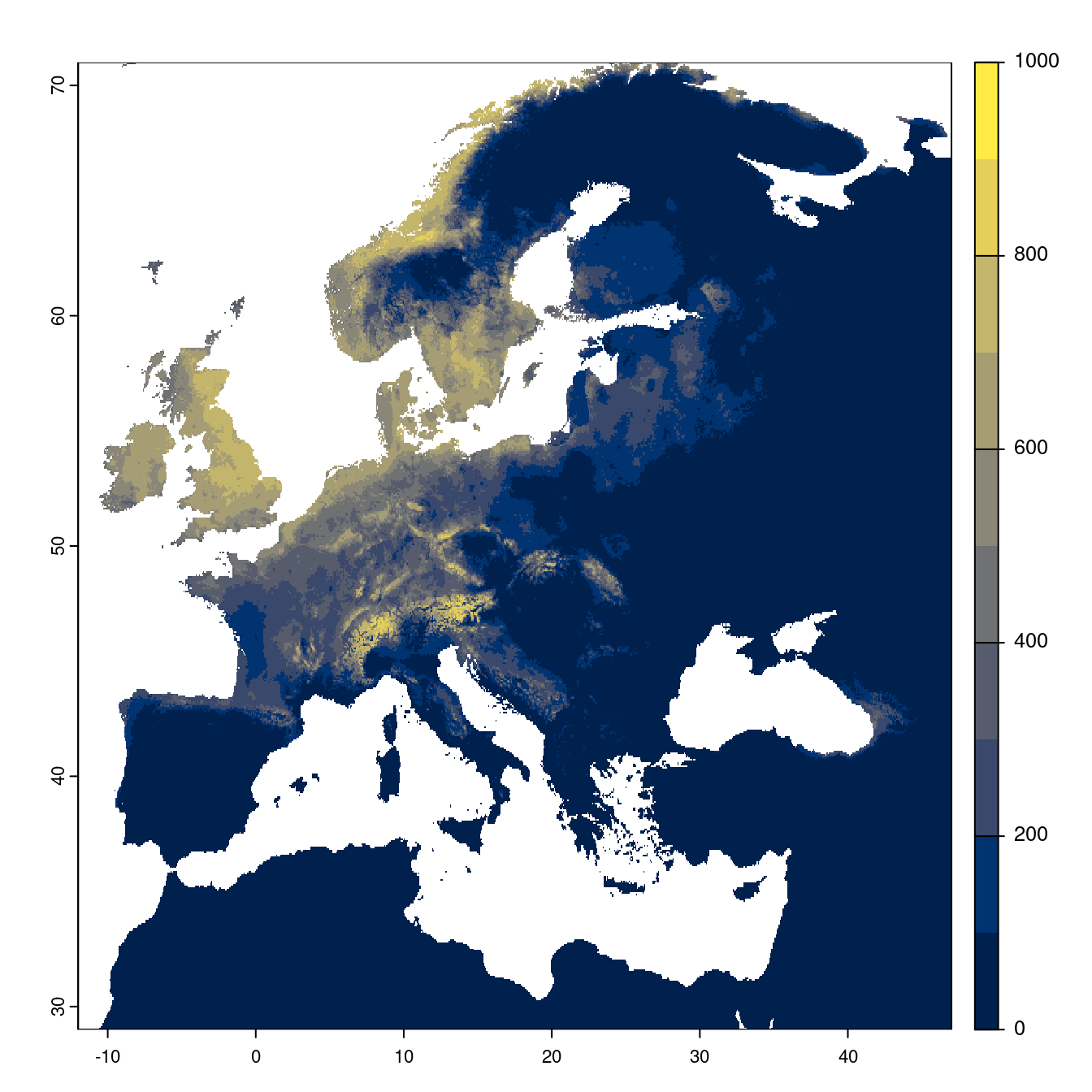
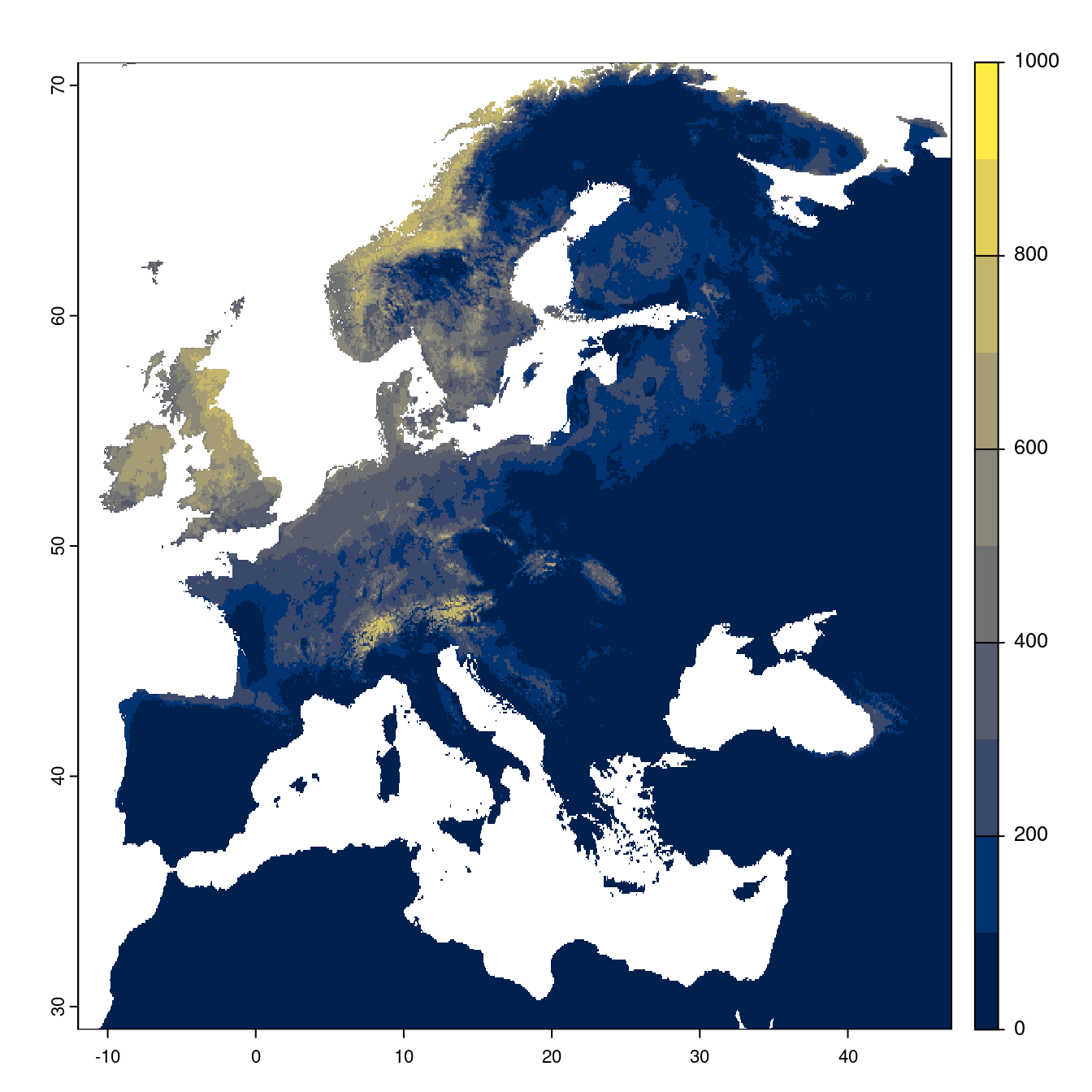
Figure 2.25: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP3-7.0 (modèle d’ensemble).
- SSP5-8.5
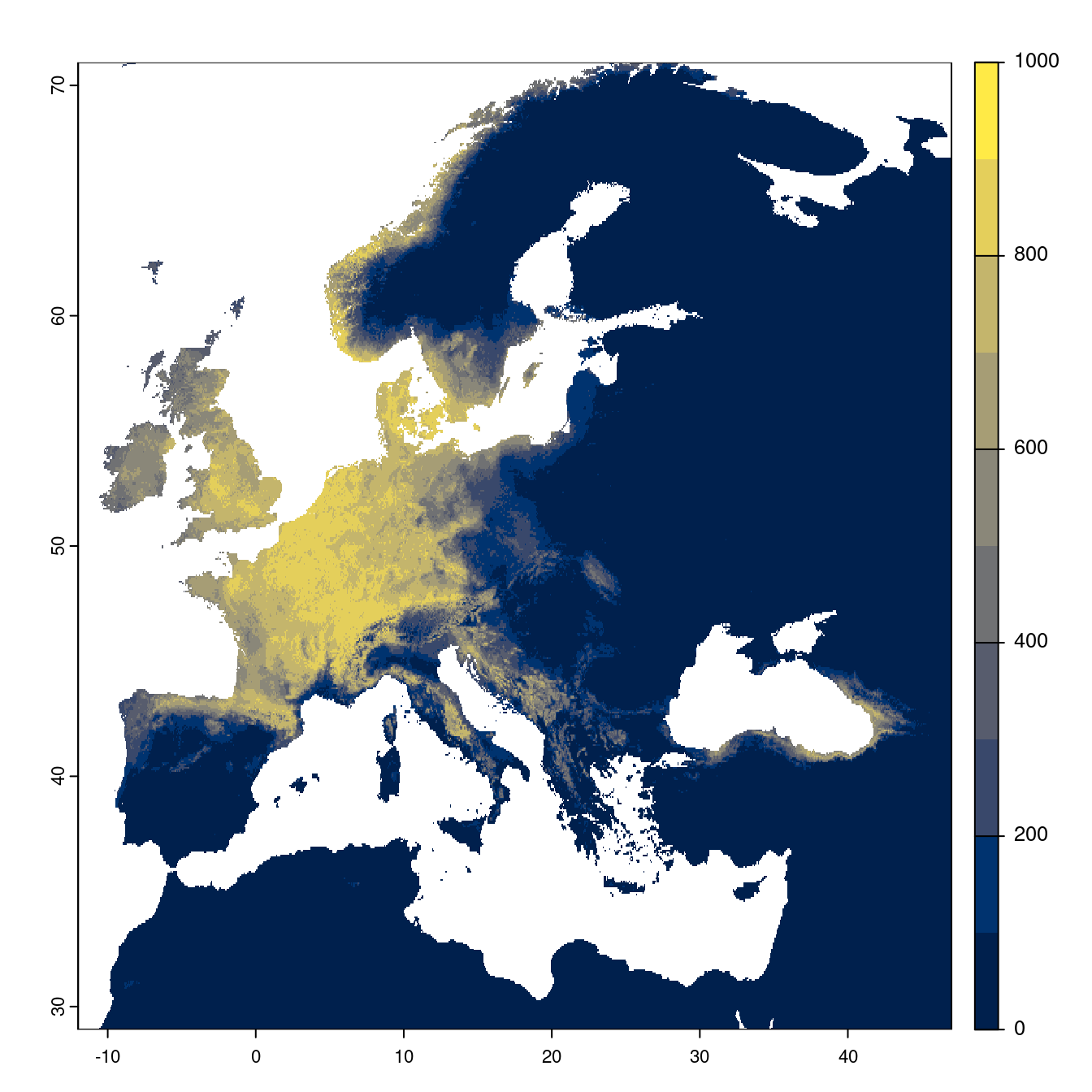
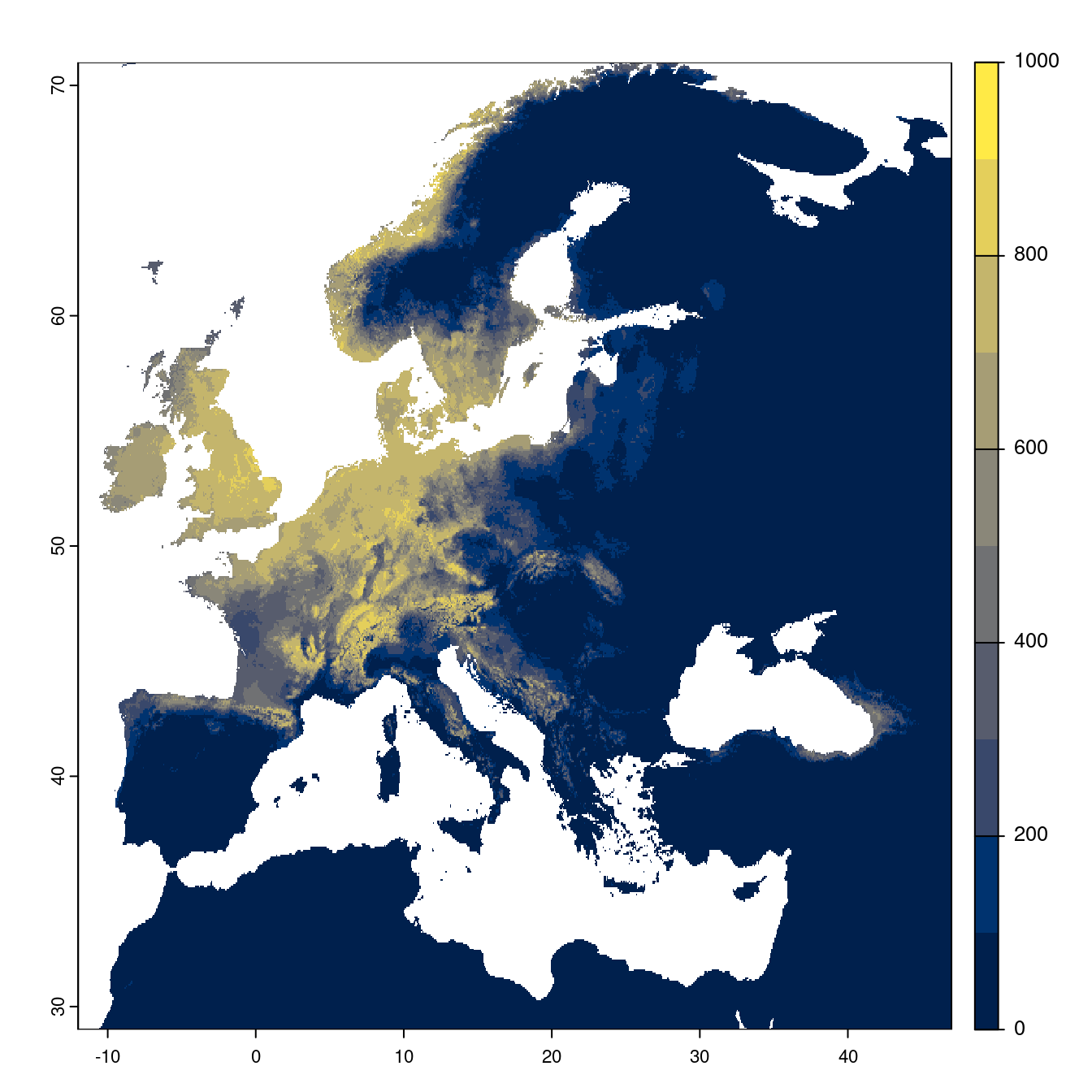
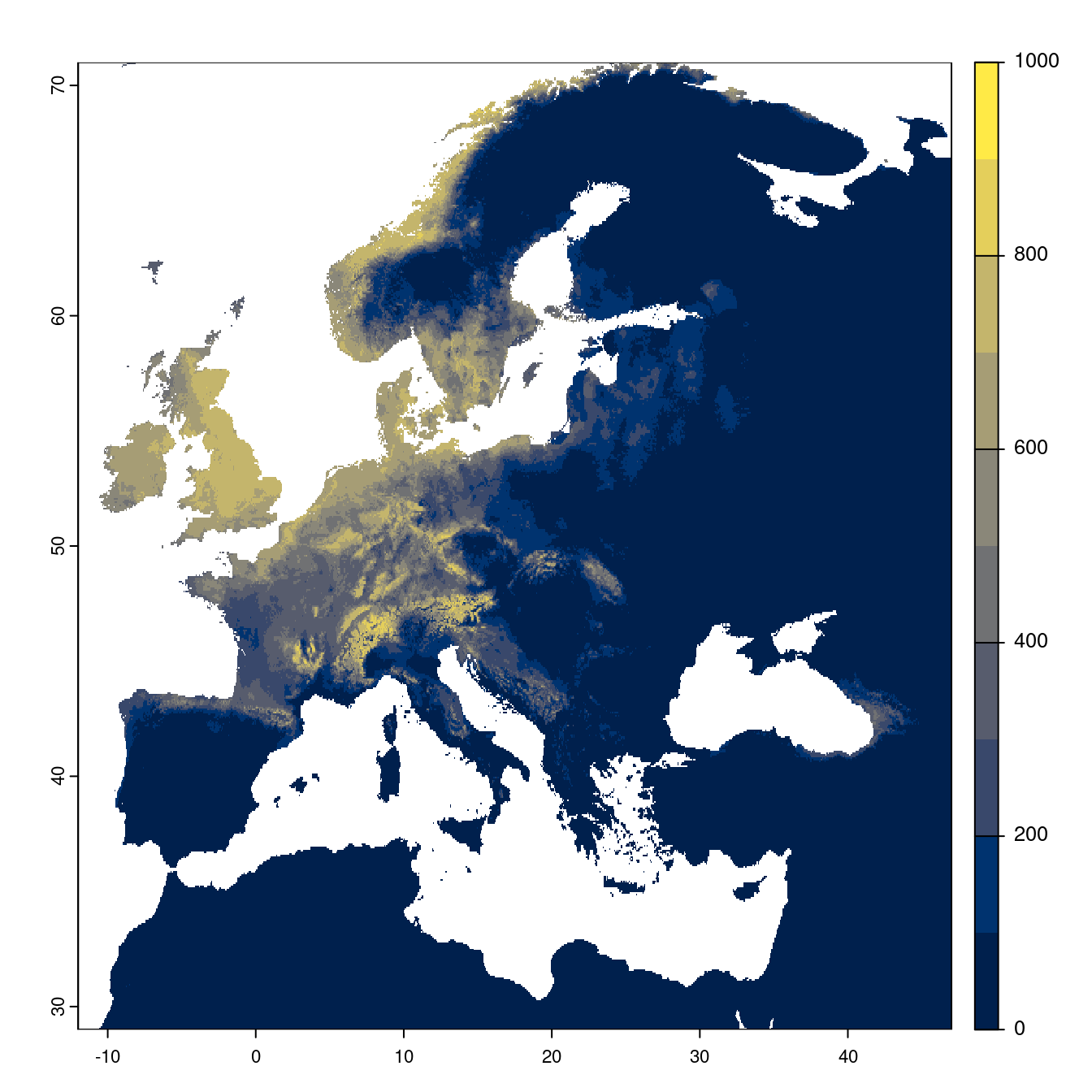
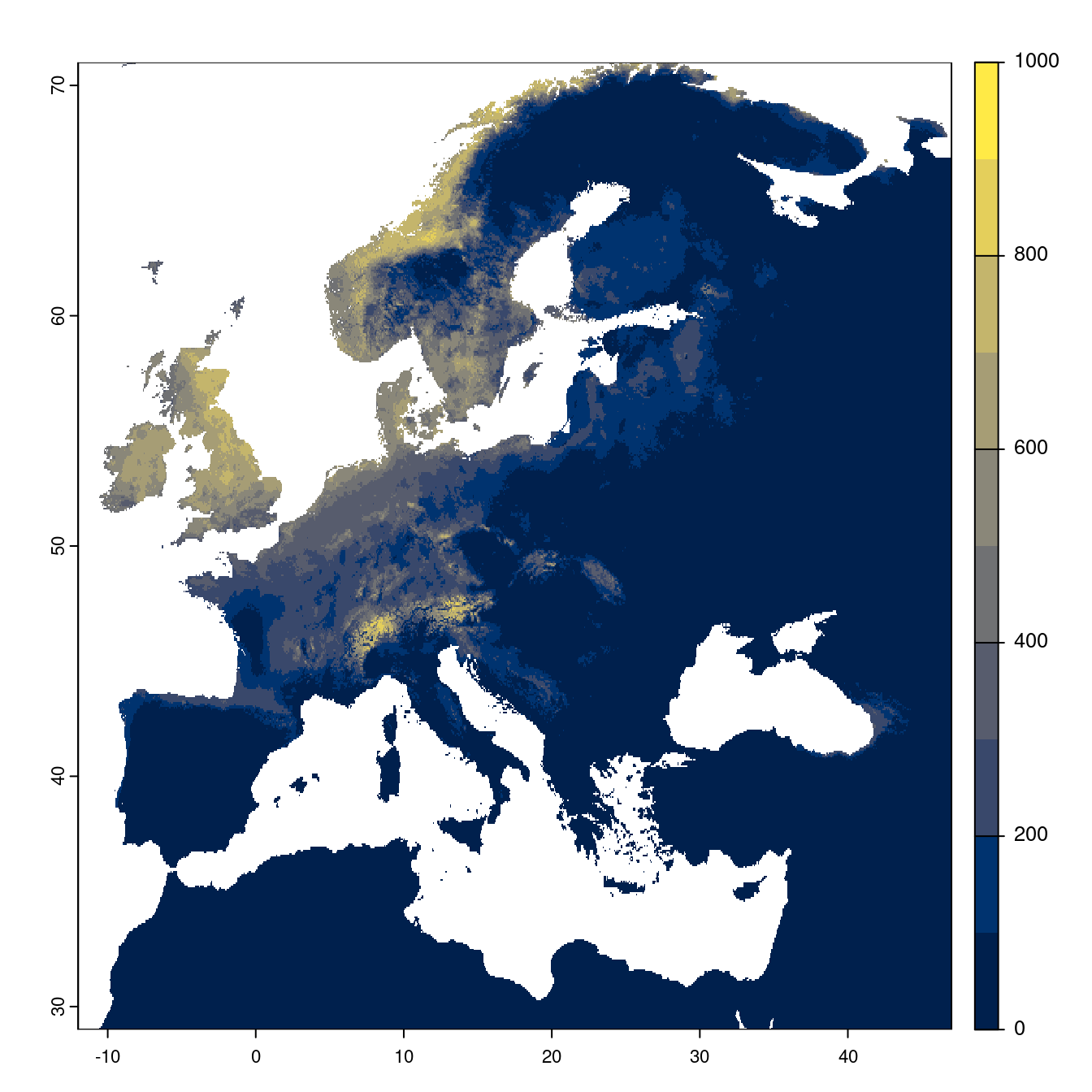
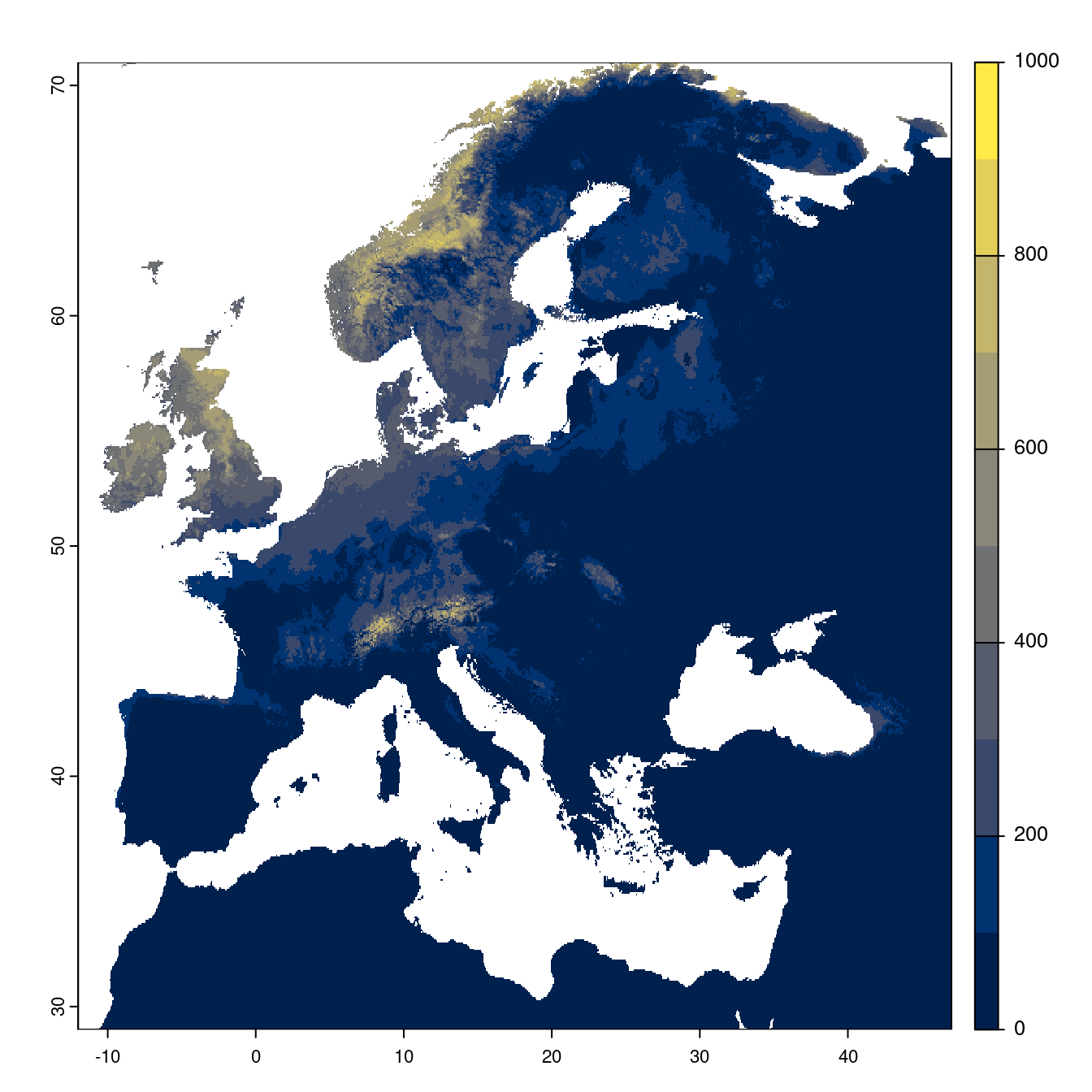
Figure 2.26: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble).
2.6 Inférence à l’échelle de la métropole
- Espèces potentielles pour Grenoble (forte probabilité de présence à l’horizon 2100)
- Croisement avec températures de surface 2019 (fournie par la métropole)
- → conditions climatiques favorables à fine échelle
à peu près 7 pixels × 9 (WorldClim)
2.6.1 Distributions potentielles futures
Même chose que précédemment mais sur les rasters de Grenoble
- SSP1-2.6

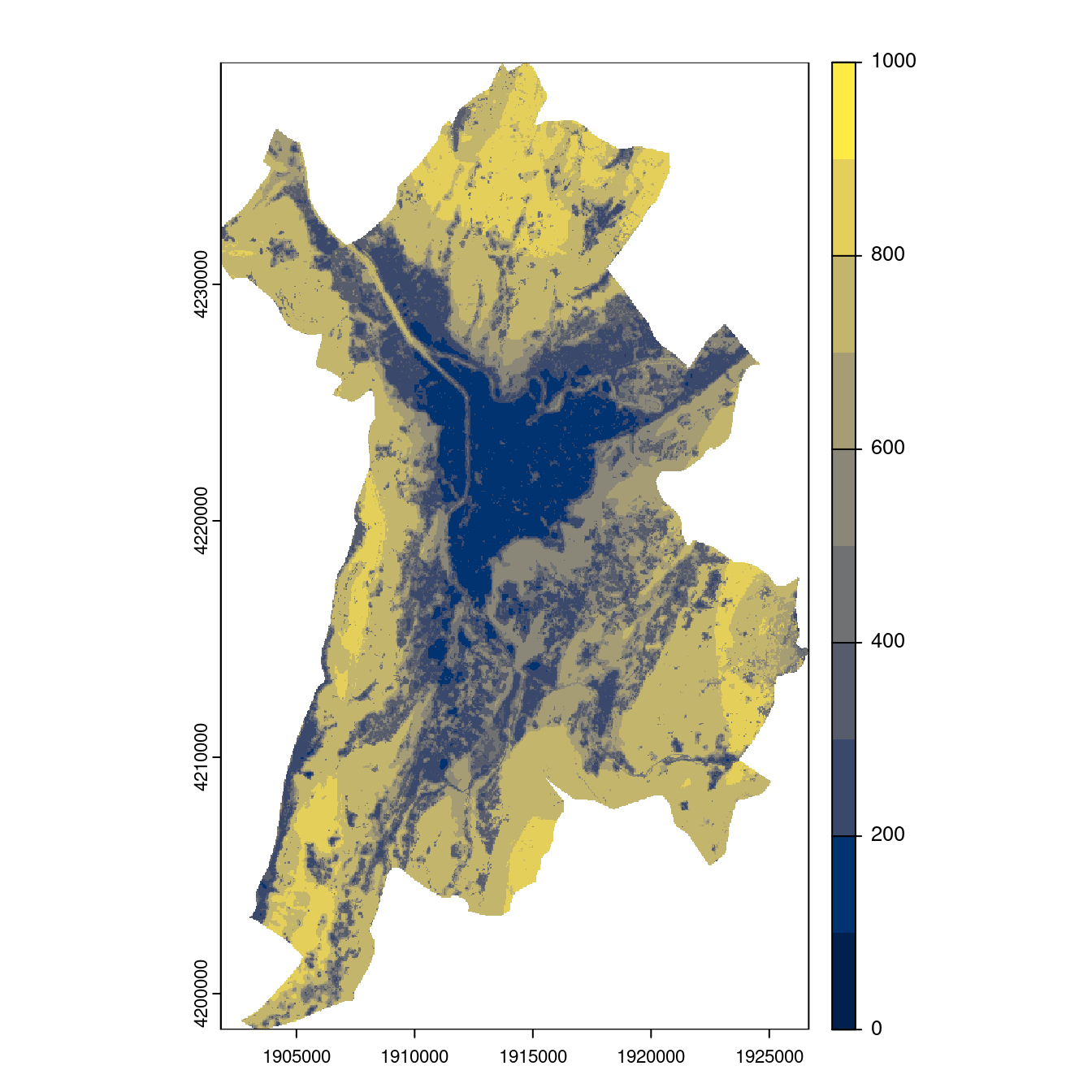
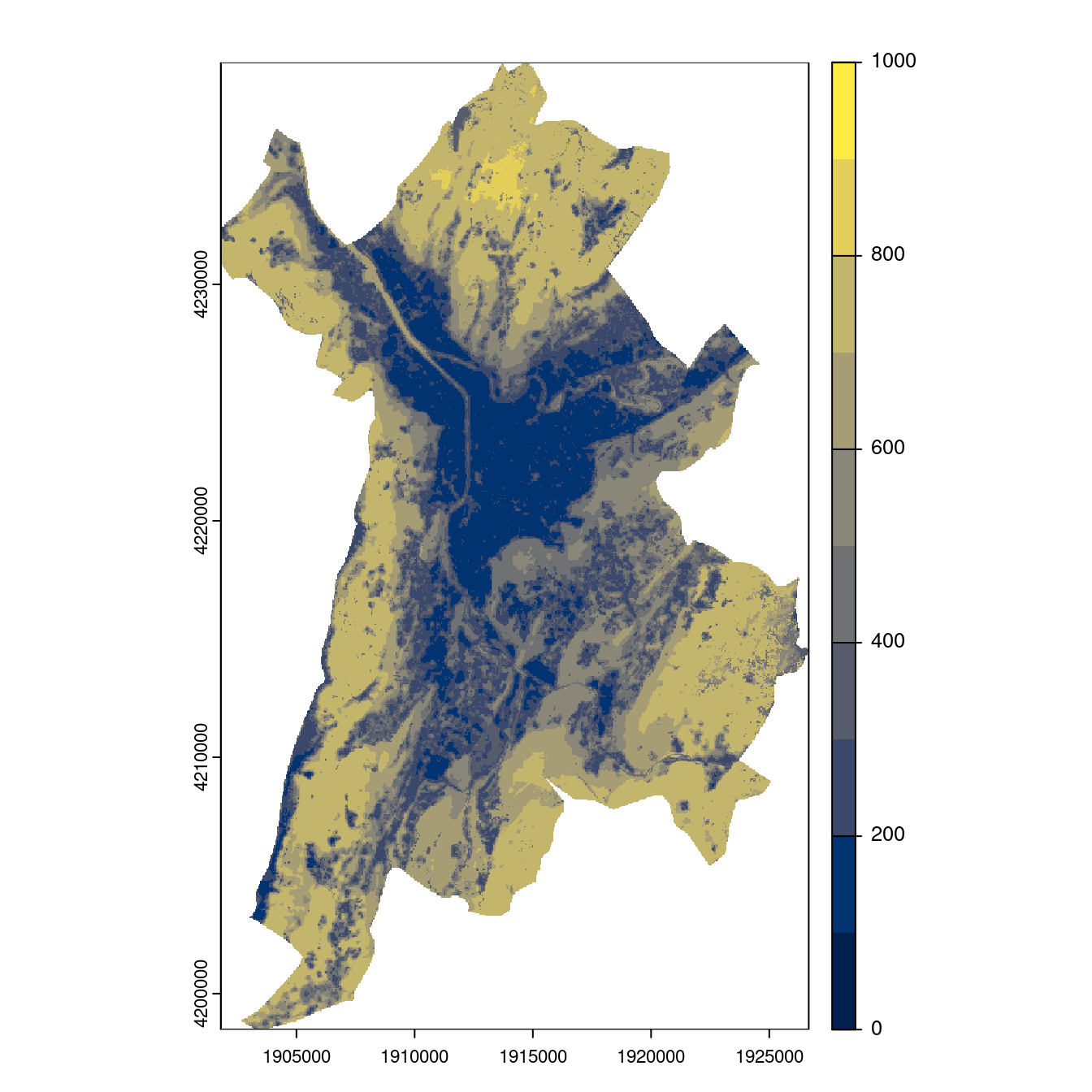
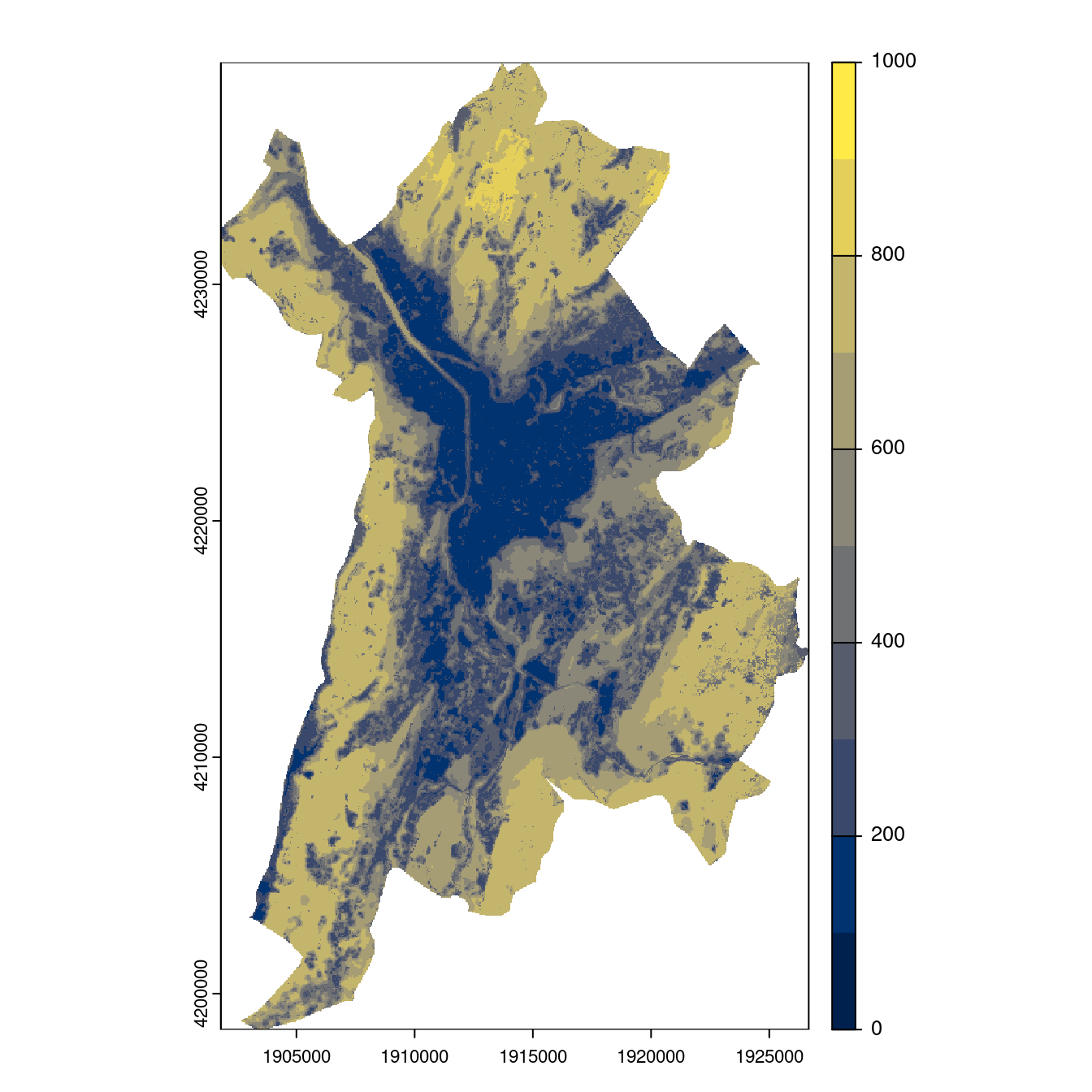
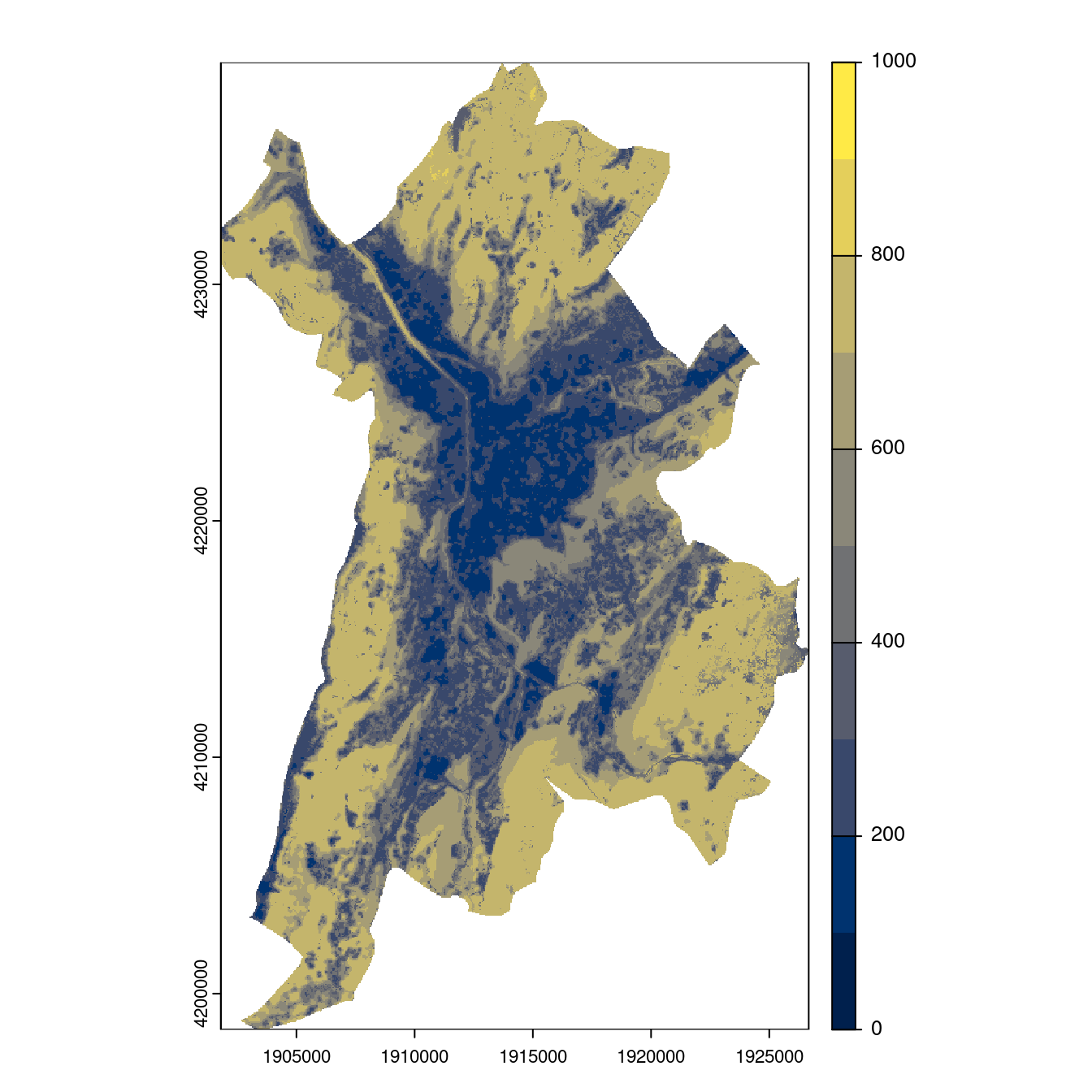
Figure 2.27: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP2-4.5

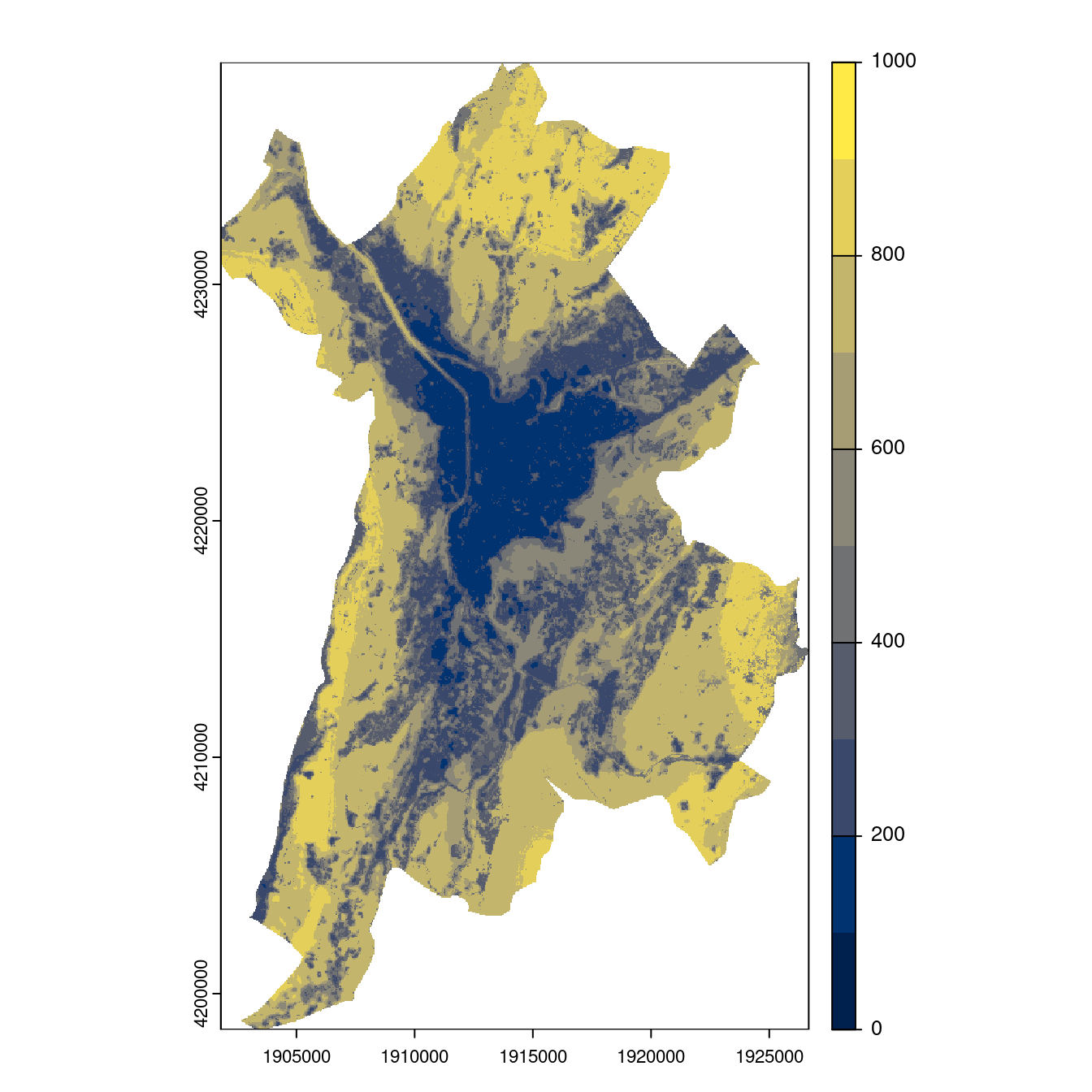
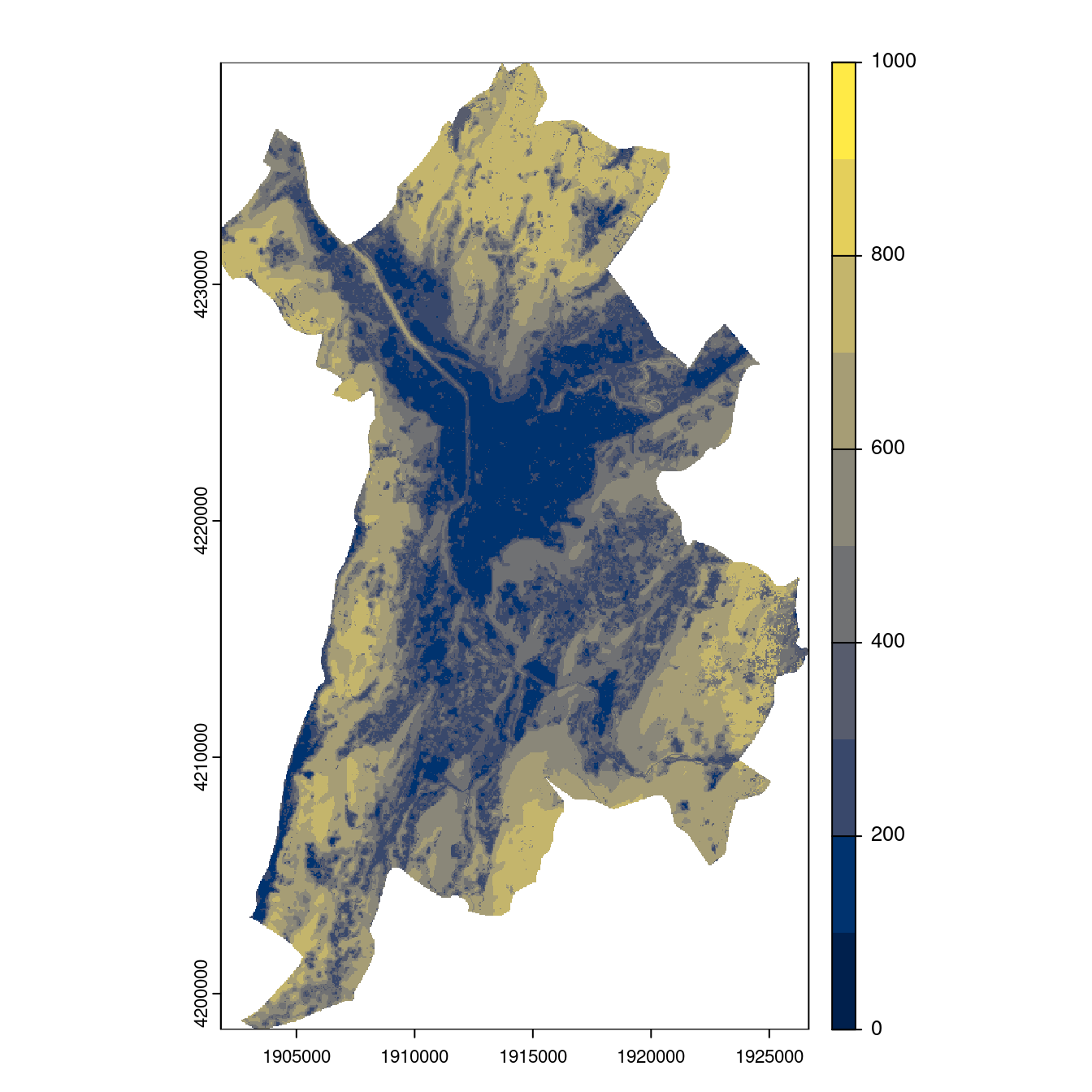
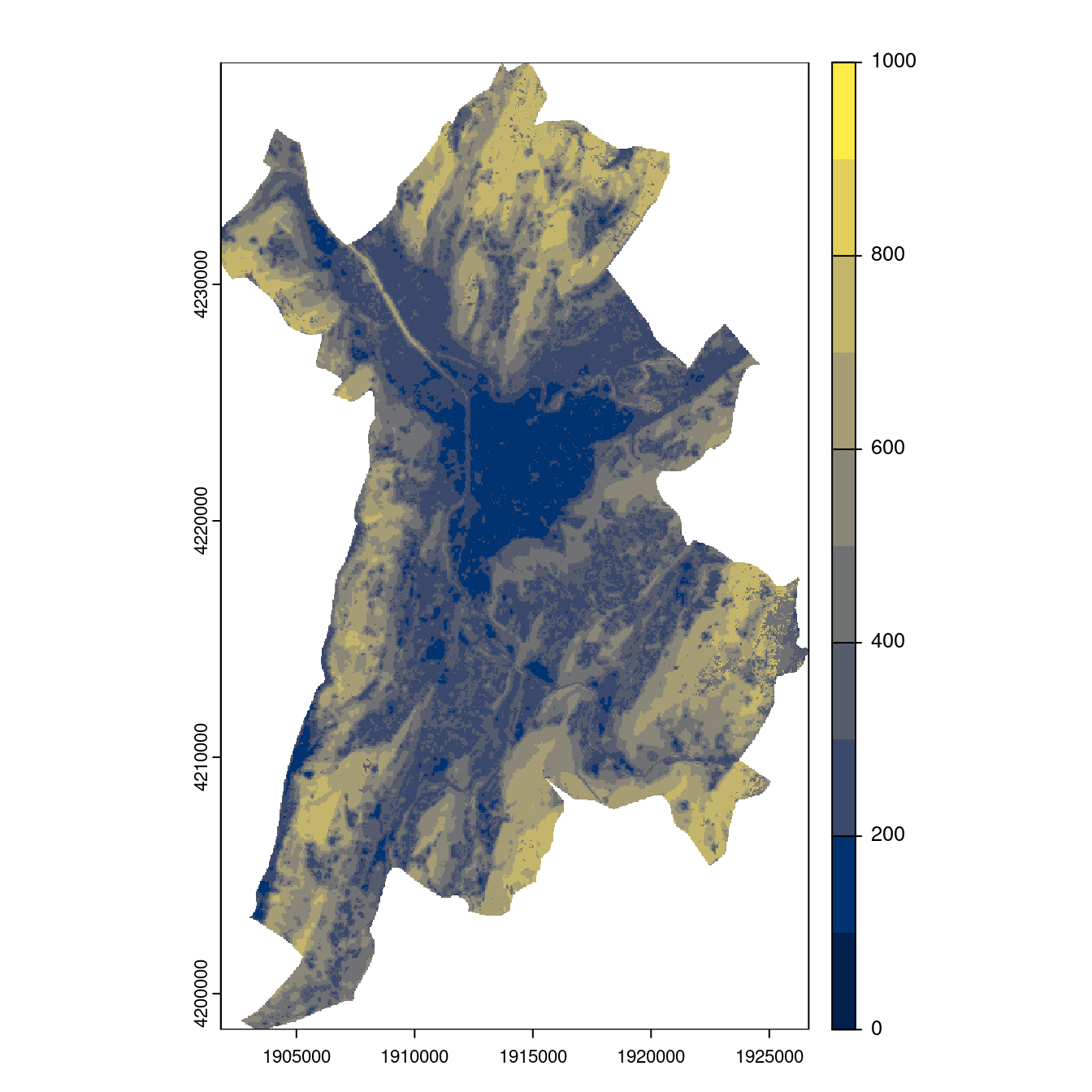
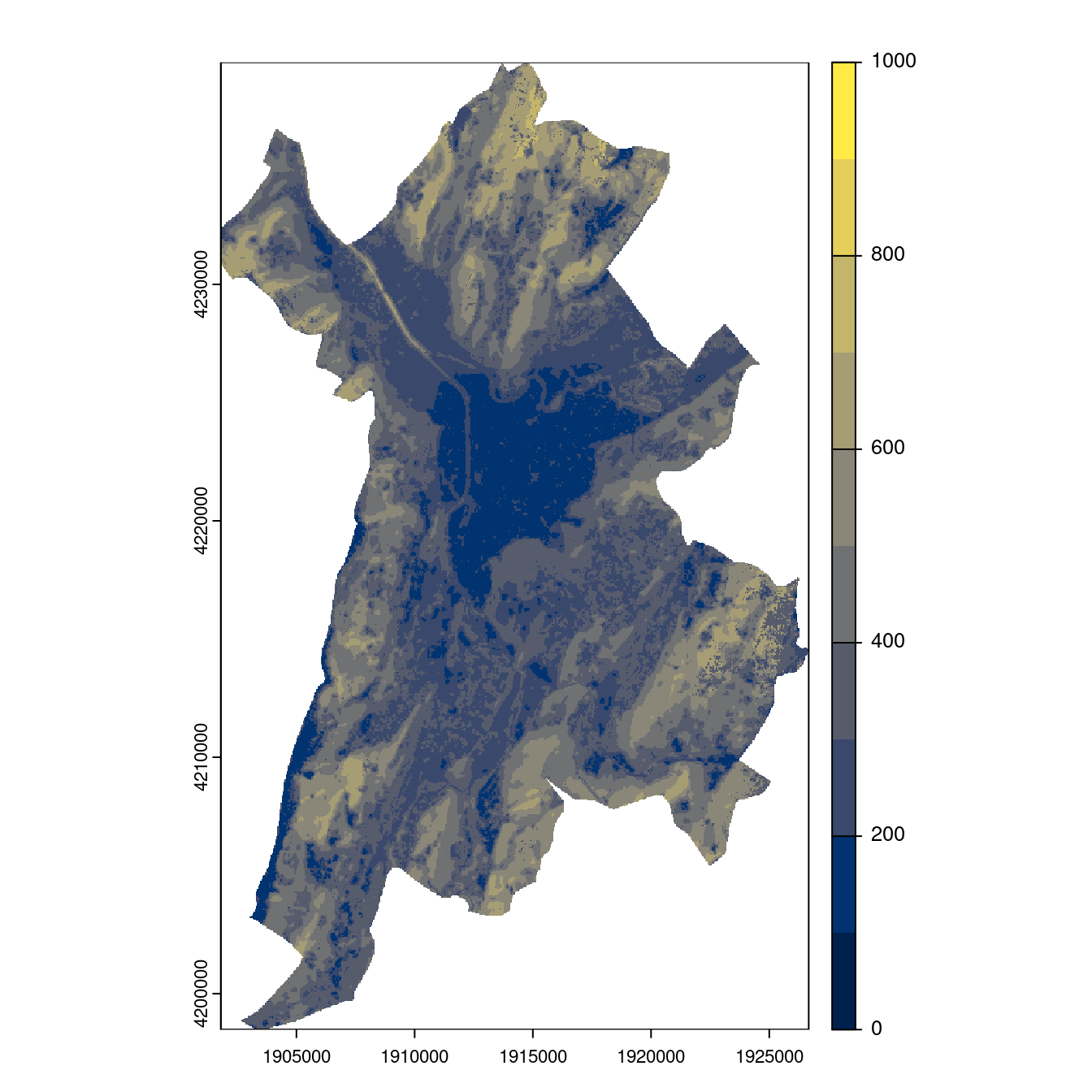
Figure 2.28: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP3-7.0
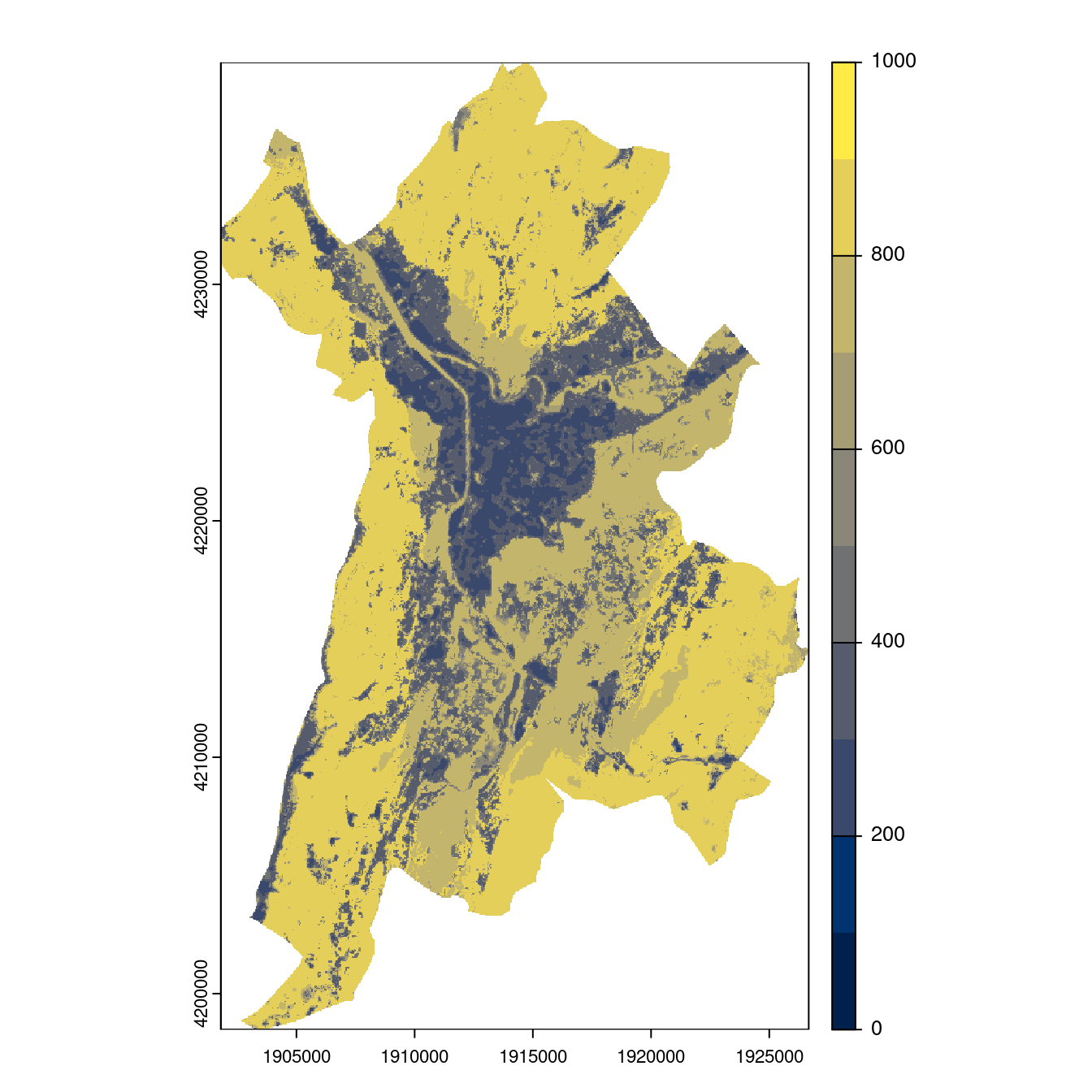
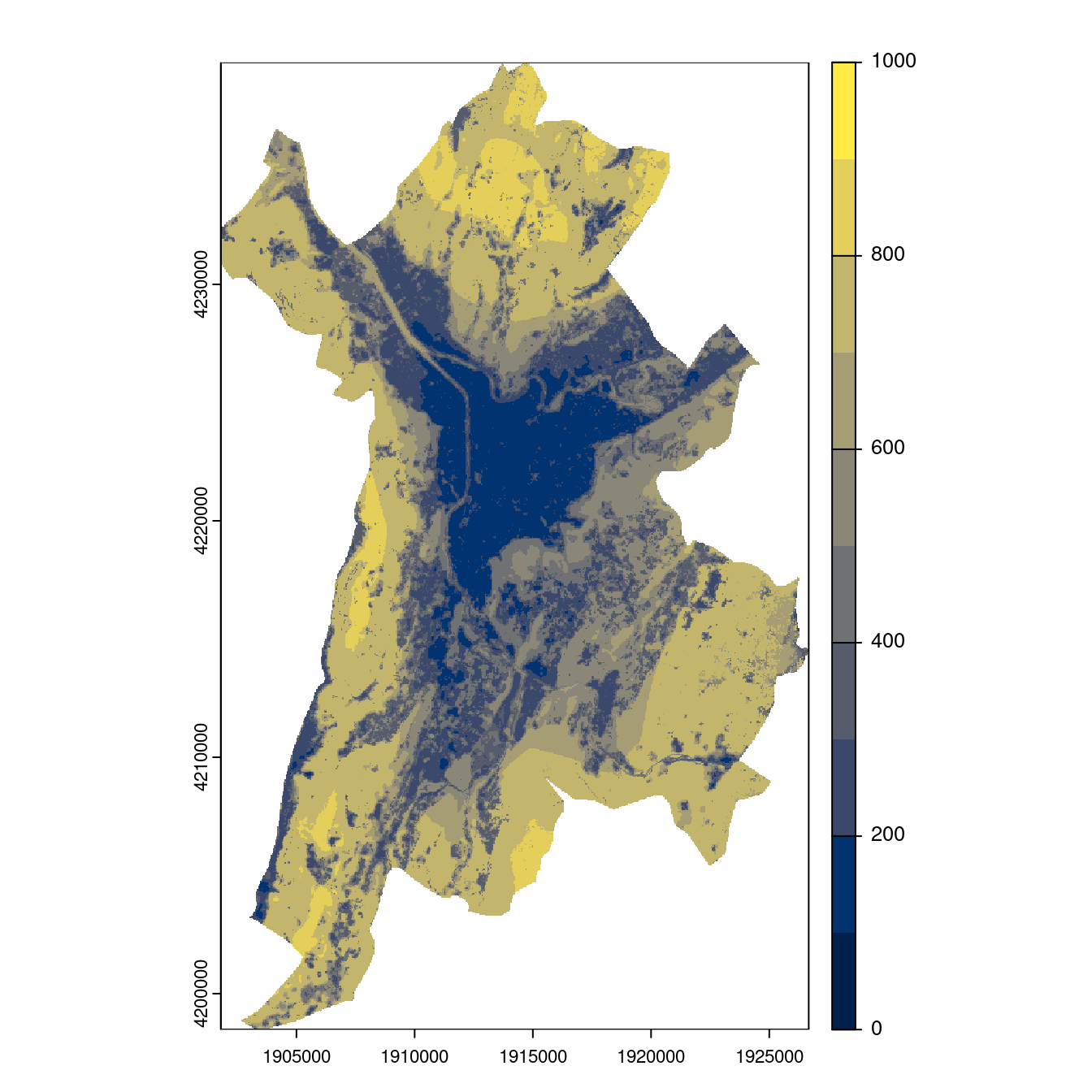
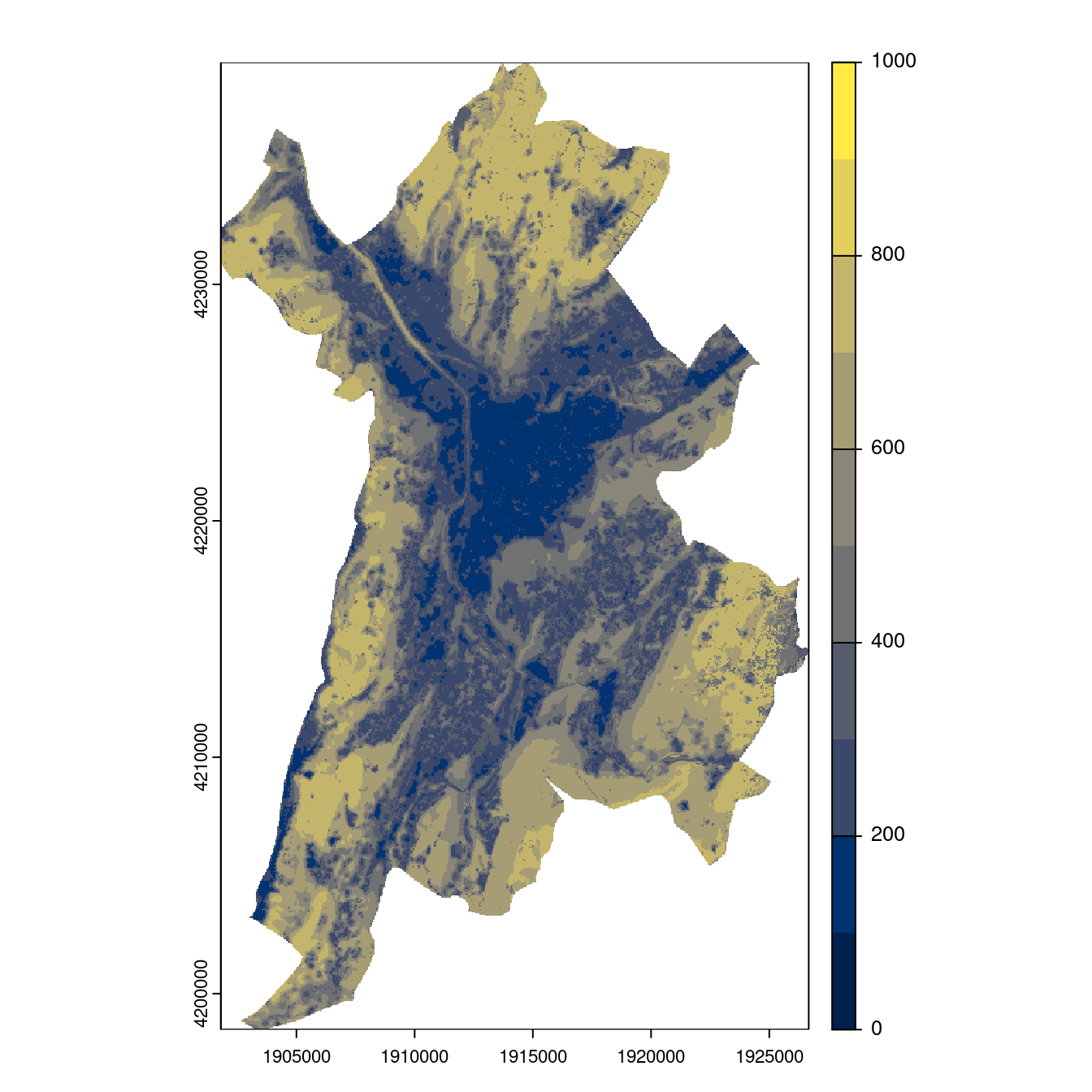
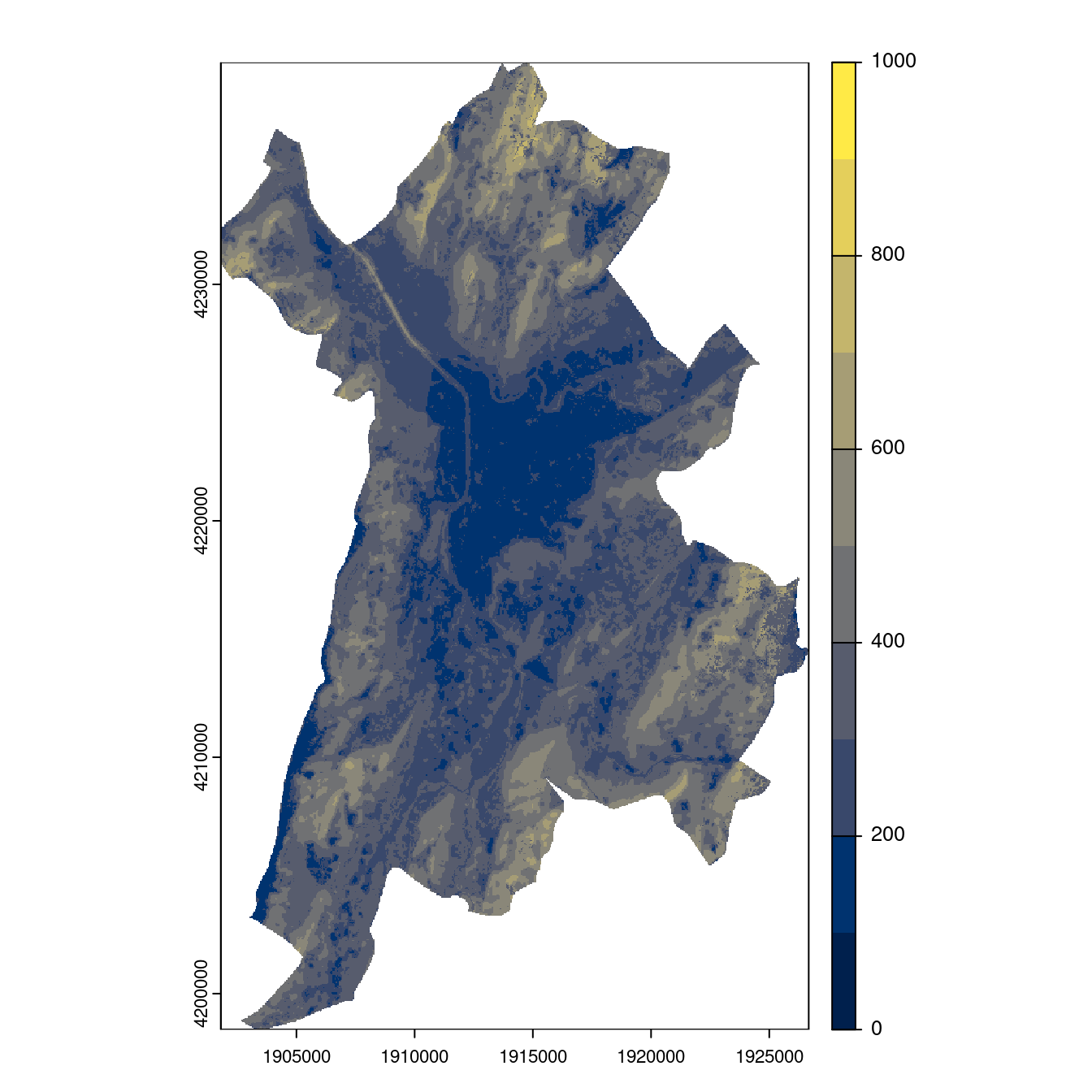
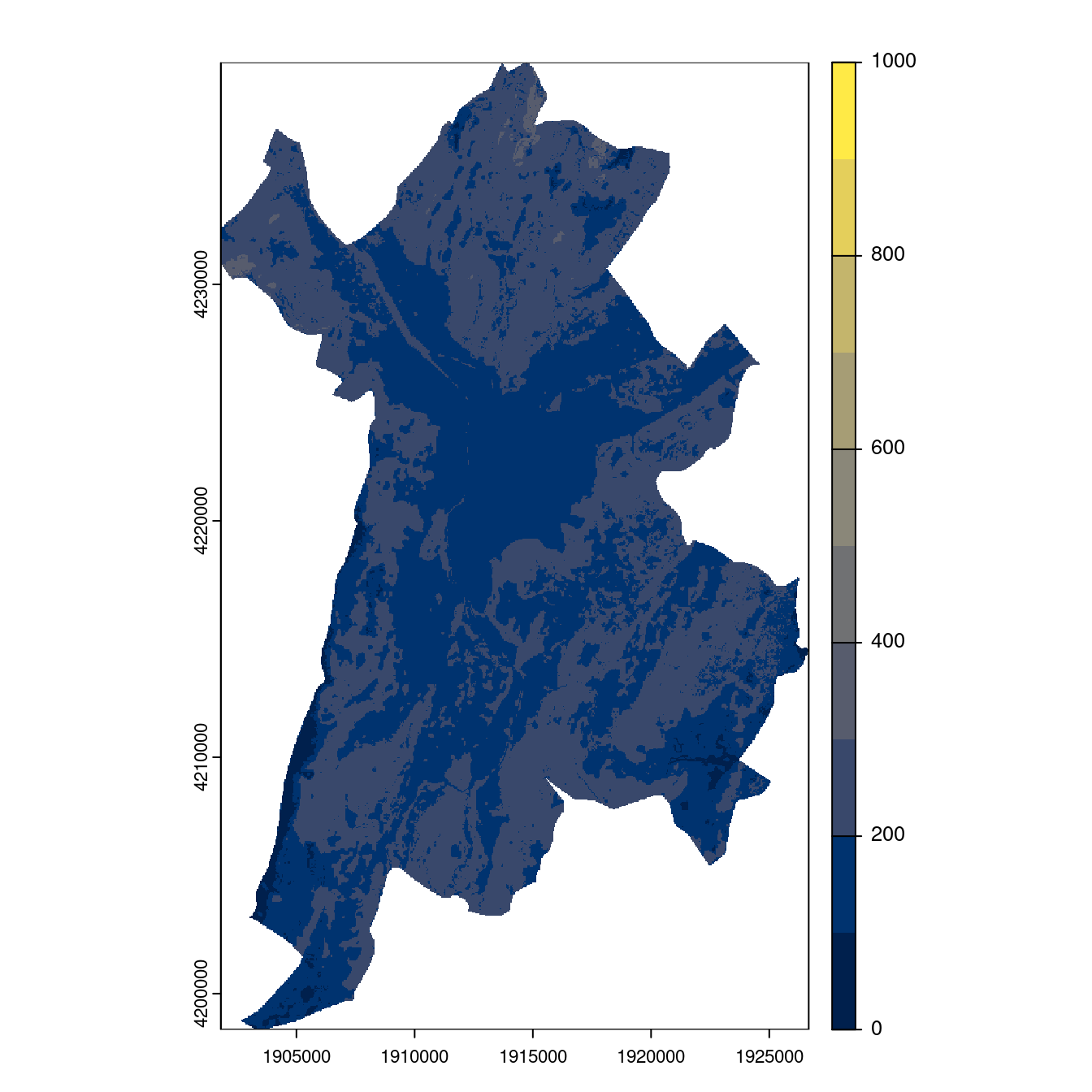
Figure 2.29: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP5-8.5
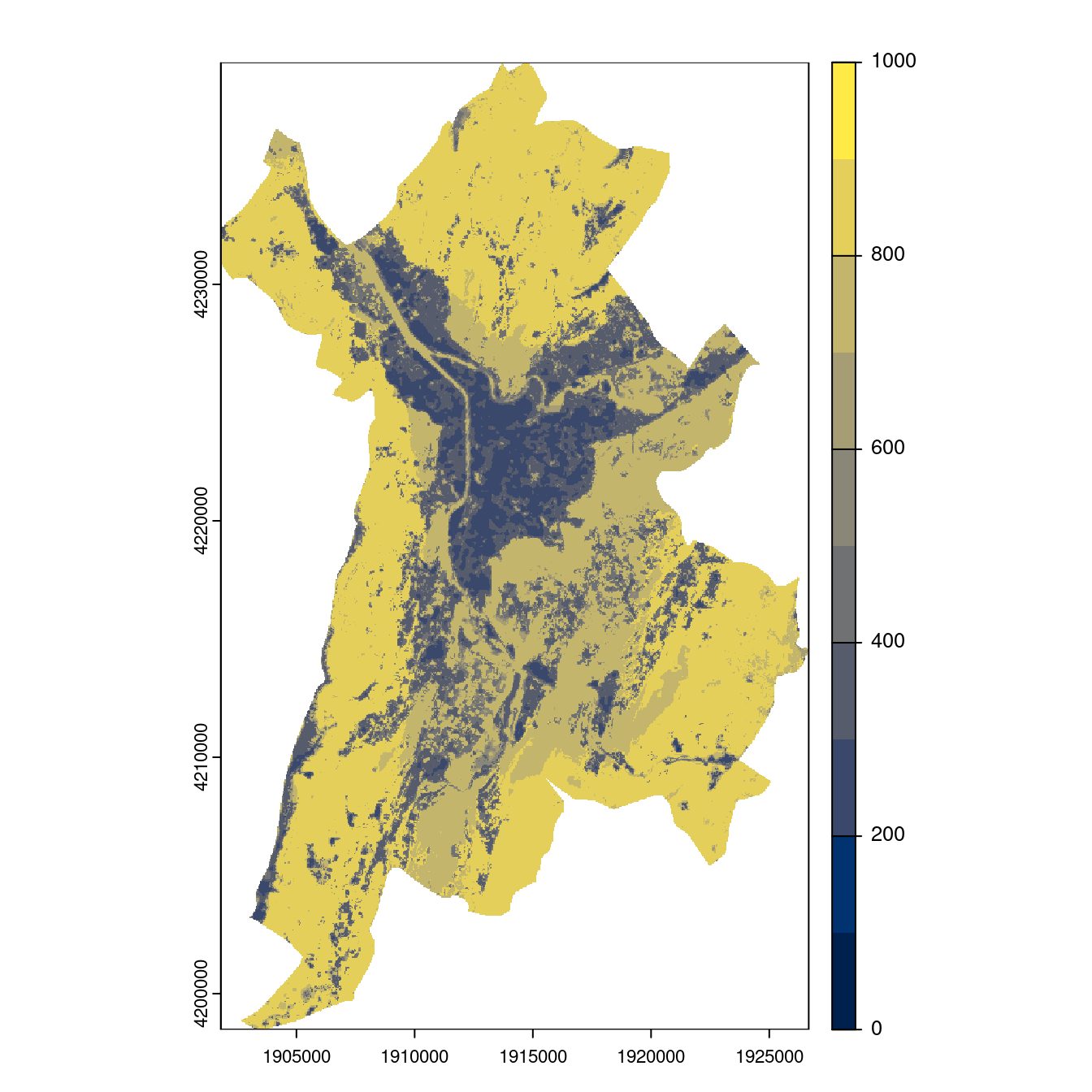

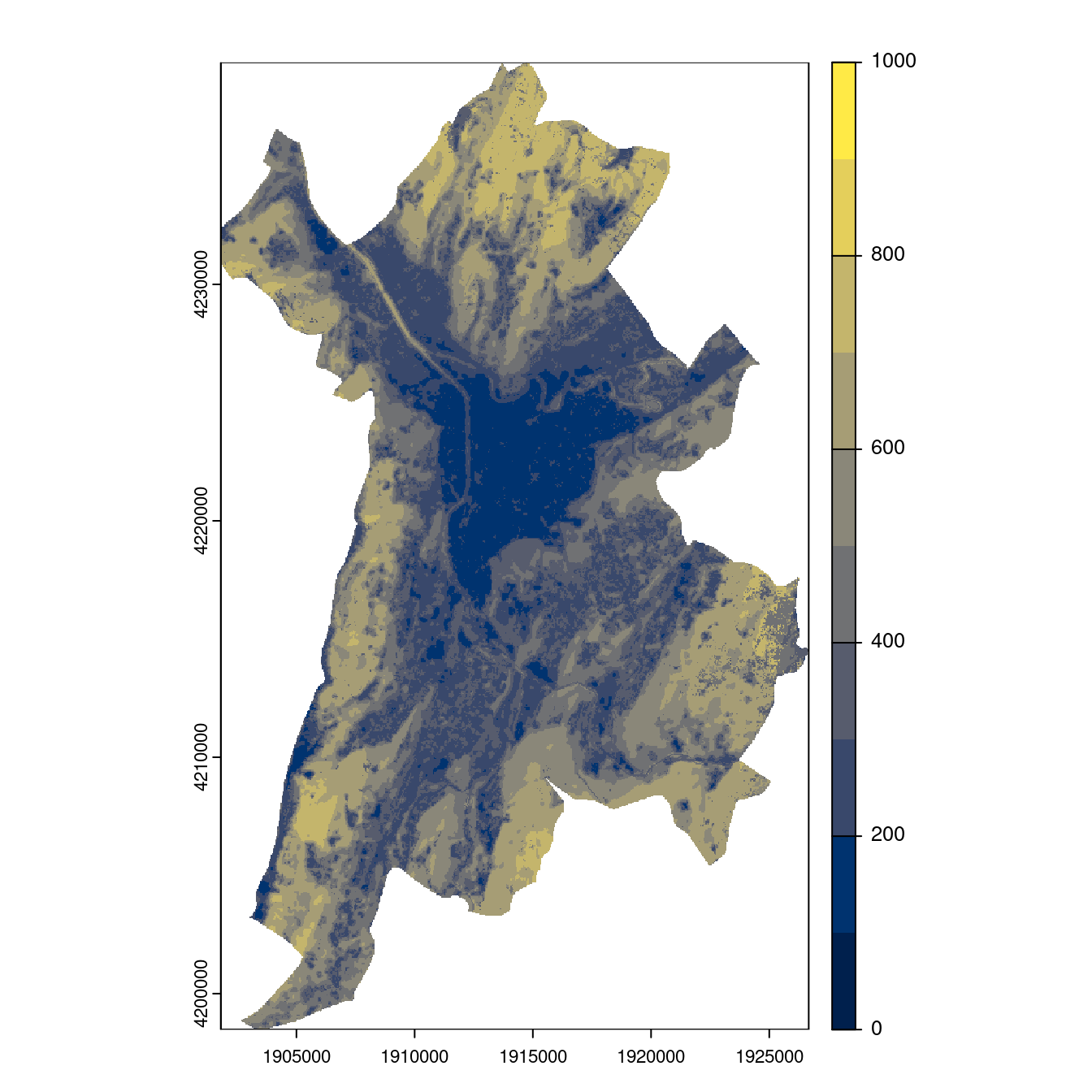
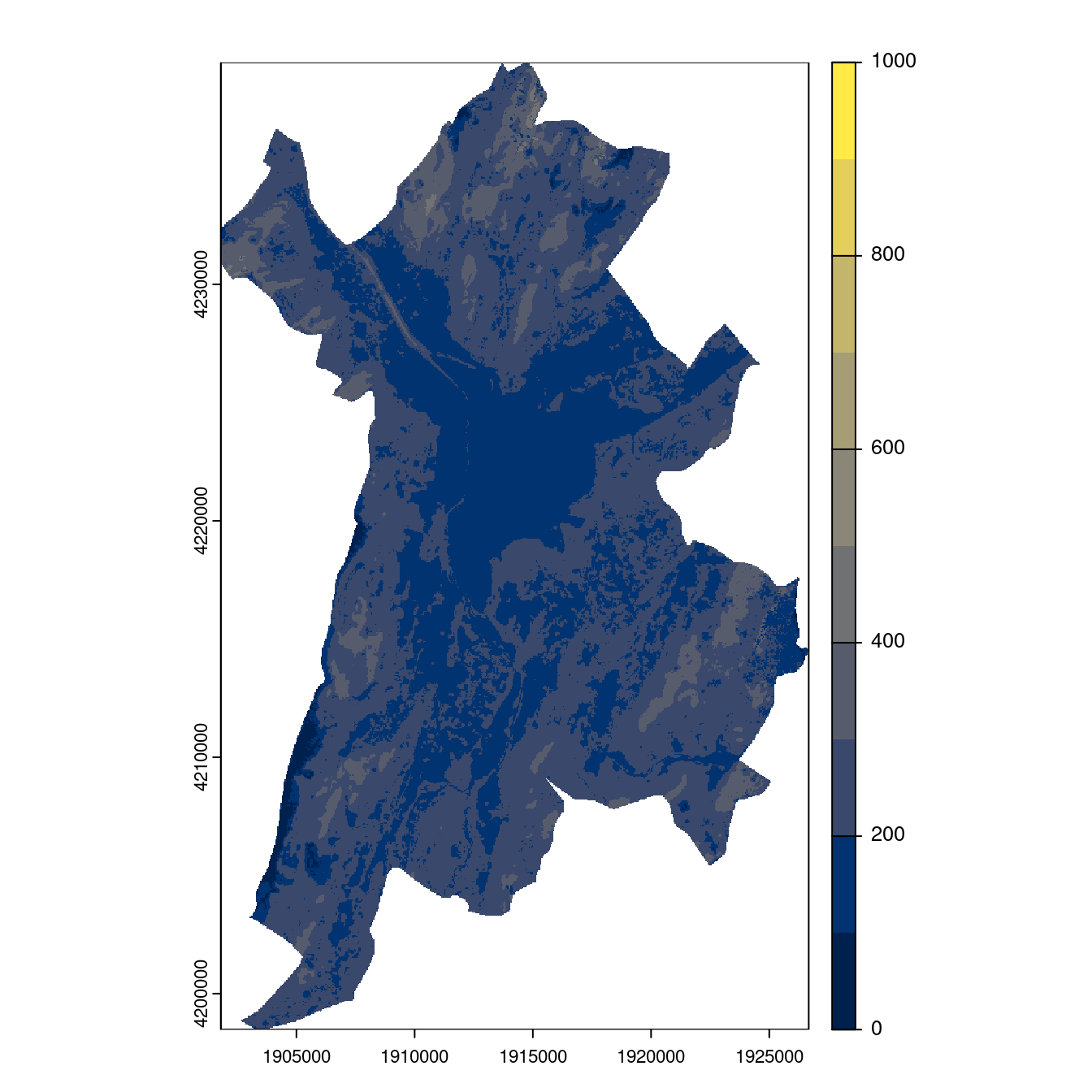
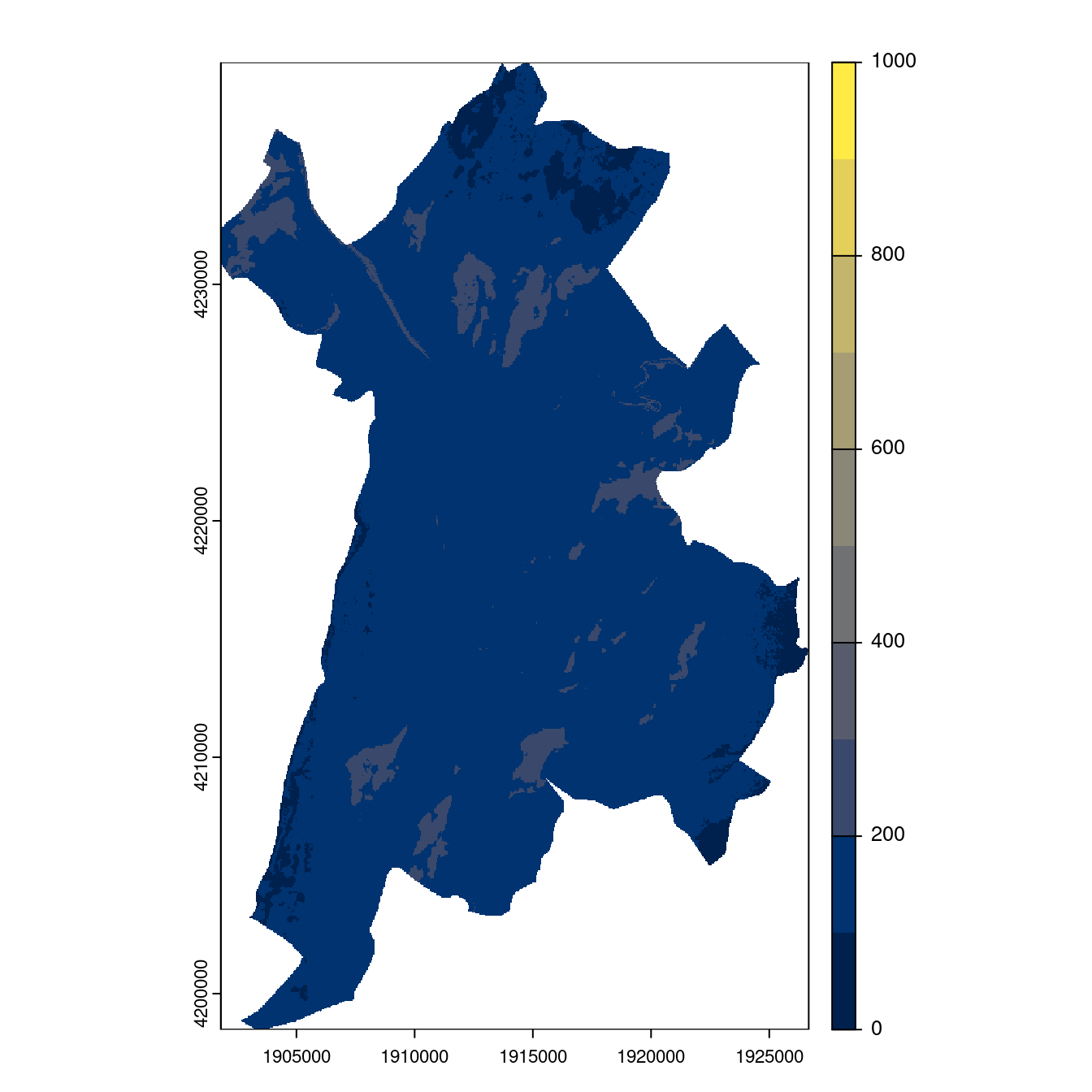
Figure 2.30: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
2.6.2 Évolution temporelle
# A tibble: 17 × 8
min q1 median mean q3 max ssp year
<table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <fct> <dbl>
1 192 477 791 679.8090796 826 856 0 2000
2 143 351 624 561.8186670 769 838 126 2040
3 136 295 528 502.8495678 712 815 126 2060
4 142 295 525 503.9722326 714 814 126 2080
5 145 299 543 512.3952685 719 805 126 2100
6 132 347 607 556.4350053 769 845 245 2040
7 131 274 463 456.7233839 646 798 245 2060
8 142 270 408 424.4407723 565 794 245 2080
9 127 255 355 363.3298285 448 734 245 2100
10 109 340 586 541.5026981 747 834 370 2040
11 143 280 466 464.7068790 658 799 370 2060
12 129 243 335 336.7282421 413 729 370 2080
13 77 136 201 190.0288996 233 430 370 2100
14 134 325 589 536.7383526 746 826 585 2040
15 132 270 414 426.3796311 575 786 585 2060
16 87 153 234 218.2031298 269 491 585 2080
17 63 112 127 138.5721538 164 249 585 2100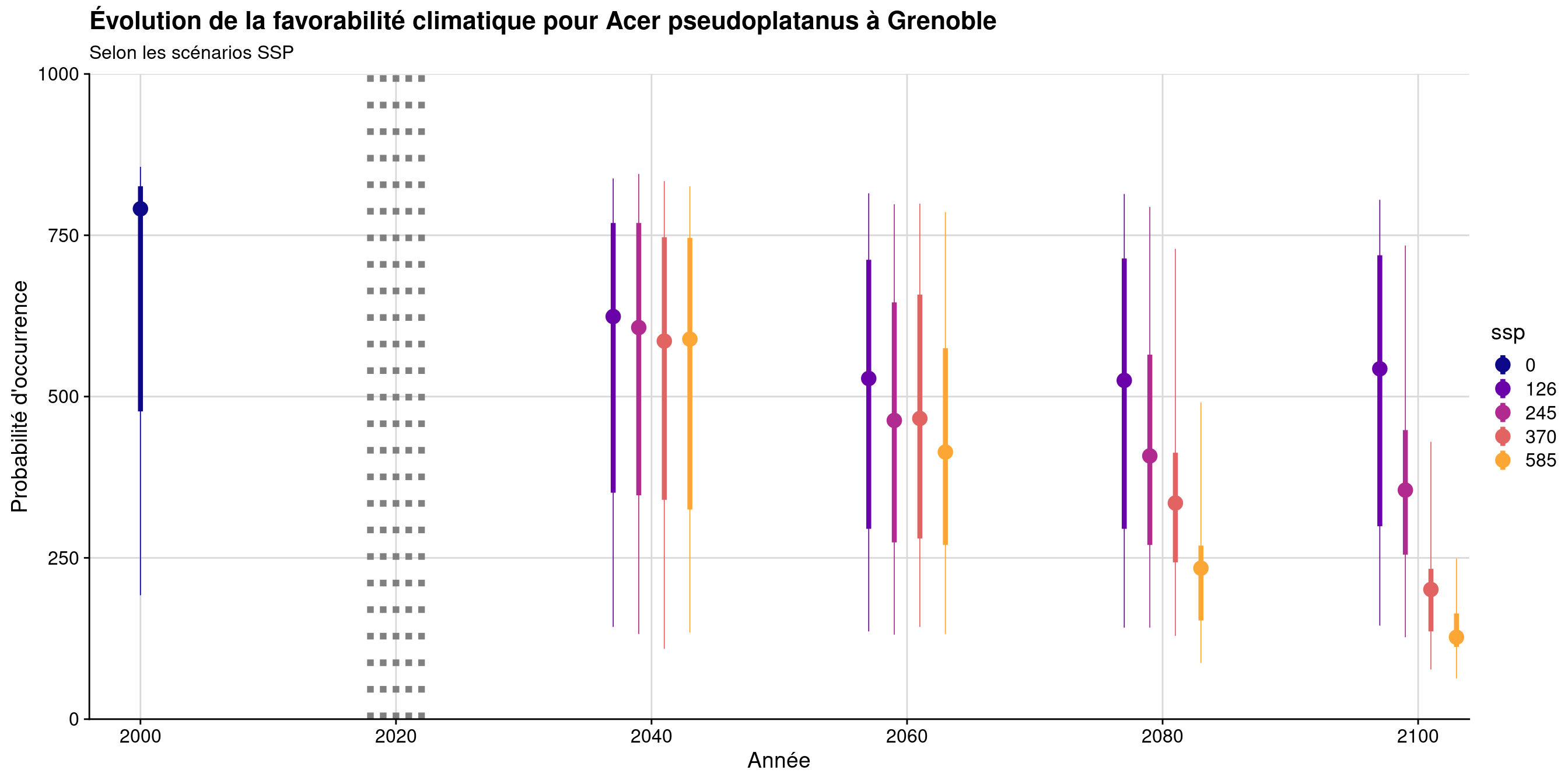
Figure 2.31: Évolution de la favorabilité climatique au cours du XXIème siècle. Le point représente la médiane sur l’étendue de la métropole (i.e. tous les pixels de la carte) ; le trait gras représente 50 % des données intermédiaires (espace inter-quartile) ; le trait fin représente le reste des données du minimum au maximum.