4 Mélèze d’Europe (Larix decidua)
On recommence la même chose pour le mélèze d’Europe, espèce à faible répartition localisée.
4.1 Taxonomie
On vérifie le statut taxonomique du mélèze d’Europe sur l’ossature de taxonomie du GBIF :
[1] "Larix decidua"# A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Larix decidua Mill. ACCEPTED FALSE Larix decidua 2686212 99 EXACT Pour le mélèze d’Europe, rien à signaler, c’est directement le nom scientifique accepté.
4.1.1 Données d’occurrences GBIF
Collecte des données du GBIF :
<<gbif download metadata>>
Status: SUCCEEDED
DOI: 10.15468/dl.gsn266
Format: SIMPLE_CSV
Download key: 0145782-230224095556074
Created: 2023-04-05T17:35:41.221+00:00
Modified: 2023-04-05T17:37:18.306+00:00
Download link: https://api.gbif.org/v1/occurrence/download/request/0145782-230224095556074.zip
Total records: 71317On peut dès lors explorer rapidement les données (classe du jeu de données et premières lignes du tableau) :
# A tibble: 71,317 × 50
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
2 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
3 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
4 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
5 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
6 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
7 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
8 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
9 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
10 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
# ℹ 71,307 more rows
# ℹ 40 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …Puis les transformer en objets géographiques (format Simple feature de sf) :
Simple feature collection with 71317 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -122.296237 ymin: -45.878241 xmax: 176.090026 ymax: 70.113781
Geodetic CRS: WGS 84
# A tibble: 71,317 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
2 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
3 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
4 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
5 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
6 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
7 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
8 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
9 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
10 4.93e-315 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
# ℹ 71,307 more rows
# ℹ 41 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
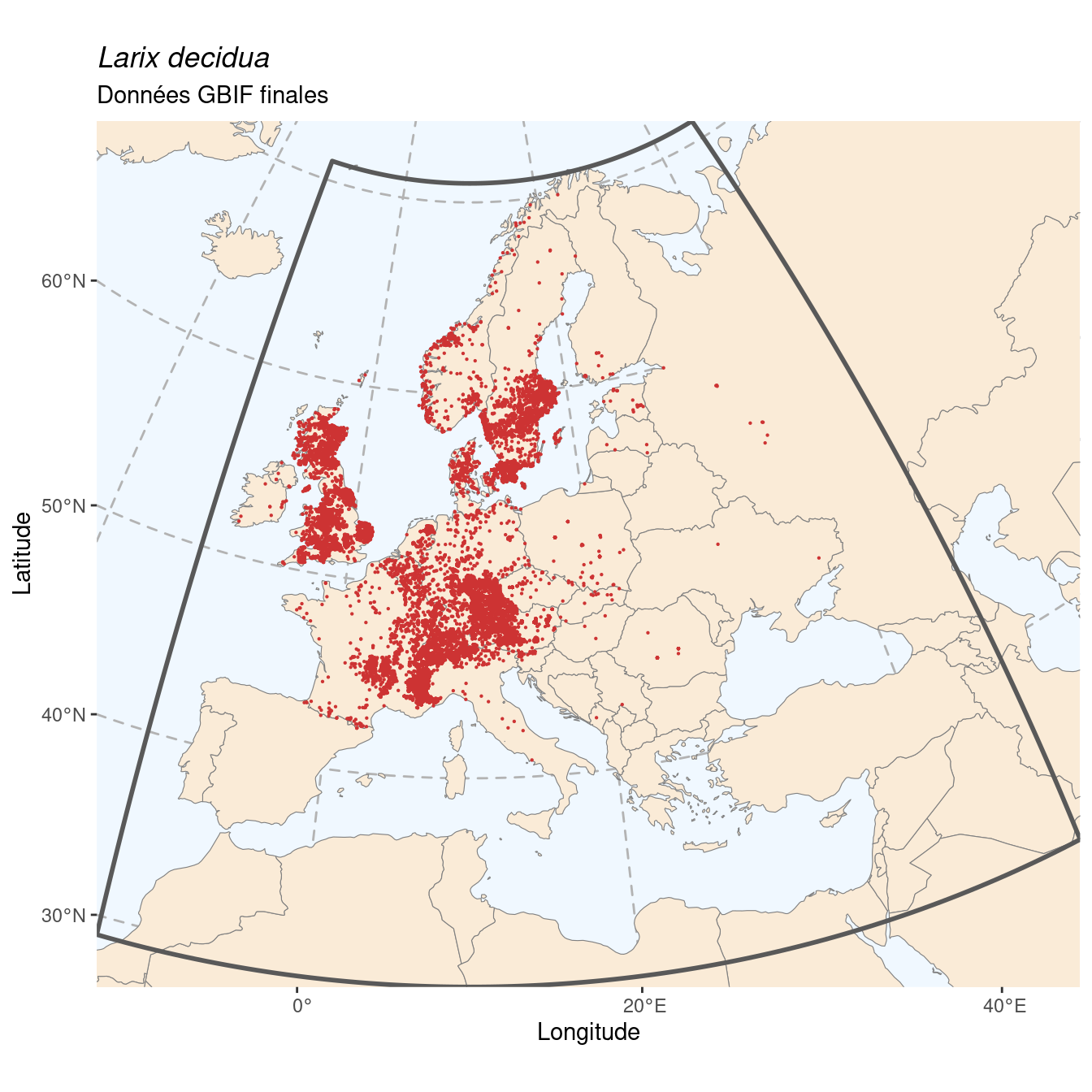
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …Il y a 71 317 occurrences dans le jeu de données. On les affiche sur la carte du monde :
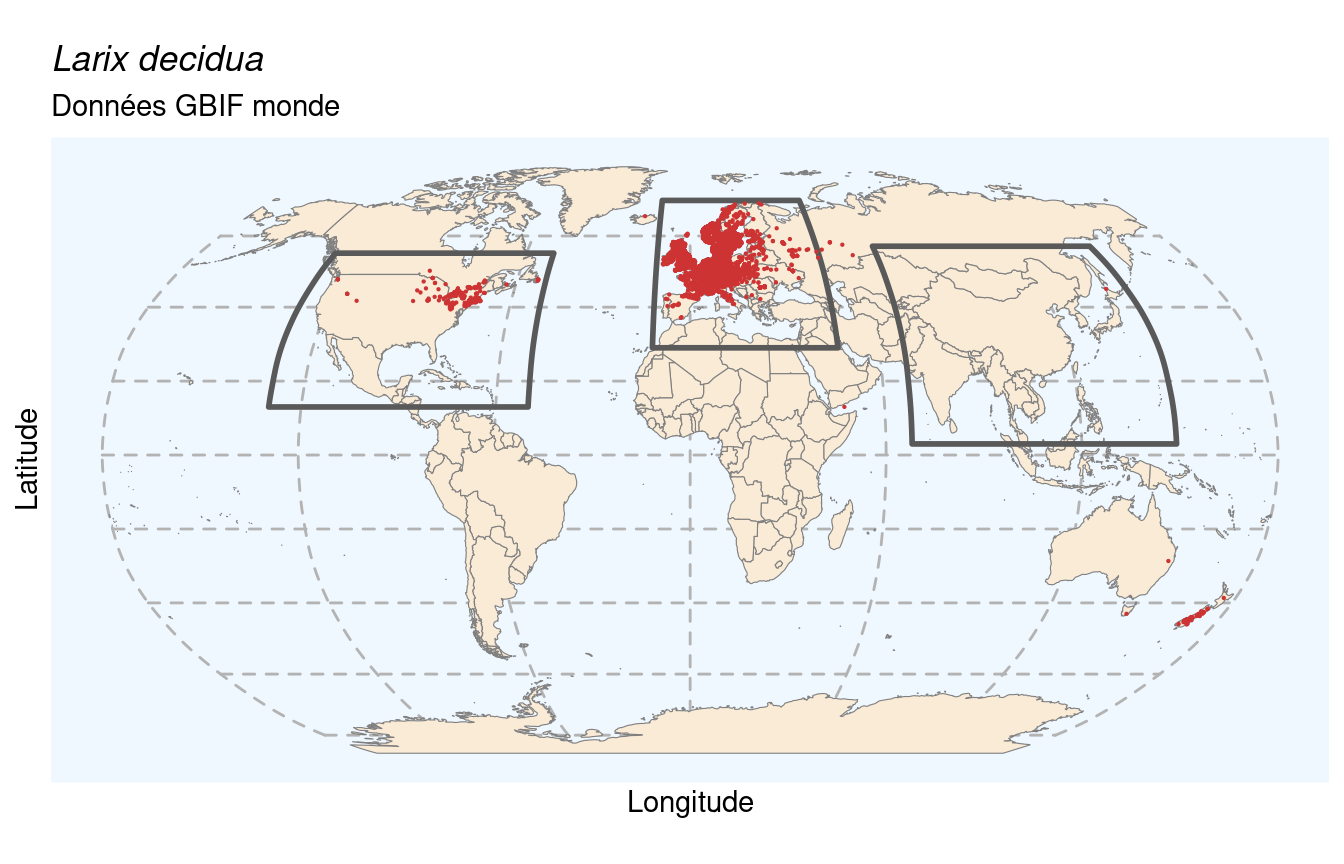
Figure 4.1: Occurrences de Larix decidua dans le monde.
4.2 Données par région
On évalue la proportion d’occurrences dans chaque région, en utilisant les 3 masques créés précédemment. Si on a au moins 50 % des occurrences dans une région, on peut continuer en travaillant uniquement sur ces données. Sinon, on vérifie qu’au moins 67 % des données tombent dans l’ensemble des régions, et on prend la région la plus représentée. Au final, s’il s’agit d’une espèce endémique d’Europe, on n’a pas besoin de manipulation supplémentaire.
[1] 0.9960037579[1] 0.002888511856[1] 1.402190221e-05On obtient 99.6 % d’occurrences en Europe, on est bien sur une espèce endémique de la région. On peut récupérer les données d’Europe, et les cartographier :
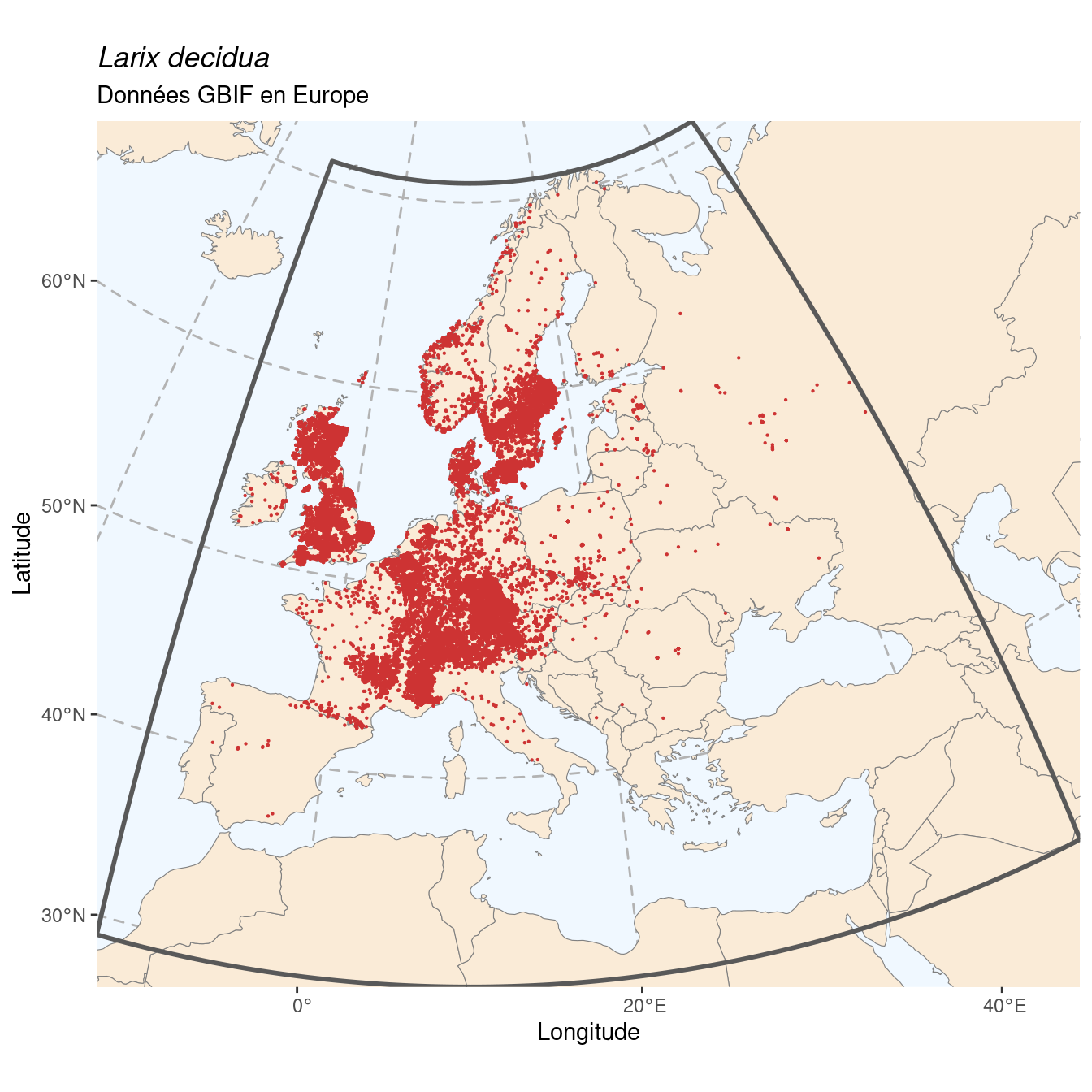
Figure 4.2: Occurrence de Larix decidua en Europe.
4.2.1 Licences
On s’intéresse ici au problème particulier des licences :
CC_BY_4_0
51567
CC_BY_NC_4_0
3328
CC0_1_0
6447
https://creativecommons.org/licenses/by-nc/4.0/legalcode
8521
https://creativecommons.org/licenses/by/4.0/legalcode
976
https://creativecommons.org/publicdomain/zero/1.0/legalcode
193 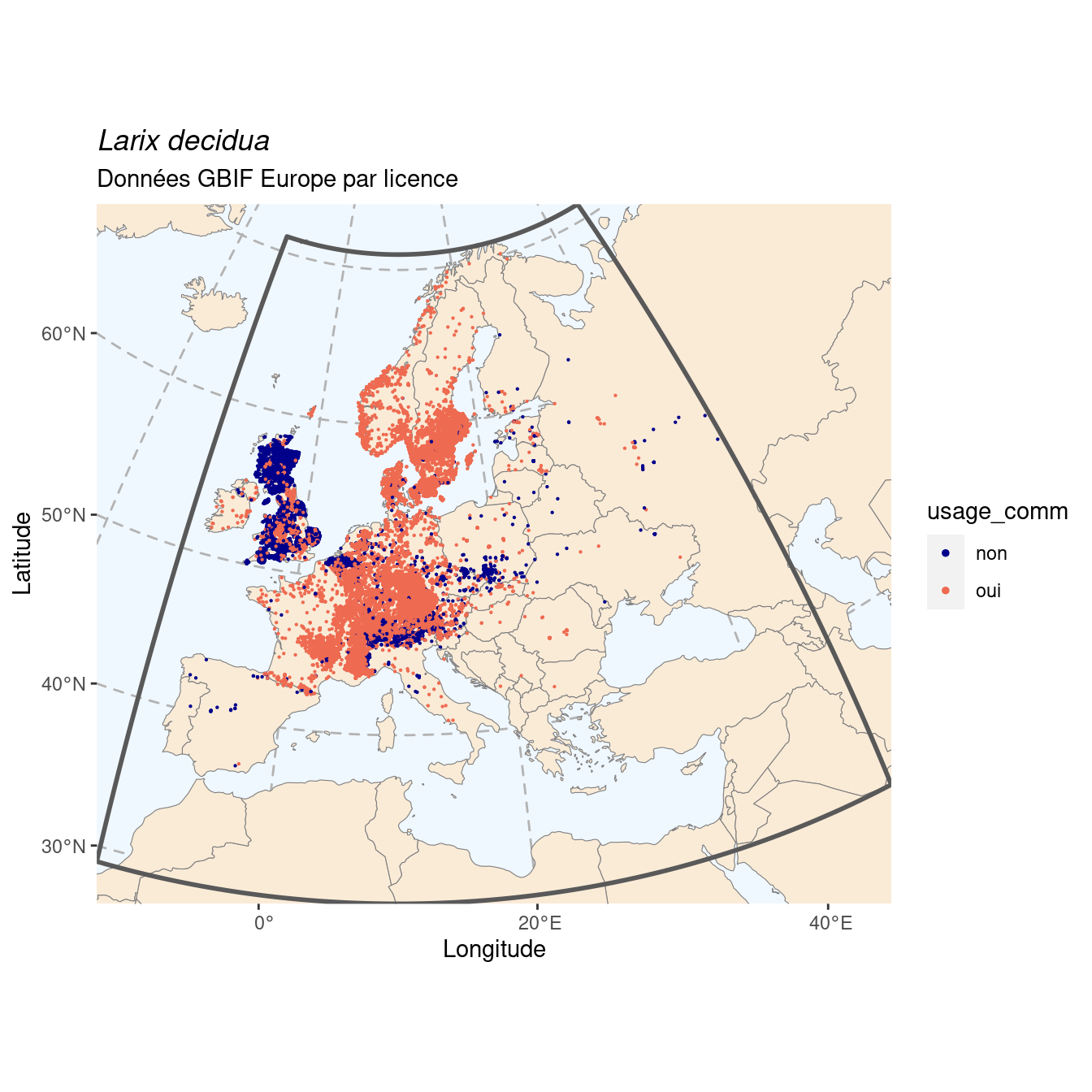
Figure 4.3: Occurrence de Larix decidua en Europe, selon la licence : CC BY-NC en bleu ; CC0 et CC BY en orange.
Les d’occurrences en CC BY forment l’essentiel du jeu de données. On peut donc supprimer les données en CC BY-NC sans risque.
4.2.2 Sous-échantillonnage
On a largement plus de 25 000 localisations ; on procède donc au sous-échantillonnage aléatoire :
Simple feature collection with 25000 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -9.58695 ymin: 40.867222 xmax: 37.788894 ymax: 69.959
Geodetic CRS: WGS 84
# A tibble: 25,000 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species
<int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 8.93e-315 64dabd3c-4f34-4520-b9dd-d227a… http://id.s… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
2 9.49e-315 8ea4250e-0ff0-44f8-812e-bffc3… http://id.s… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
3 2.00e-314 017f23ba-bf04-4eb7-a076-04ab2… 79bee565-fa… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
4 1.49e-314 c1492854-5d15-40e9-9ee7-24a18… 3632B7D1-2A… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
5 9.49e-315 8ea4250e-0ff0-44f8-812e-bffc3… http://id.s… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
6 1.46e-314 53f13c8f-413e-4537-bda6-98666… 2cd4p9h.bep… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
7 1.53e-314 36555a0c-8361-42c4-9f78-80581… 2cd4p9h.wy6… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
8 1.28e-314 4ebe5835-851f-43fd-beed-afade… d53e1ac4-7e… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
9 1.42e-314 38b4c89f-584c-41bb-bd8f-cd1de… urn:lsid:ar… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
10 1.19e-314 b124e1e0-4755-430f-9eab-894f2… urn:uuid:1e… Plantae Trach… Pino… Pina… Pinac… Larix Larix …
# ℹ 24,990 more rows
# ℹ 41 more variables: infraspecificEpithet <chr>, taxonRank <chr>, scientificName <chr>,
# verbatimScientificName <chr>, verbatimScientificNameAuthorship <chr>, countryCode <chr>,
# locality <chr>, stateProvince <chr>, occurrenceStatus <chr>, individualCount <int>,
# publishingOrgKey <chr>, decimalLatitude <dbl>, decimalLongitude <dbl>,
# coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>, elevation <dbl>,
# elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>, day <int>, …4.3 Modélisation de la niche climatique
4.3.1 Préparer les données
Organisation des données d’entrées (occurrences et données environnementales) et des données produites de pseudo-absences :
class : SpatVector
geometry : points
dimensions : 25000, 0 (geometries, attributes)
extent : -9.58695, 37.788894, 40.867222, 69.959 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= lade Data Formating -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
! Response variable is considered as only presences... Is it really what you want?
! No data has been set aside for modeling evaluation
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
Checking Pseudo-absence selection arguments...
> random pseudo absences selection
> Pseudo absences are selected in explanatory variables
! Some NAs have been automatically removed from your data
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.formated.data -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
dir.name = Output
sp.name = lade
24987 presences, 0 true absences and 72872 undefined points in dataset
6 explanatory variables
temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart
Min. : 0.50000 Min. :-22.000000 Min. :-11.803333 Min. : 0.0000 Min. : 5.0000
1st Qu.:18.89200 1st Qu.: -9.142000 1st Qu.: 7.116667 1st Qu.: 622.6122 1st Qu.: 181.0000
Median :22.43600 Median : -4.640000 Median : 12.368668 Median : 725.7363 Median : 222.0000
Mean :23.96931 Mean : -4.879591 Mean : 11.242705 Mean : 752.6974 Mean : 239.4476
3rd Qu.:27.82000 3rd Qu.: 0.412000 3rd Qu.: 15.975000 3rd Qu.: 895.0248 3rd Qu.: 294.0000
Max. :45.60000 Max. : 12.148000 Max. : 26.406666 Max. :1394.1293 Max. :1248.0000
prec_season
Min. : 4.719104
1st Qu.: 22.455683
Median : 30.734993
Mean : 35.298865
3rd Qu.: 39.210051
Max. :123.602058
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=On visualise les pseudo-absences :
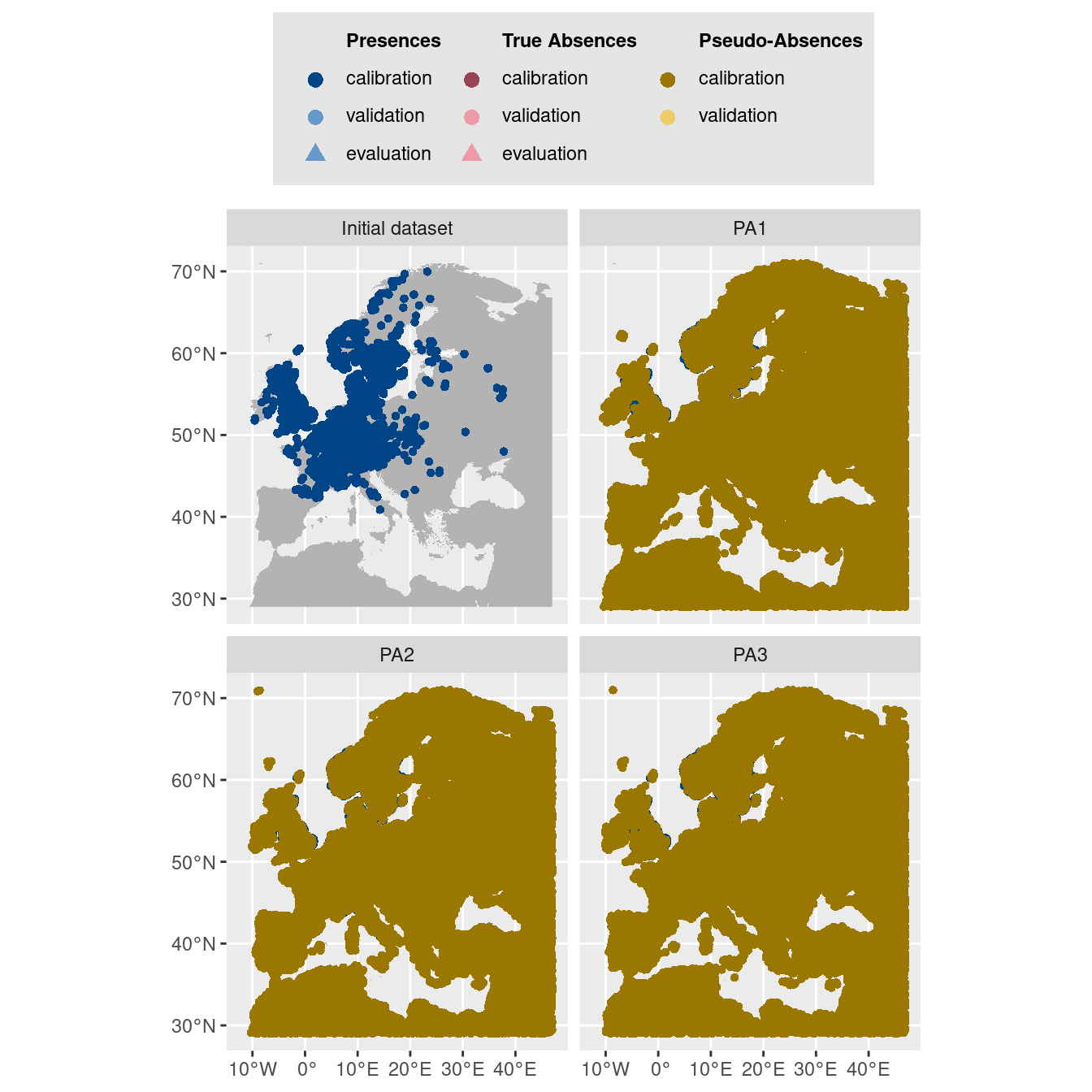
Figure 4.5: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
$data.vect
class : SpatVector
geometry : points
dimensions : 174948, 2 (geometries, attributes)
extent : -10.52083333, 46.97916667, 29.02083333, 70.97916667 (xmin, xmax, ymin, ymax)
coord. ref. :
names : resp dataset
type : <num> <chr>
values : 10 Initial dataset
10 Initial dataset
10 Initial dataset
$data.label
9 10 11
"**Presences**" "Presences (calibration)" "Presences (validation)"
12 19 20
"Presences (evaluation)" "**True Absences**" "True Absences (calibration)"
21 22 29
"True Absences (validation)" "True Absences (evaluation)" "**Pseudo-Absences**"
30 31 1
"Pseudo-Absences (calibration)" "Pseudo-Absences (validation)" "Background"
$data.plot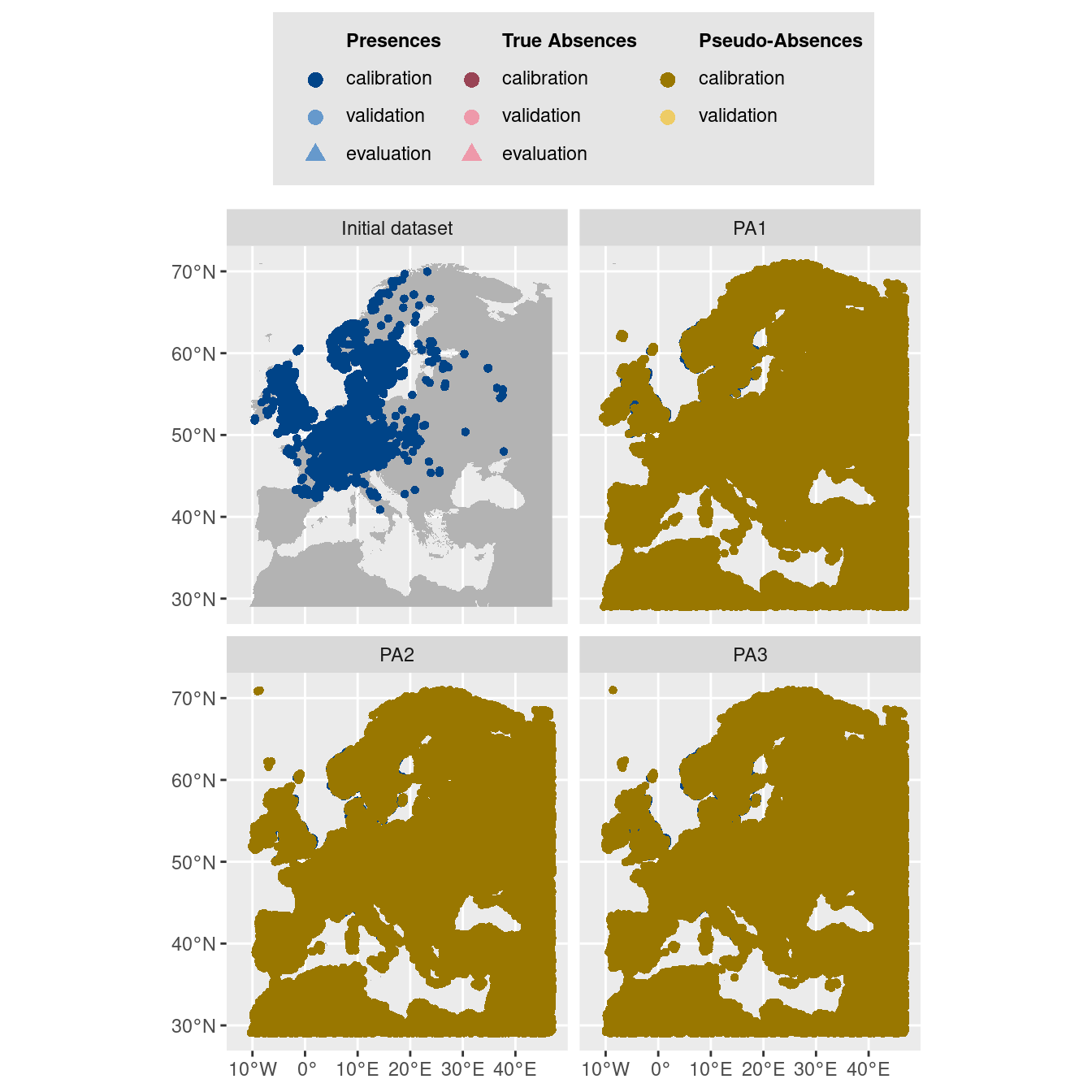
Figure 4.6: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
4.3.2 Modèles individuels de niche
prendre en compte jeu de données validation
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Build Single Models -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Checking Models arguments...
Creating suitable Workdir...
! Weights where automatically defined for lade_PA1 to rise a 0.5 prevalence !
! Weights where automatically defined for lade_PA2 to rise a 0.5 prevalence !
! Weights where automatically defined for lade_PA3 to rise a 0.5 prevalence !
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= lade Modeling Summary -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
6 environmental variables ( temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season )
Number of evaluation repetitions : 1
Models selected : GAM MARS MAXNET GBM ANN RF
Total number of model runs: 18
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=- Run : lade_PA1
-=-=-=--=-=-=- lade_PA1_RUN1
-=-=-=- Run : lade_PA2
-=-=-=--=-=-=- lade_PA2_RUN1
-=-=-=- Run : lade_PA3
-=-=-=--=-=-=- lade_PA3_RUN1
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=4.3.2.1 Évaluation de la performance
full.name PA run algo metric.eval cutoff sensitivity specificity calibration
1 lade_PA1_RUN1_GAM PA1 RUN1 GAM TSS 566.0 95.628 60.200 0.558
2 lade_PA1_RUN1_GAM PA1 RUN1 GAM ROC 566.5 95.613 60.235 0.724
3 lade_PA1_RUN1_MARS PA1 RUN1 MARS TSS 401.0 97.679 81.445 0.791
4 lade_PA1_RUN1_MARS PA1 RUN1 MARS ROC 409.5 97.519 81.630 0.946
5 lade_PA1_RUN1_MAXNET PA1 RUN1 MAXNET TSS 330.0 97.589 80.570 0.781
6 lade_PA1_RUN1_MAXNET PA1 RUN1 MAXNET ROC 329.5 97.589 80.570 0.944
validation evaluation
1 0.547 NA
2 0.720 NA
3 0.783 NA
4 0.942 NA
5 0.774 NA
6 0.938 NA
Figure 4.7: Évaluation de la performance des modèles individuels : True Skill Statistic & aire sous la courbe de ROC.
Peu de modèles ont une performance suffisante au sens de la TSS : GBF et RF, qui ont aussi les meilleures performances au sens du ROC. Les GAMs et ANNs ont les performances les plus faibles.
4.3.2.2 Importance des variables environnementales
L’importance de chaque variable est estimée par randomisation de la variable
d’intérêt (moyenne/variance sur tous les algorithmes). On utilise la fonction
get_variables_importance() pour récupérer l’importance des variables, que l’on
peut visualiser par algorithme ou par variable :
full.name PA run algo expl.var rand var.imp
1 lade_PA1_RUN1_GAM PA1 RUN1 GAM temp_max_august 1 0.515309
2 lade_PA1_RUN1_GAM PA1 RUN1 GAM temp_min 1 0.392117
3 lade_PA1_RUN1_GAM PA1 RUN1 GAM temp_wet_quart 1 0.007239
4 lade_PA1_RUN1_GAM PA1 RUN1 GAM temp_season 1 0.940940
5 lade_PA1_RUN1_GAM PA1 RUN1 GAM prec_wet_quart 1 0.020971
6 lade_PA1_RUN1_GAM PA1 RUN1 GAM prec_season 1 0.186381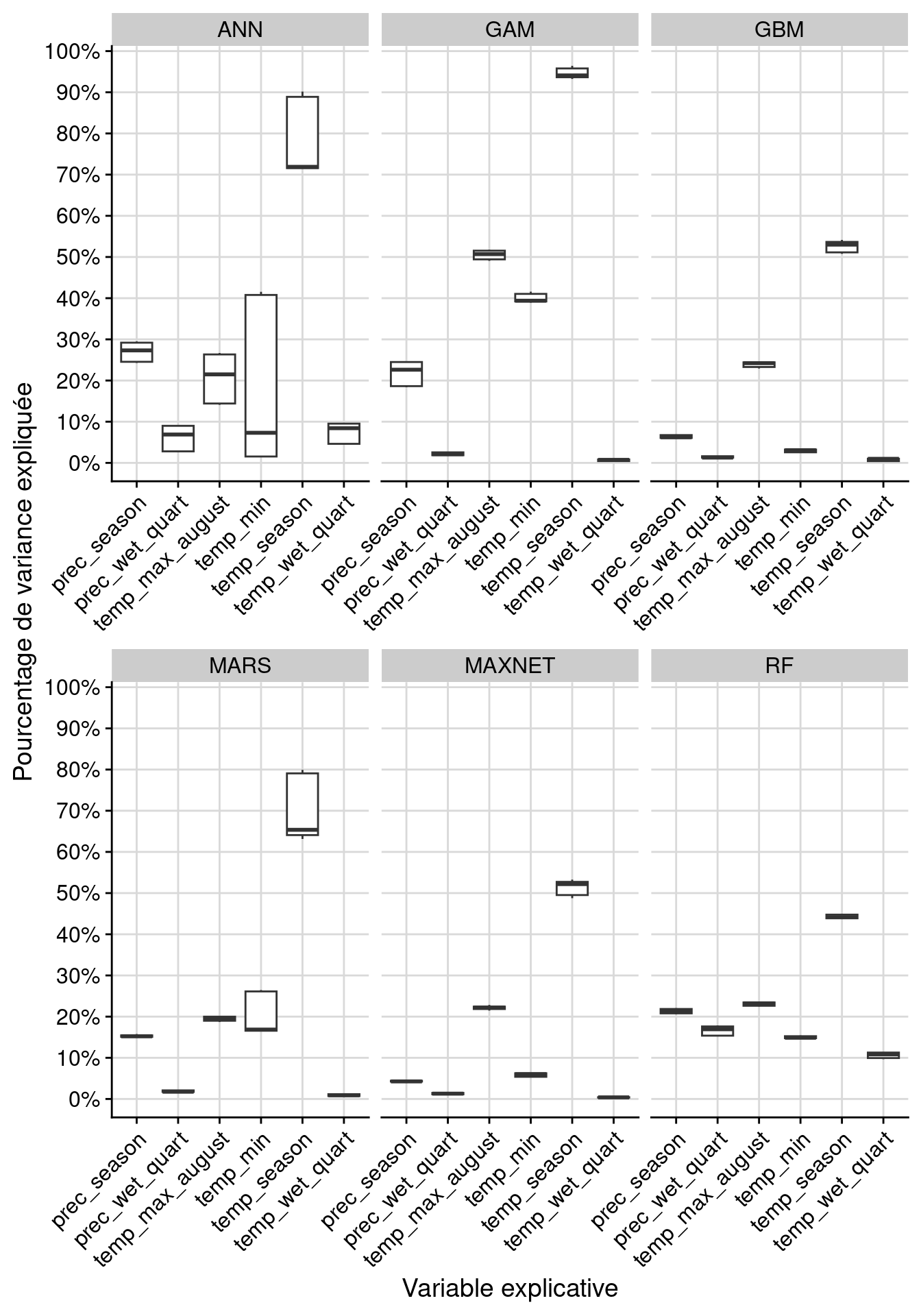
Figure 4.8: Pourcentage de variance expliquée pour chaque variable, décomposé par algorithme.

Figure 4.9: Pourcentage de variance expliquée pour chaque variable, décomposé par variable.
4.3.2.3 Courbes de réponse
Finalement, le modèle permet d’établir des courbes de réponse, c’est-à-dire la
variation de la probabilité d’occurrence selon chaque variable
environnementale. On utilise pour cela la fonction bm_PlotResponseCurves() qui
permet d’extraire ces valeurs et de les afficher :
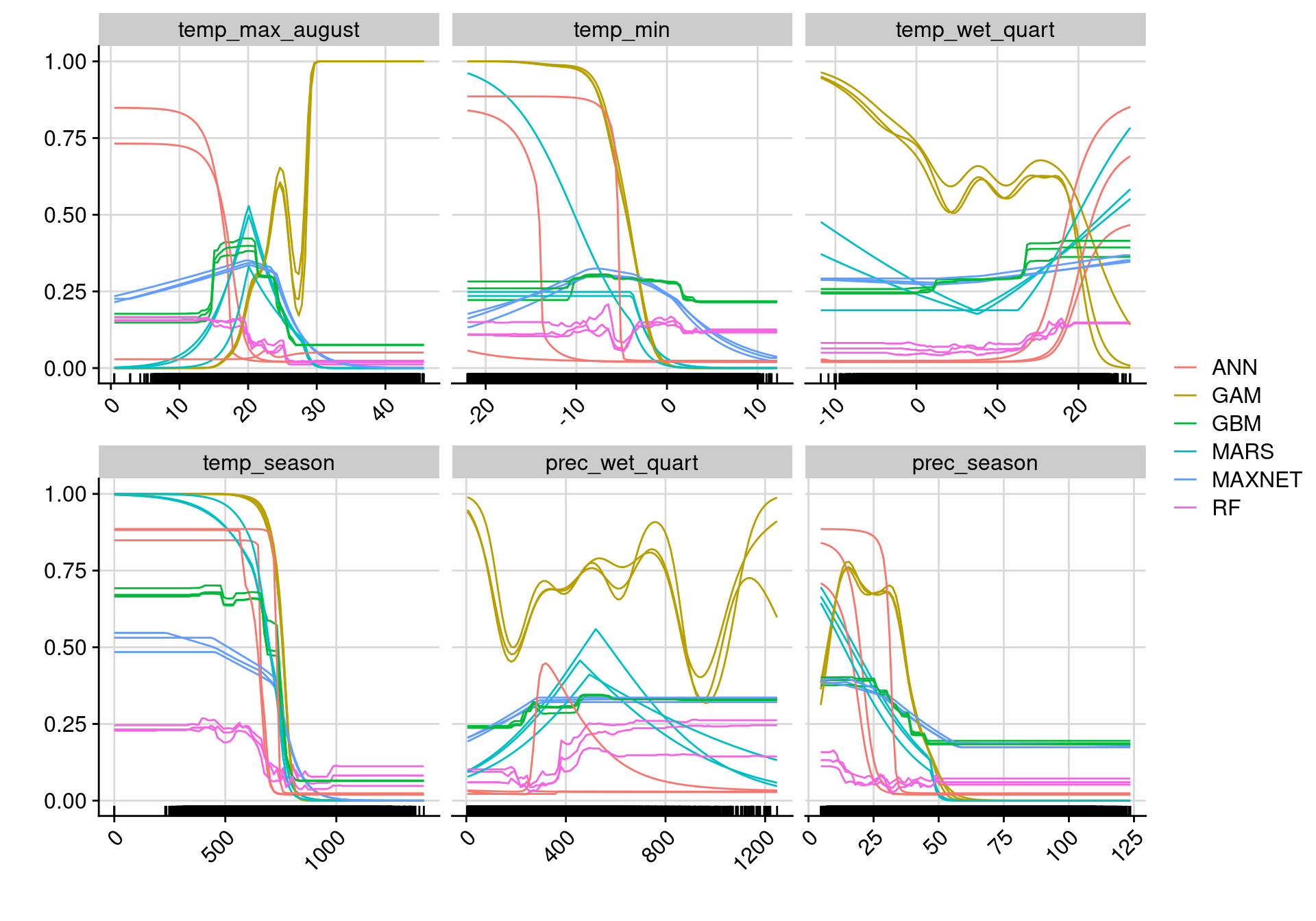
Figure 4.10: Courbes de réponse de chaque variable climatique selon l’algorithme (en couleurs).
4.3.3 Modèles d’ensemble de niche
Seulement modèles avec TSS > 0.8
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.ensemble.models.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
sp.name : lade
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
models computed:
lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo, lade_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
models failed: none
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=→ prend les modèles individuels en entrée → on décide sur quel critère les modèles sont gardés
même idée que l’interférence multi-modèles, mais basé sur métrique de performance (e.g. TSS > 0. et AUC/ROC > 0.9)
→ on peut calculer moyenne ou médiane → retourne des coefficients de variation → retourne des probabilités pondérées par la métrique choisie
→ grouping level stratifié jusqu’à “all”
4.3.3.1 Évaluation de la qualité
get_evaluations() pour récupérer les stats d’intérêt
full.name merged.by.PA merged.by.run merged.by.algo
1 lade_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
2 lade_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
filtered.by algo metric.eval cutoff sensitivity specificity calibration validation evaluation
1 TSS EMwmean TSS 649.0 96.53 94.482 0.910 NA NA
2 TSS EMwmean ROC 649.5 96.51 94.505 0.993 NA NA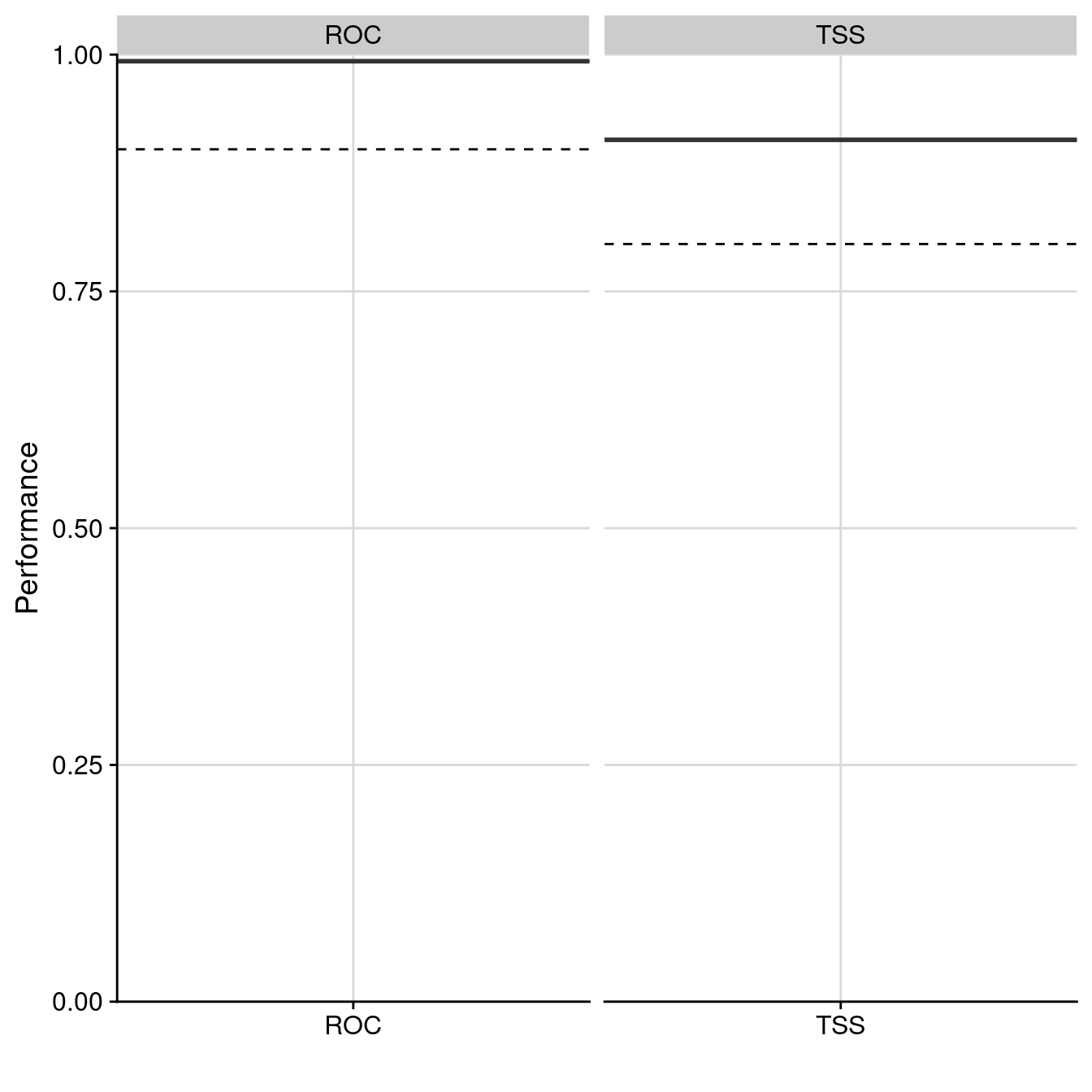
Figure 4.11: Évaluation de la qualité du modèle d’ensemble : Aire sous la courbe de ROC & True Skill Statistic.
full.name merged.by.PA merged.by.run merged.by.algo filtered.by
1 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
2 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
3 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
4 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
5 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
6 lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
algo expl.var rand var.imp
1 EMcv temp_max_august 1 0.492983
2 EMcv temp_min 1 0.393762
3 EMcv temp_wet_quart 1 0.177169
4 EMcv temp_season 1 0.451009
5 EMcv prec_wet_quart 1 0.249359
6 EMcv prec_season 1 0.341532Par variable :
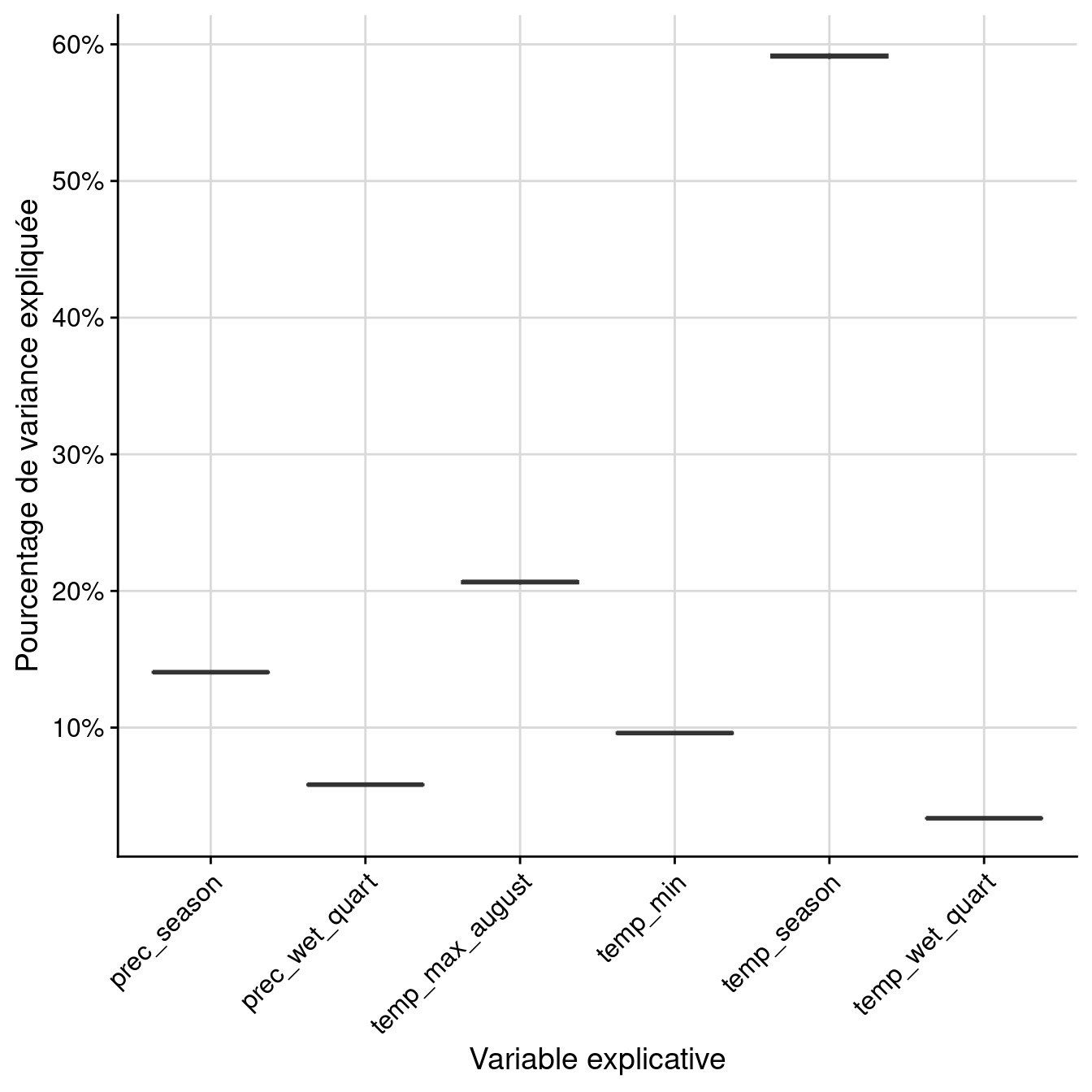
Figure 4.12: Pourcentage de variance expliquée par variable dans le modèle d’ensemble.
bm_PlotResponseCurves() pour forme de la réponse sur chaque variable
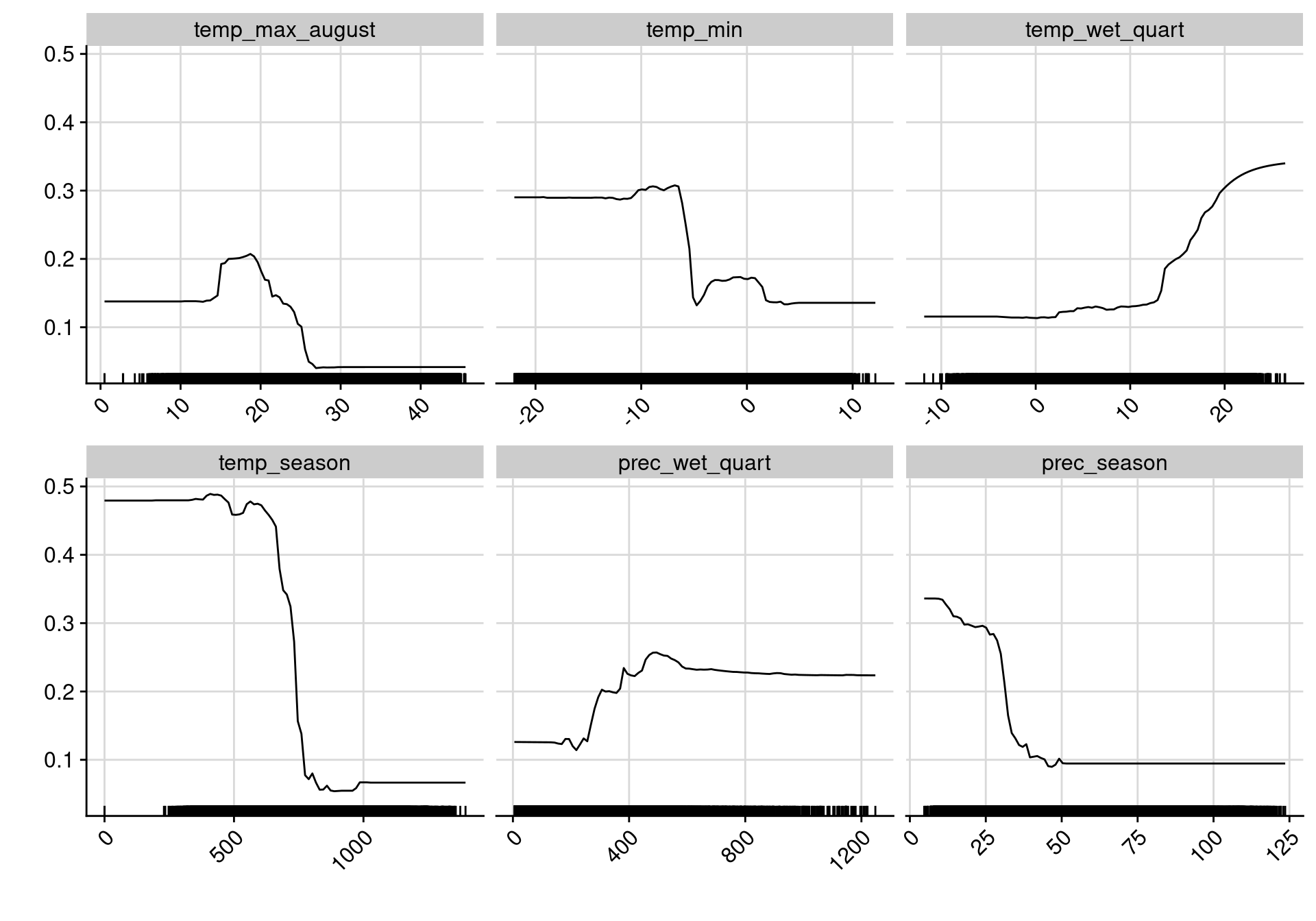
Figure 4.13: Courbes de réponse de chaque variable climatique dans le modèle d’ensemble.
4.4 Projections
4.4.1 Distribution potentielle contemporaine
Contemporaine : Cartographie des observations sur couches climatiques Extraction des conditions climatiques favorables
BIOMOD_Projection() un seul modèle
BIOMOD_EnsembleForecasting() modèle d’ensemble
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/lade/current
sp.name : lade
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/lade/lade.AllModels.models.out )
models.projected :
lade_PA1_RUN1_GAM, lade_PA1_RUN1_MARS, lade_PA1_RUN1_MAXNET, lade_PA1_RUN1_GBM, lade_PA1_RUN1_ANN, lade_PA1_RUN1_RF, lade_PA2_RUN1_GAM, lade_PA2_RUN1_MARS, lade_PA2_RUN1_MAXNET, lade_PA2_RUN1_GBM, lade_PA2_RUN1_ANN, lade_PA2_RUN1_RF, lade_PA3_RUN1_GAM, lade_PA3_RUN1_MARS, lade_PA3_RUN1_MAXNET, lade_PA3_RUN1_GBM, lade_PA3_RUN1_ANN, lade_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=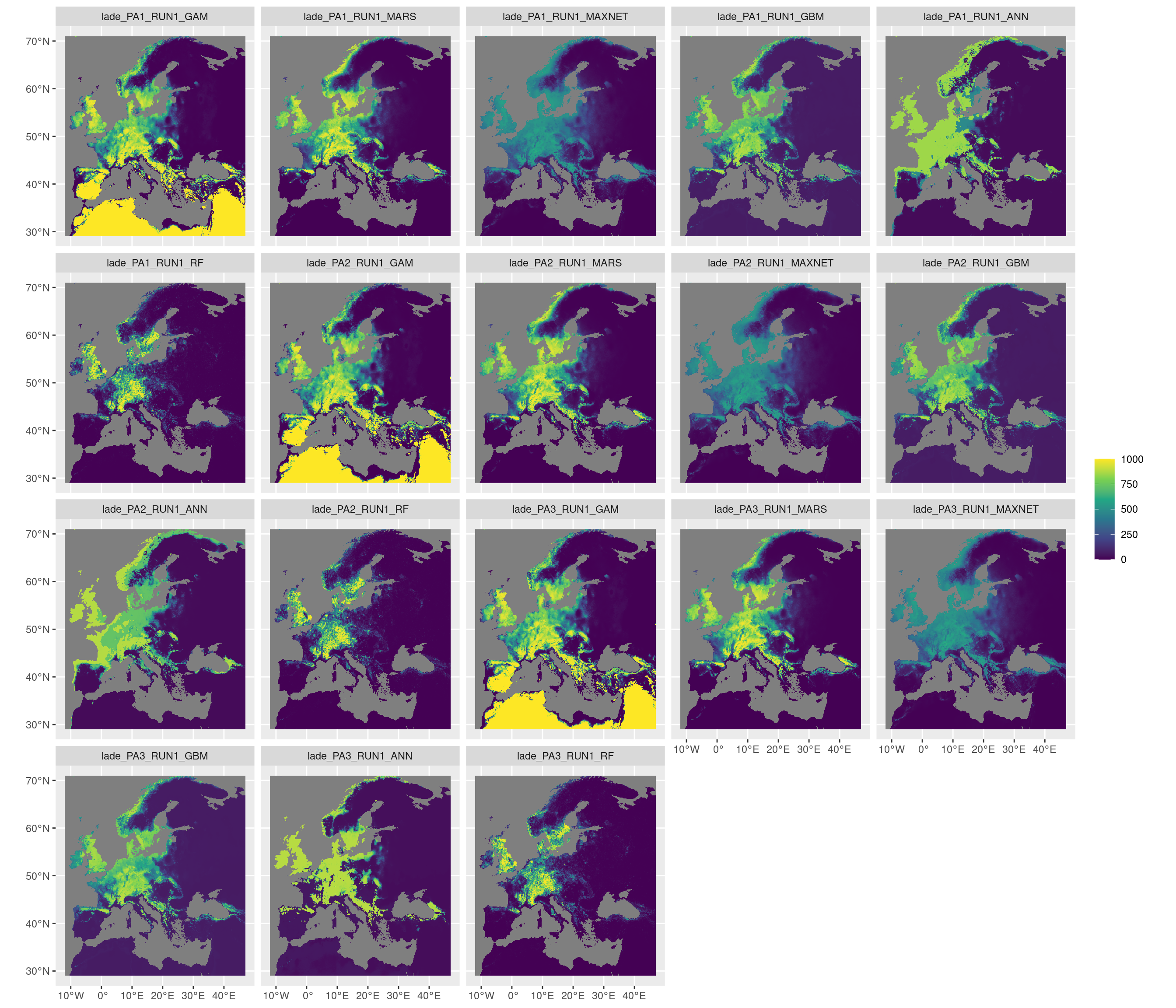
Figure 4.14: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
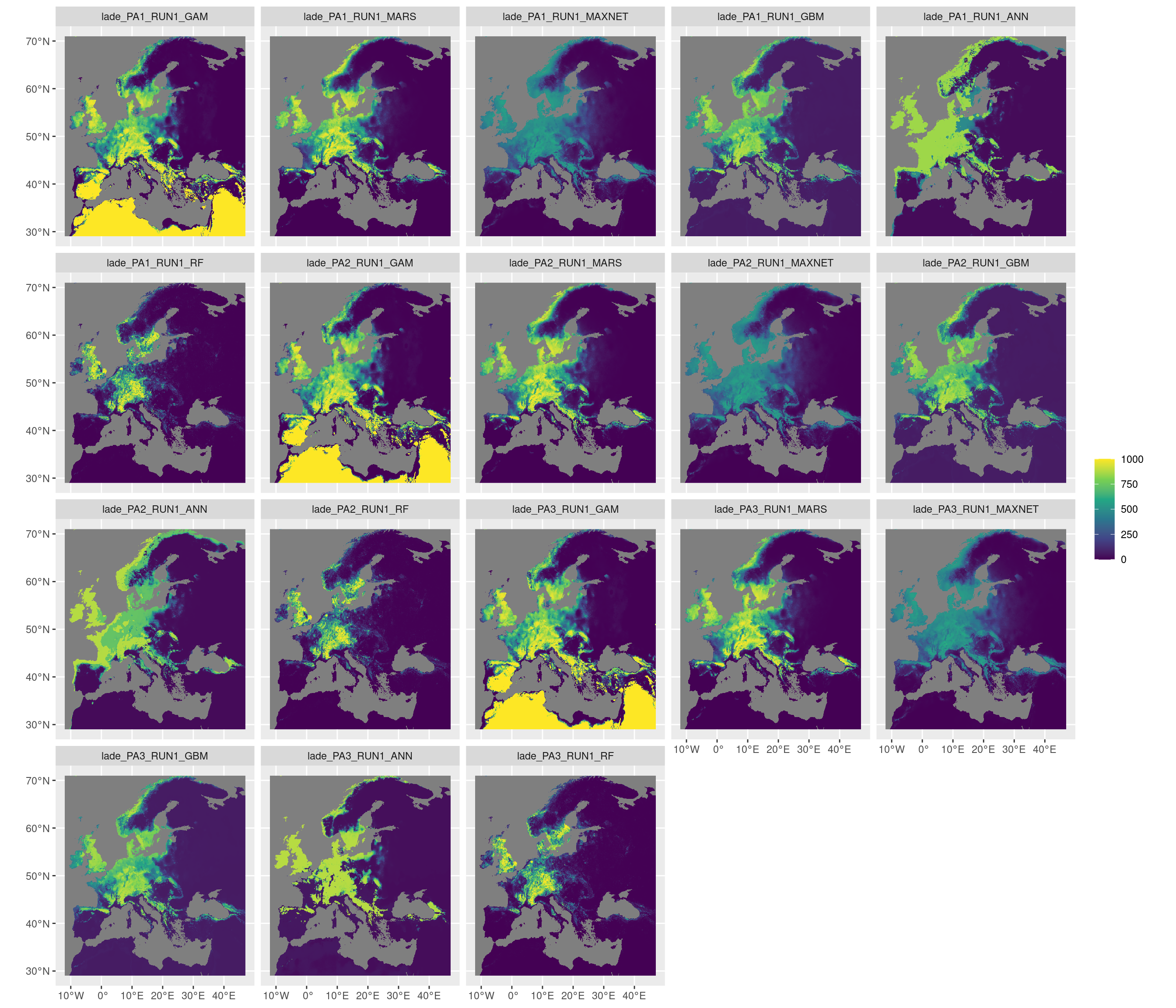
Figure 4.15: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/lade/current
sp.name : lade
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/lade/lade.AllModels.ensemble.models.out )
models.projected :
lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo, lade_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=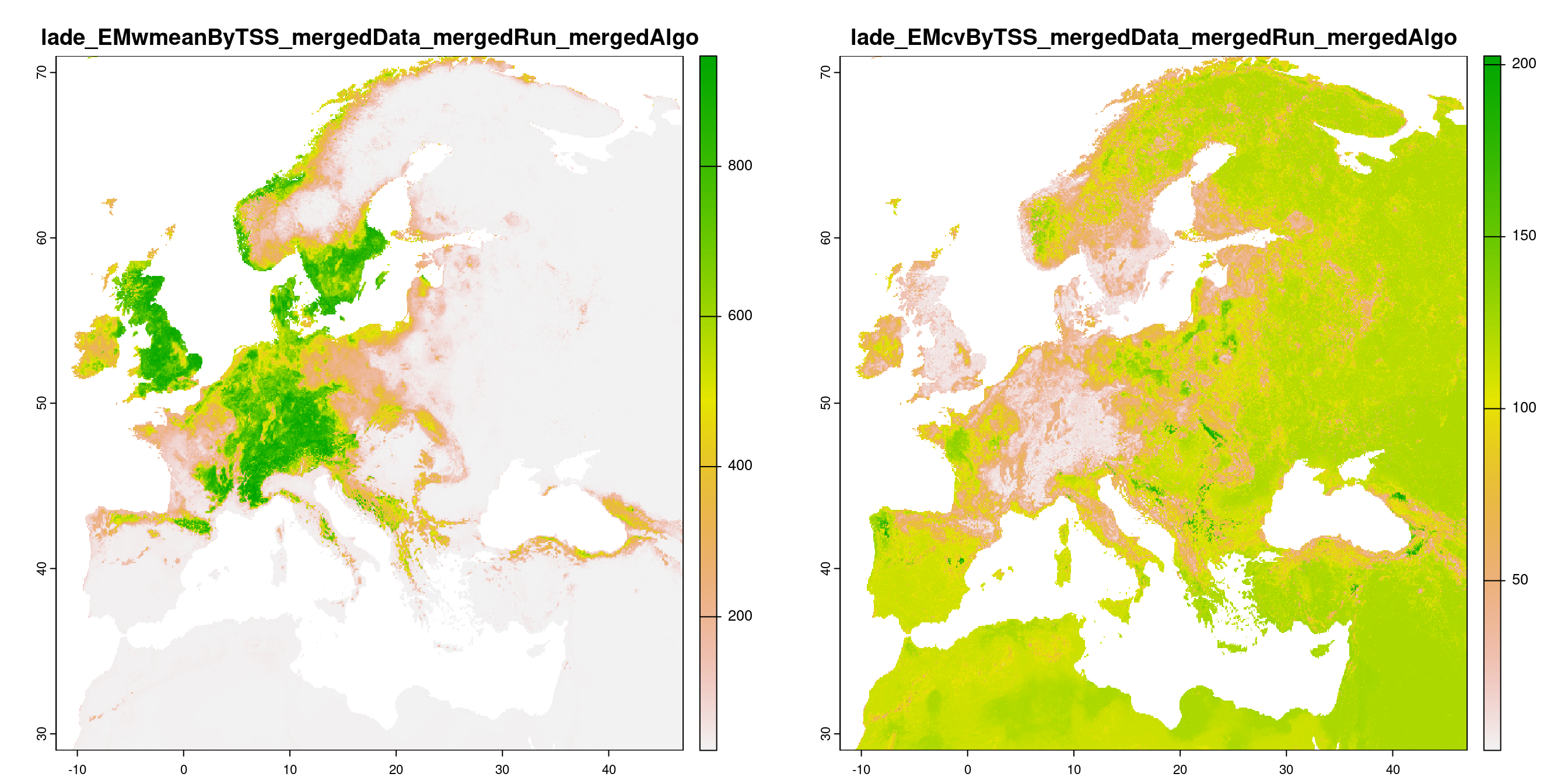
Figure 4.16: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) ; B) Incertitude associée à la projection.
4.4.2 Interpolation à l’échelle de la métropole
- → conditions climatiques favorables à fine échelle
On projette la niche climatique sur ce raster à fine échelle.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/lade/cont_gre
sp.name : lade
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/lade/lade.AllModels.models.out )
models.projected :
lade_PA1_RUN1_GAM, lade_PA1_RUN1_MARS, lade_PA1_RUN1_MAXNET, lade_PA1_RUN1_GBM, lade_PA1_RUN1_ANN, lade_PA1_RUN1_RF, lade_PA2_RUN1_GAM, lade_PA2_RUN1_MARS, lade_PA2_RUN1_MAXNET, lade_PA2_RUN1_GBM, lade_PA2_RUN1_ANN, lade_PA2_RUN1_RF, lade_PA3_RUN1_GAM, lade_PA3_RUN1_MARS, lade_PA3_RUN1_MAXNET, lade_PA3_RUN1_GBM, lade_PA3_RUN1_ANN, lade_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=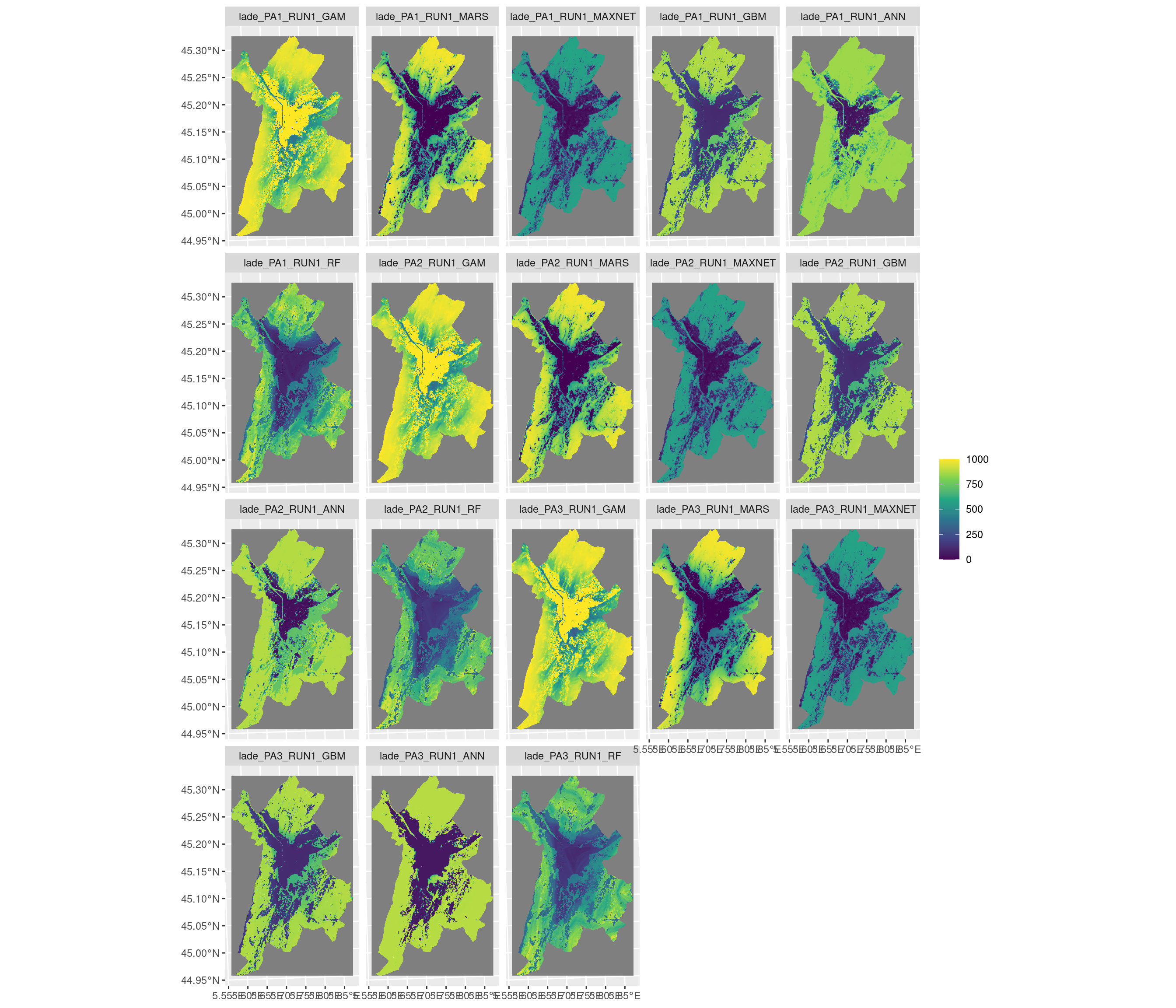
Figure 4.17: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
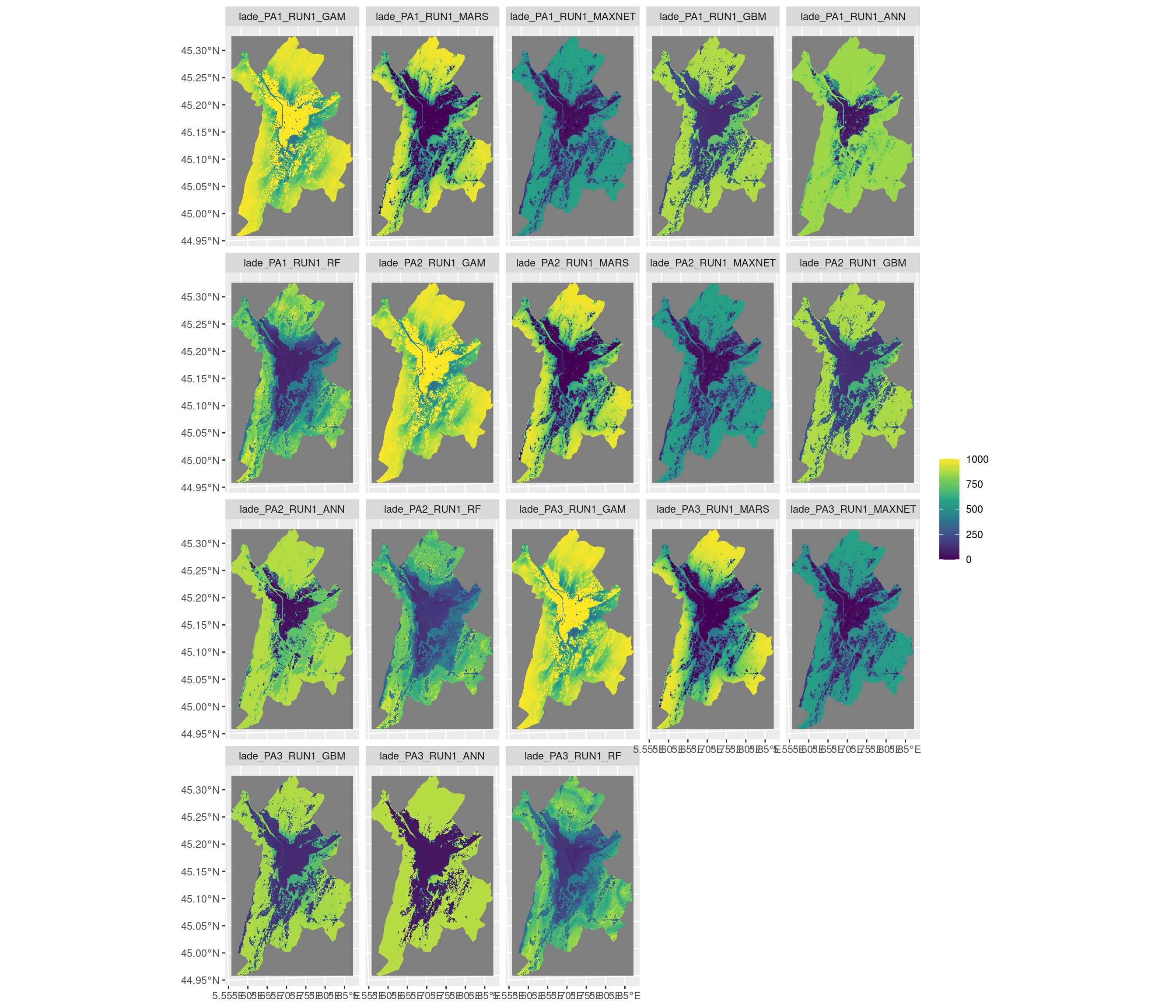
Figure 4.18: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/lade/cont_gre
sp.name : lade
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/lade/lade.AllModels.ensemble.models.out )
models.projected :
lade_EMcvByTSS_mergedData_mergedRun_mergedAlgo, lade_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=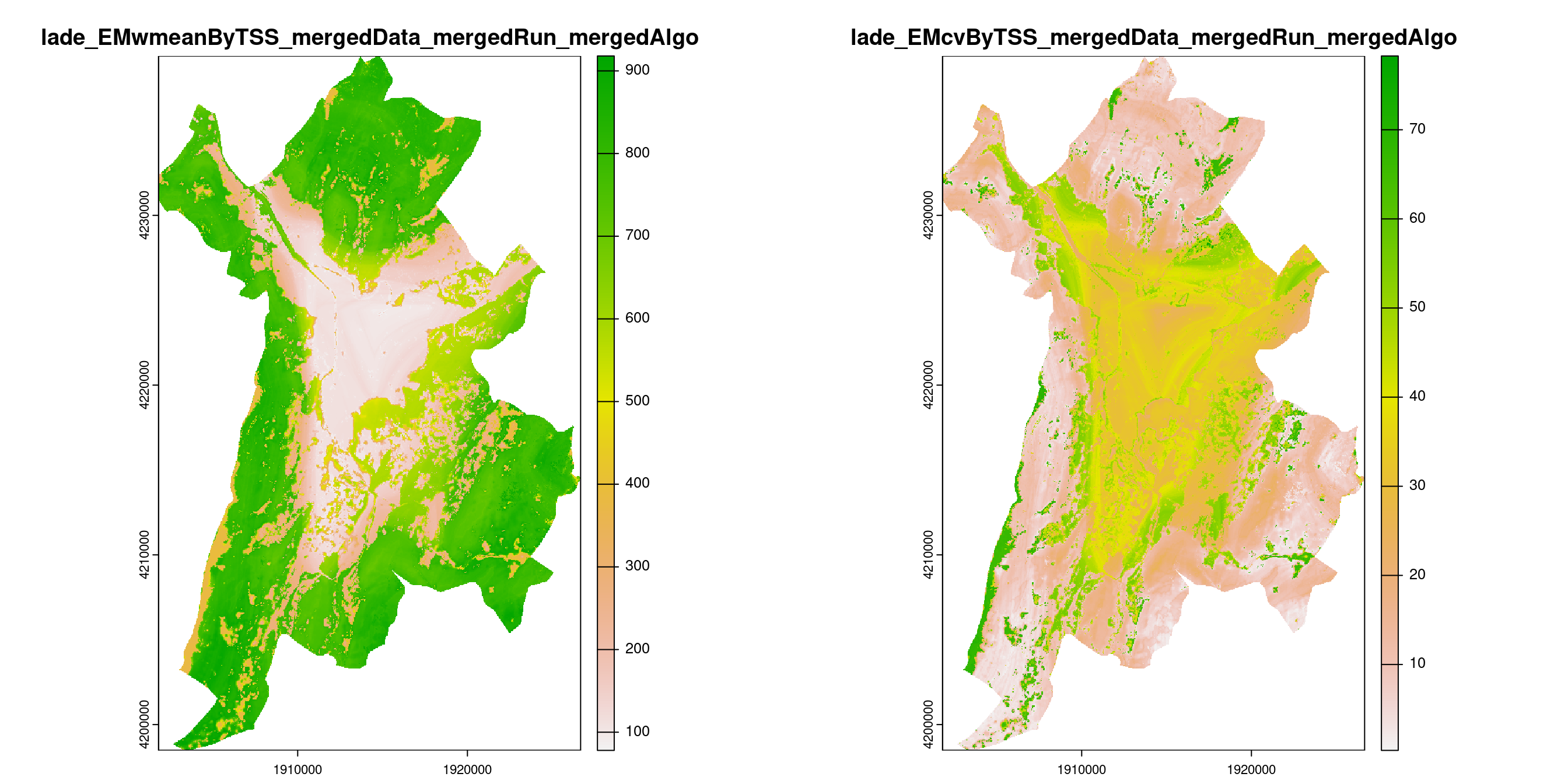
Figure 4.19: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) sur la métropole de Grenoble ; B) Incertitude associée à la projection.
4.4.3 Validation avec une référence française (IFN)
extraction des pixels par polygone (écorégion) et moyenne arithmétique pour chacun :
lade_density lade_current_proj
1 0.002610966057 314.3954183
2 0.000000000000 208.1770335
3 0.000000000000 288.7753780
4 0.000000000000 213.2168022
5 0.000000000000 131.0458221
6 0.000000000000 110.7361702IFN pour LADE, densité + Carte de projection actuelle France


Figure 4.20: A) Carte de densité selon l’IFN ; B) Probabilité d’occurrence selon le modèle d’ensemble.
corrélation non paramétrique de Spearman (par les rangs) :
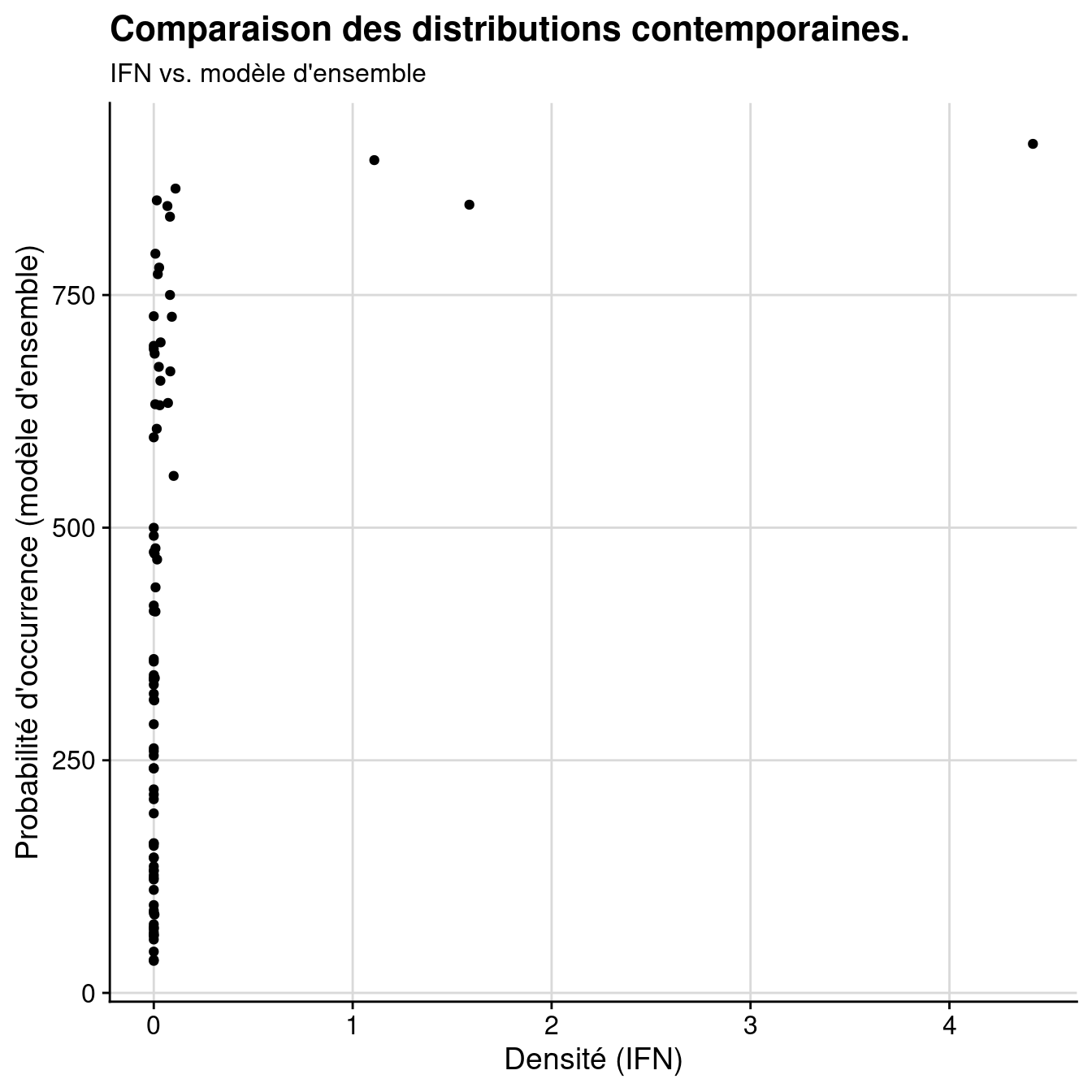
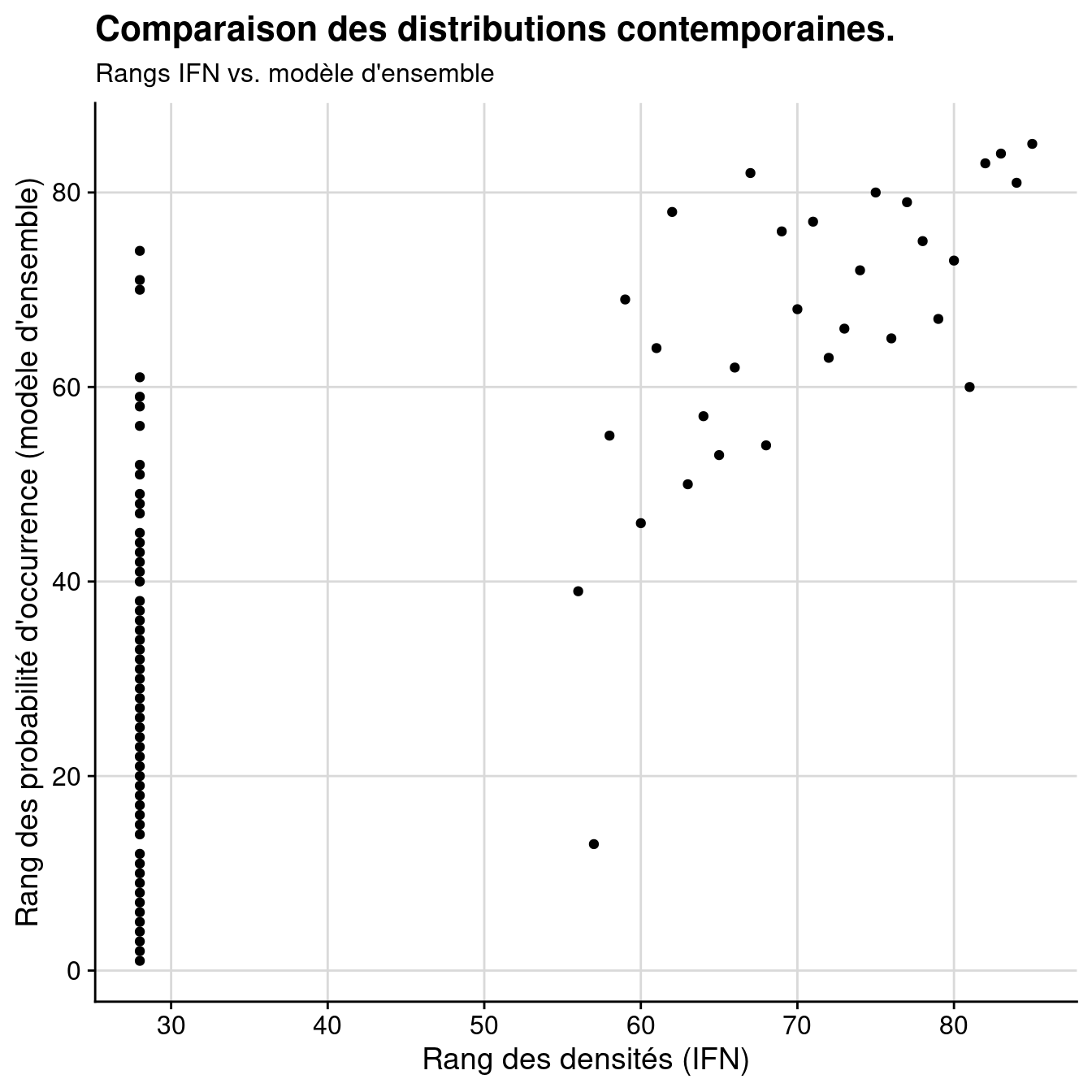
Figure 4.21: Comparaison des distributions contemporaines données par l’IFN vs. modèle d’ensemble : A) Données brutes ; B) Rangs.
[1] 0.7291643694.4.4 Distributions potentielles futures
Future : Projection à l’échelle de l’Europe jusqu’à l’horizon 2100
Toutes les fenêtres temporelles × scénarios pour le GCM « IPSL-CM6A-LR »
- SSP1-2.6
On peut évaluer directement la sortie du modèle d’ensemble.
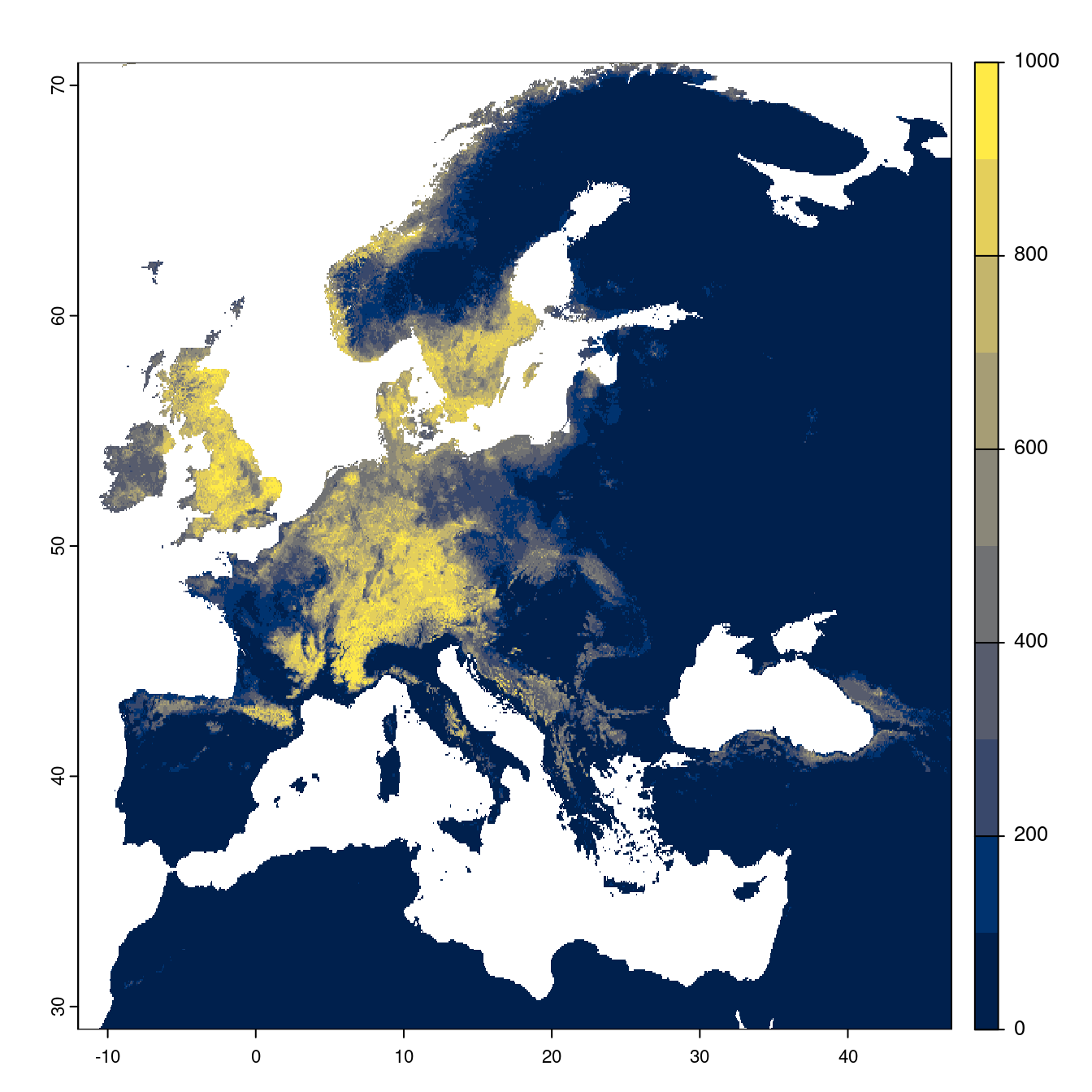
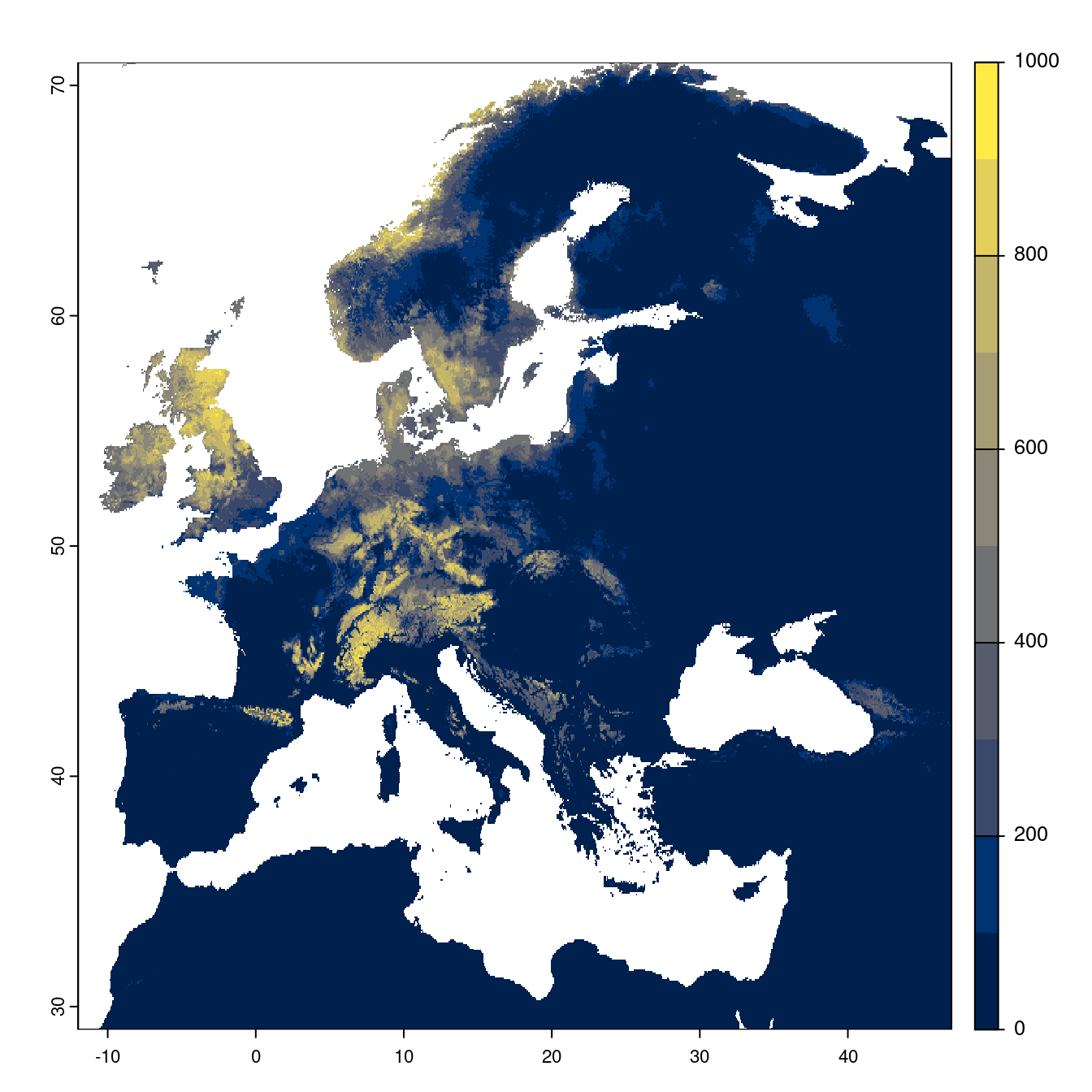

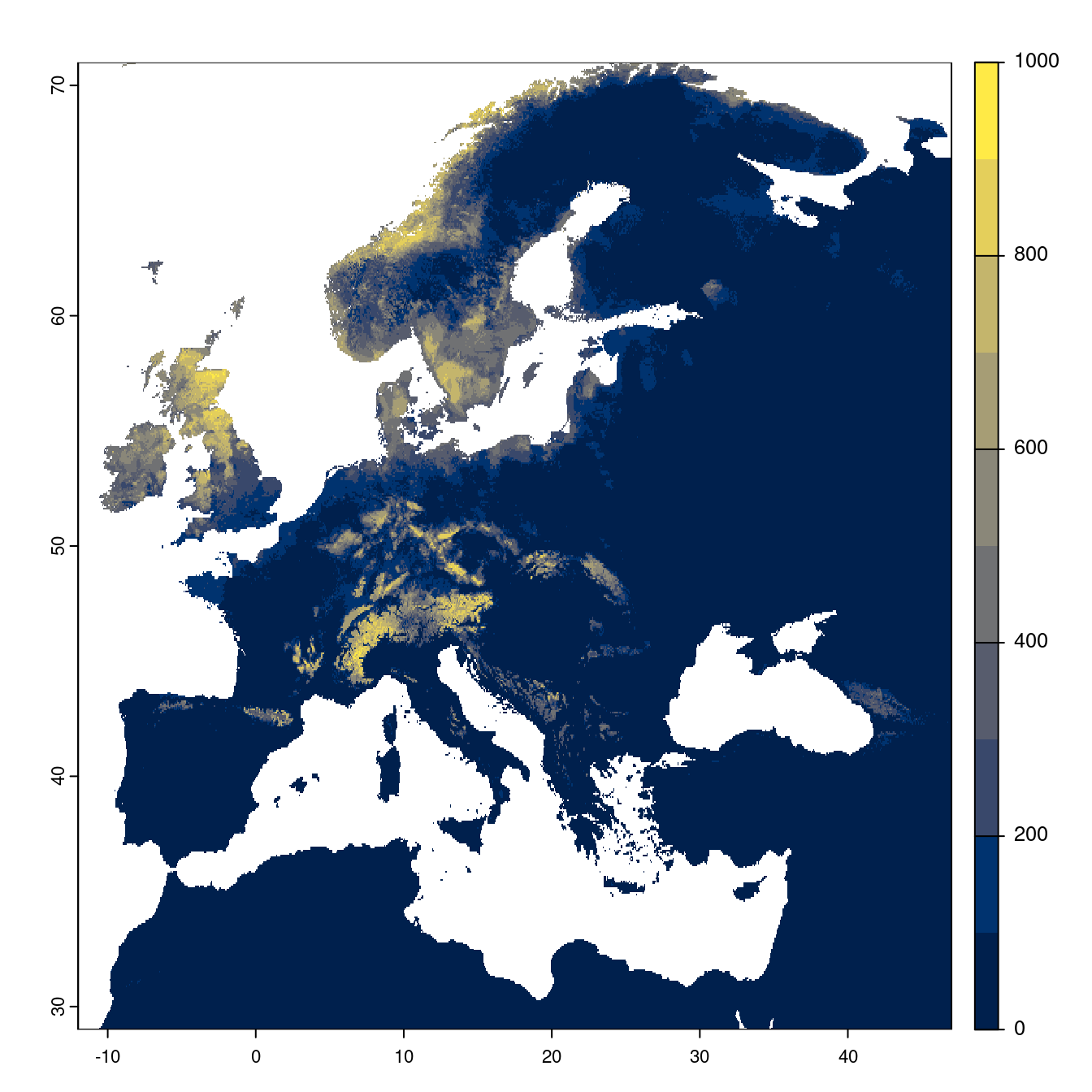
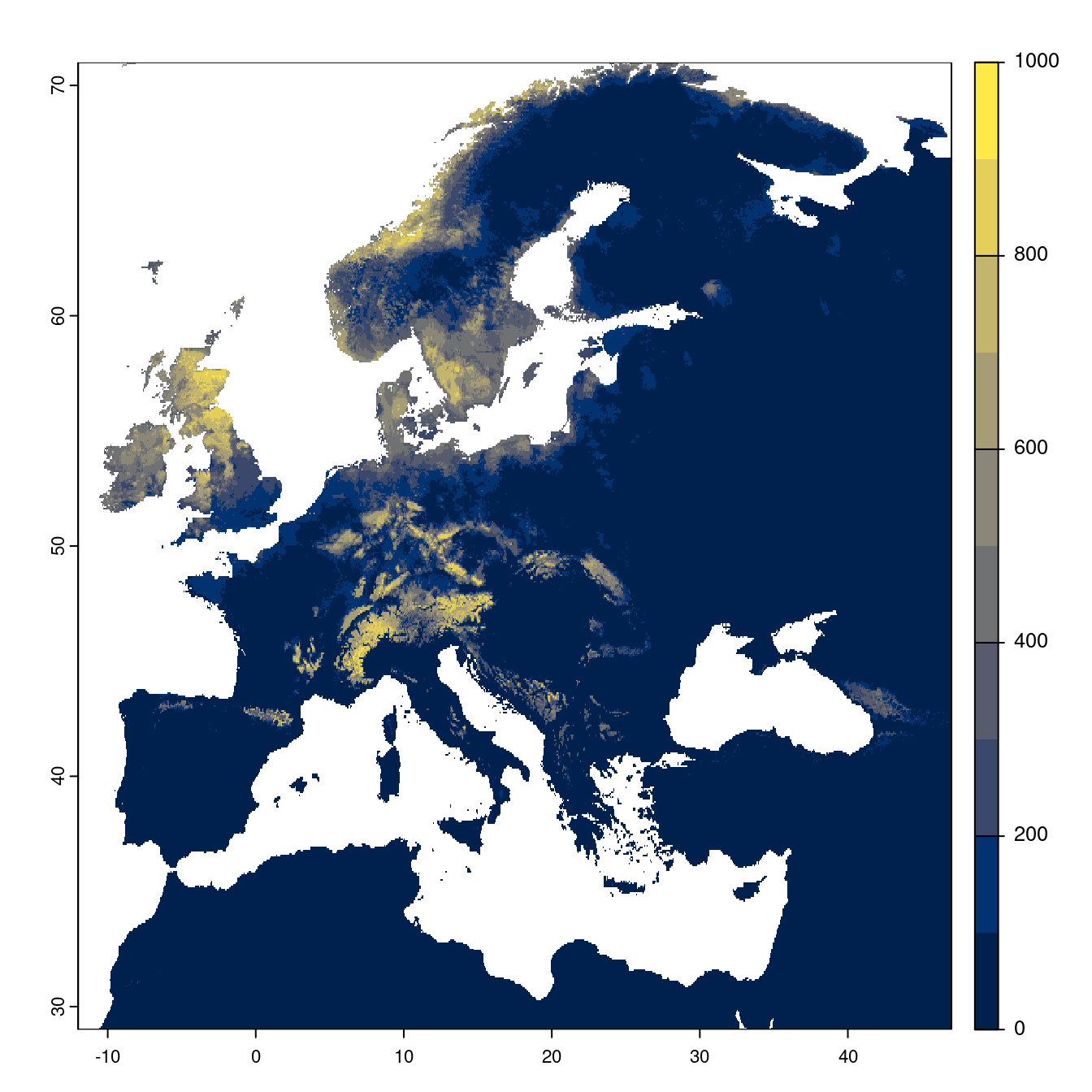
Figure 4.22: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP1-2.6 (modèle d’ensemble).
On obtient pour la métrique TSS une carte unique de moyenne pondérée sur les algorithmes et sur les jeux de pseudo-absences.
- SSP2-4.5

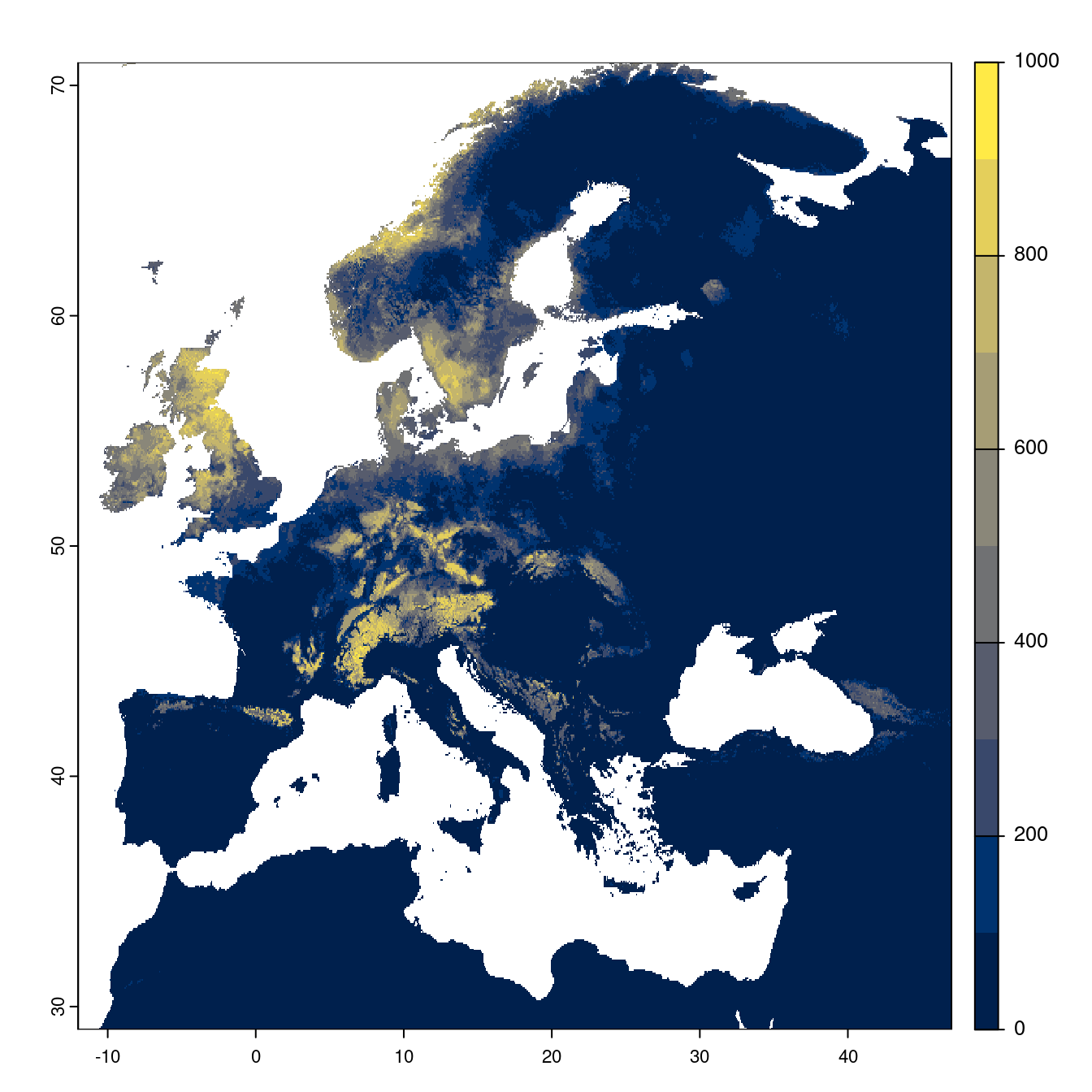

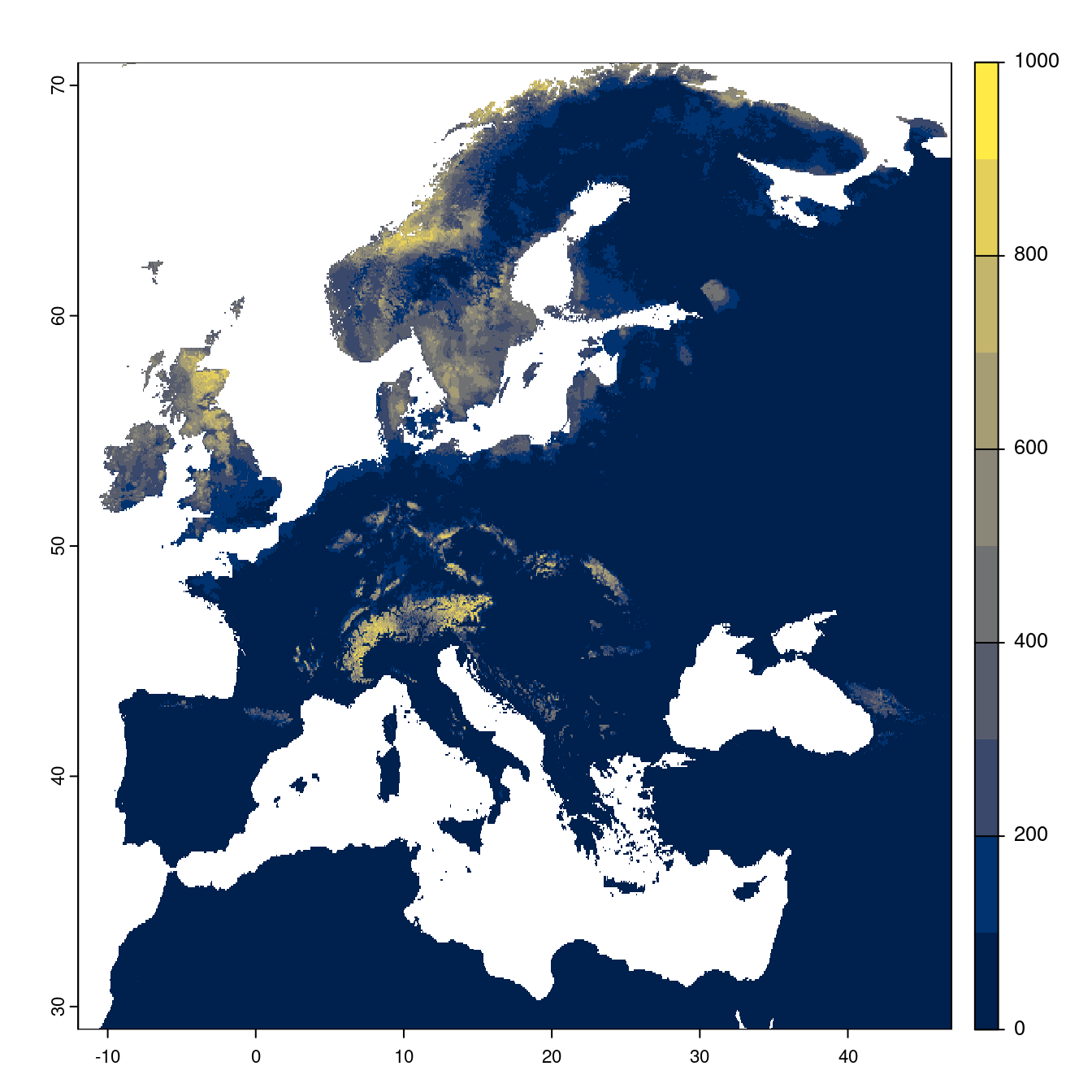
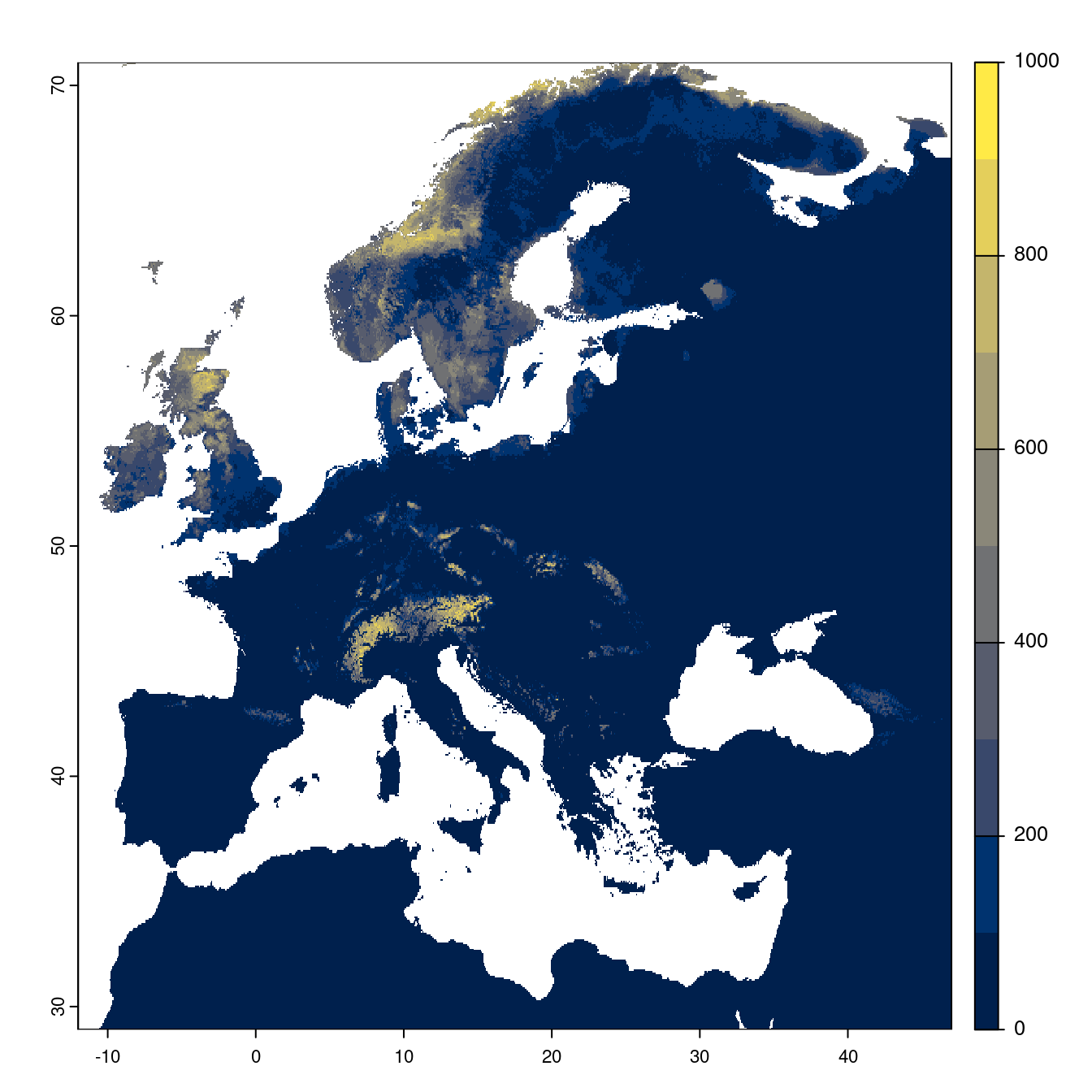
Figure 4.23: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP2-4.5 (modèle d’ensemble).
- SSP3-7.0
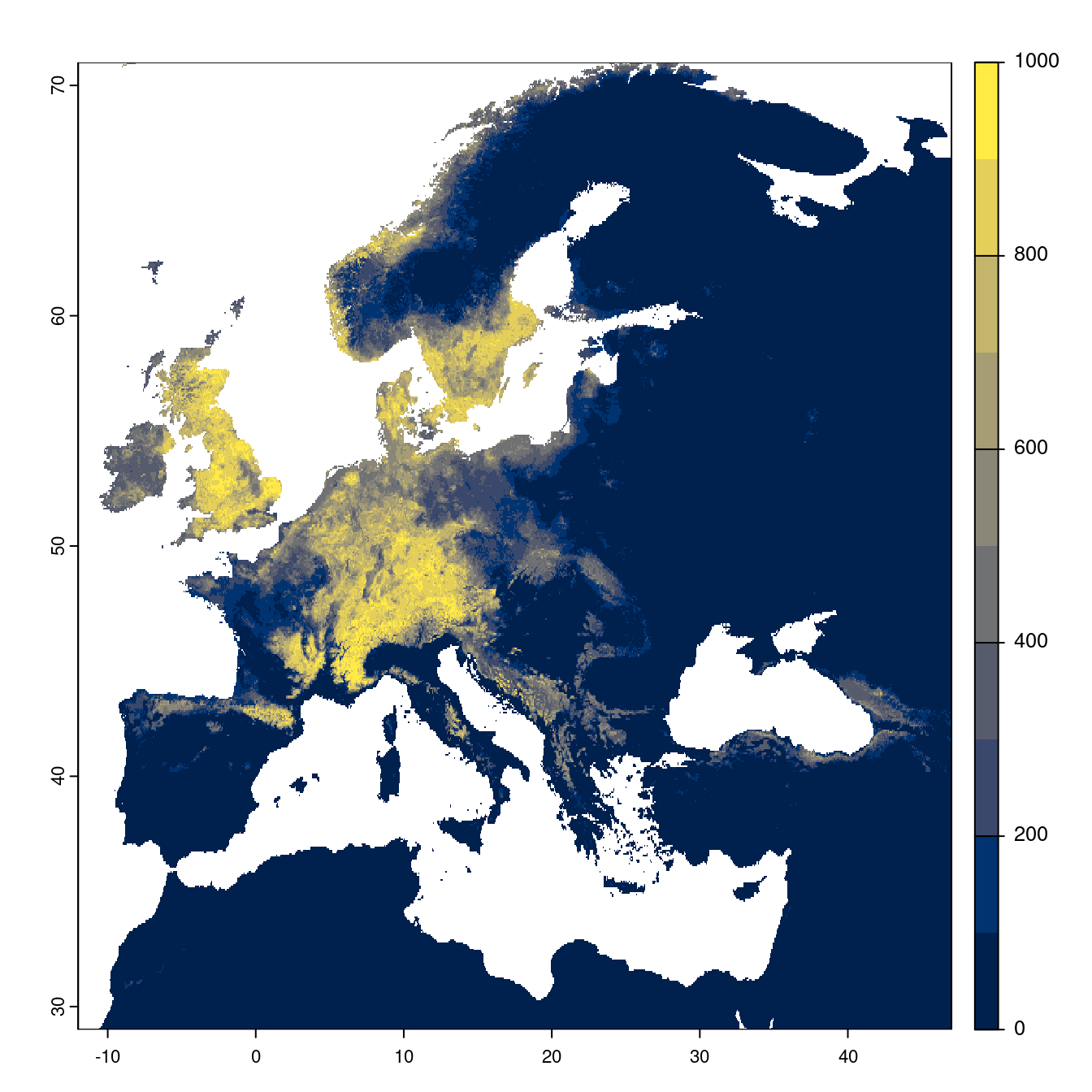
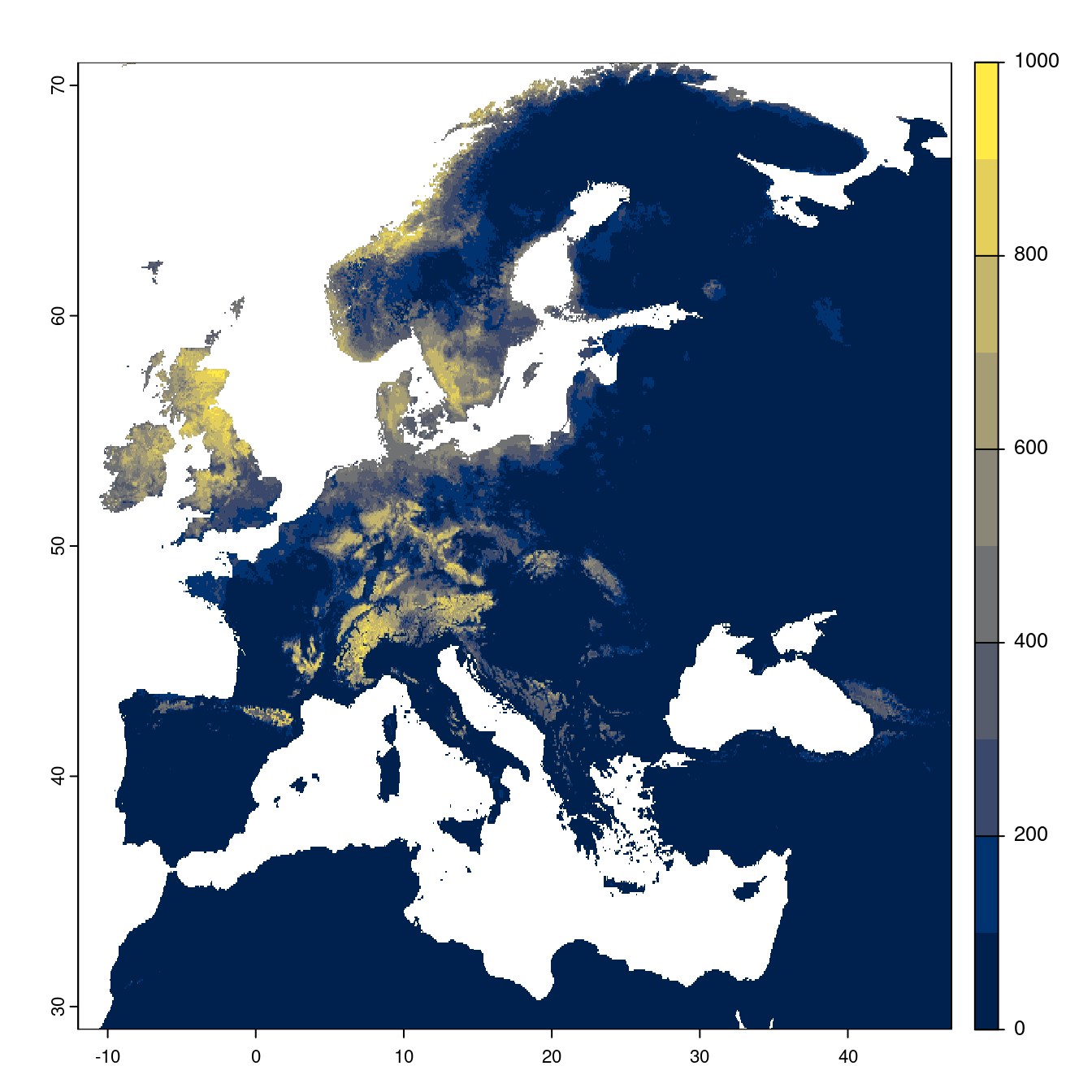

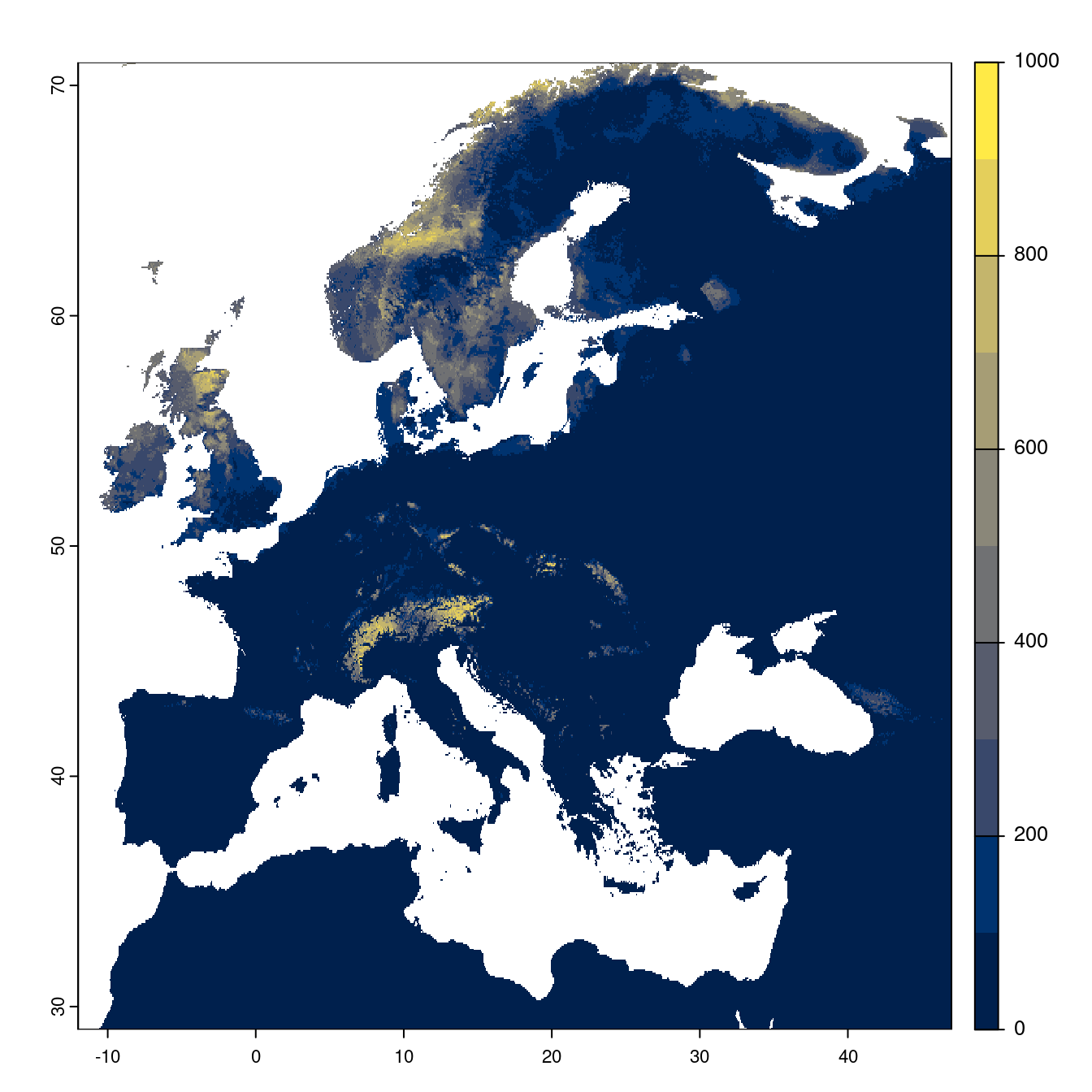

Figure 4.24: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP3-7.0 (modèle d’ensemble).
- SSP5-8.5
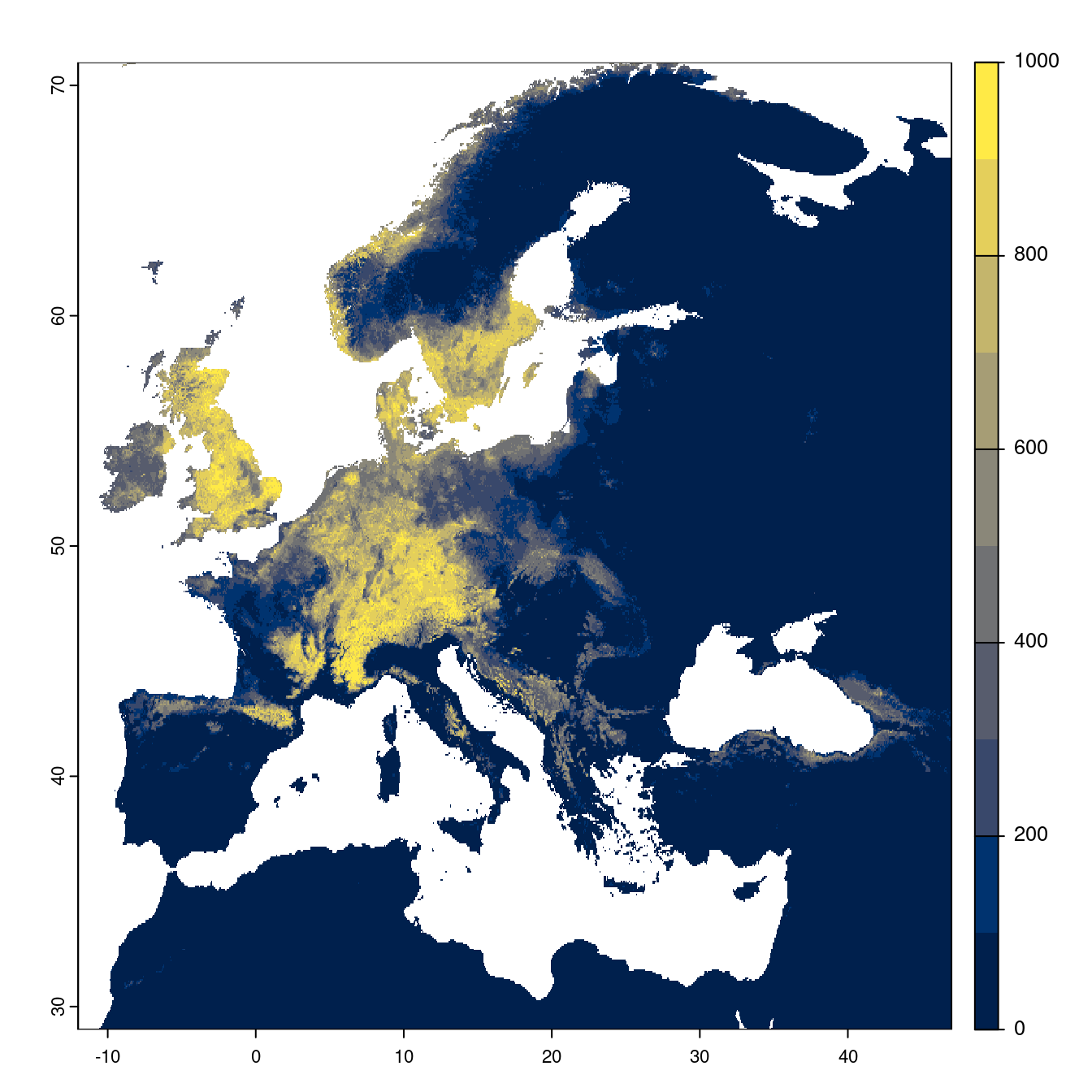
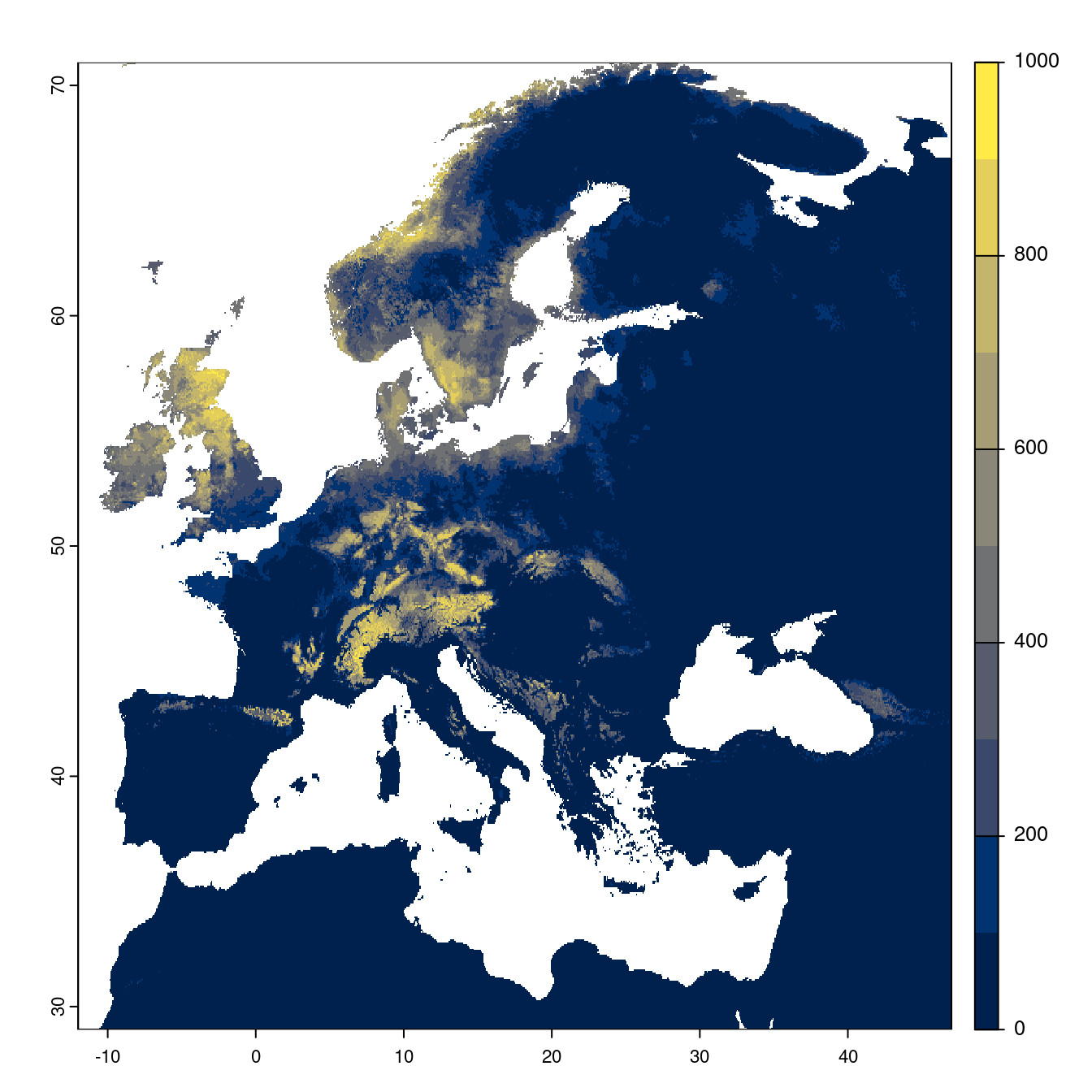
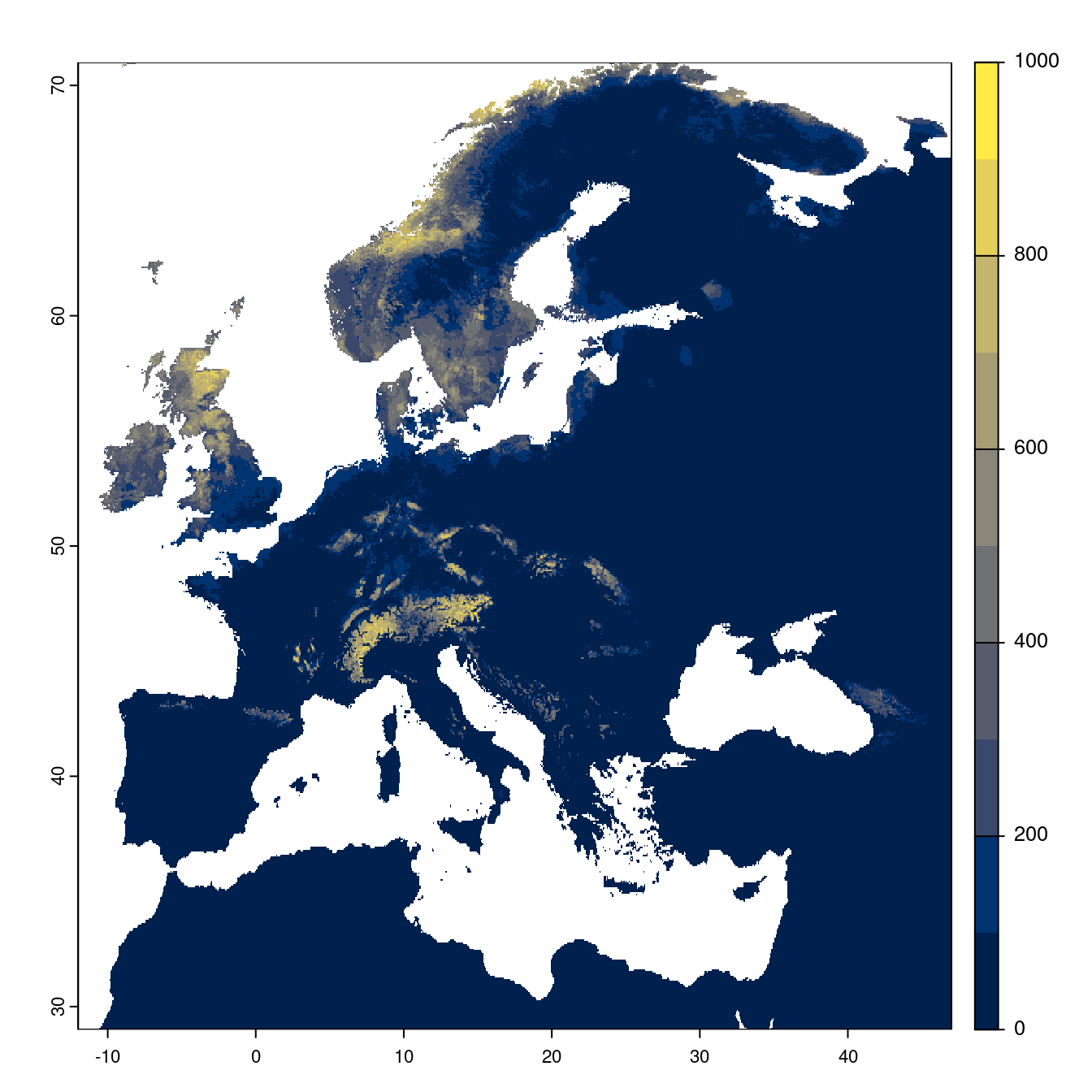
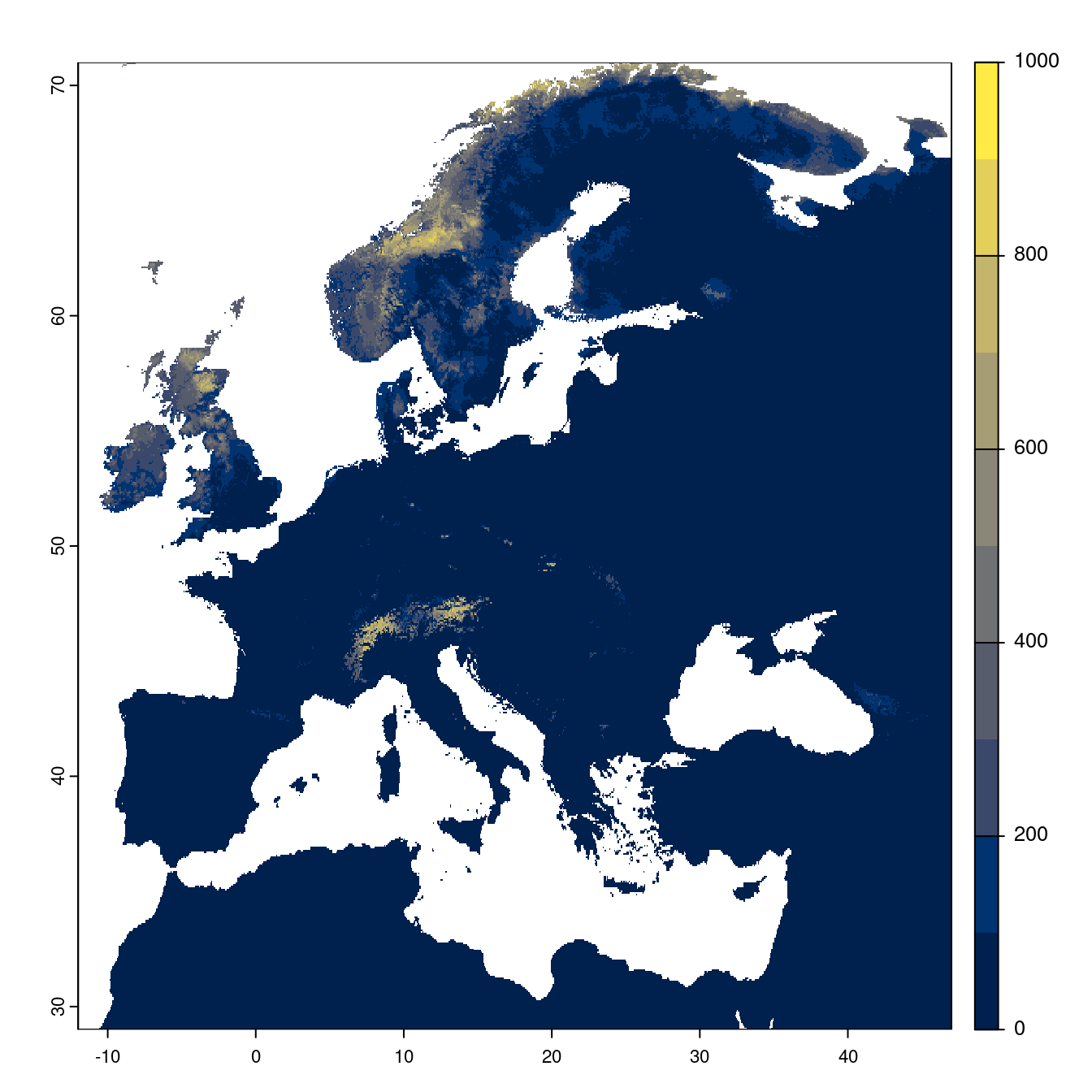
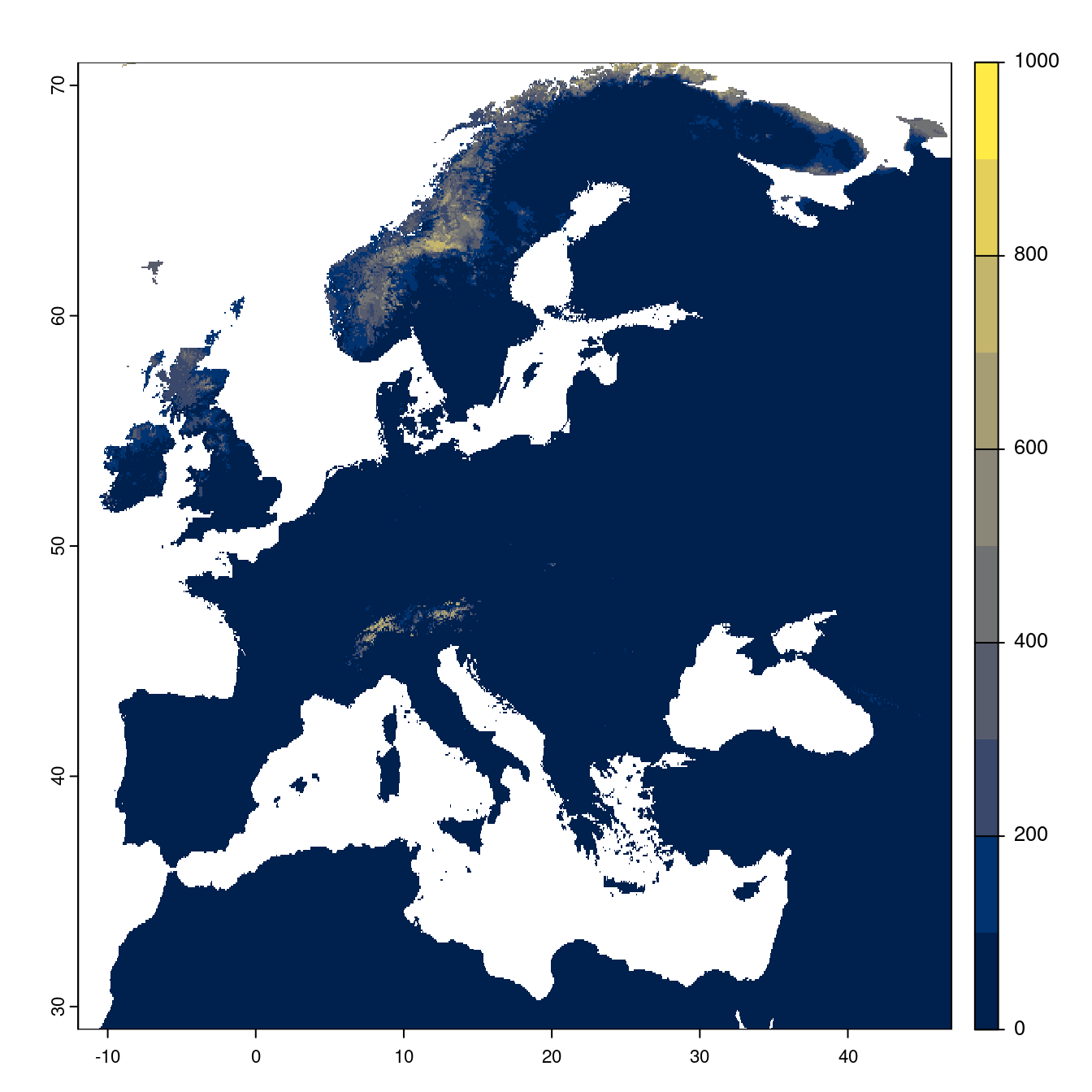
Figure 4.25: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble).
4.5 Inférence à l’échelle de la métropole
- Espèces potentielles pour Grenoble (forte probabilité de présence à l’horizon 2100)
- Croisement avec températures de surface 2019 (fournie par la métropole)
- → conditions climatiques favorables à fine échelle
à peu près 7 pixels × 9 (WorldClim)
4.5.1 Distributions potentielles futures
Même chose que précédemment mais sur les rasters de Grenoble
- SSP1-2.6
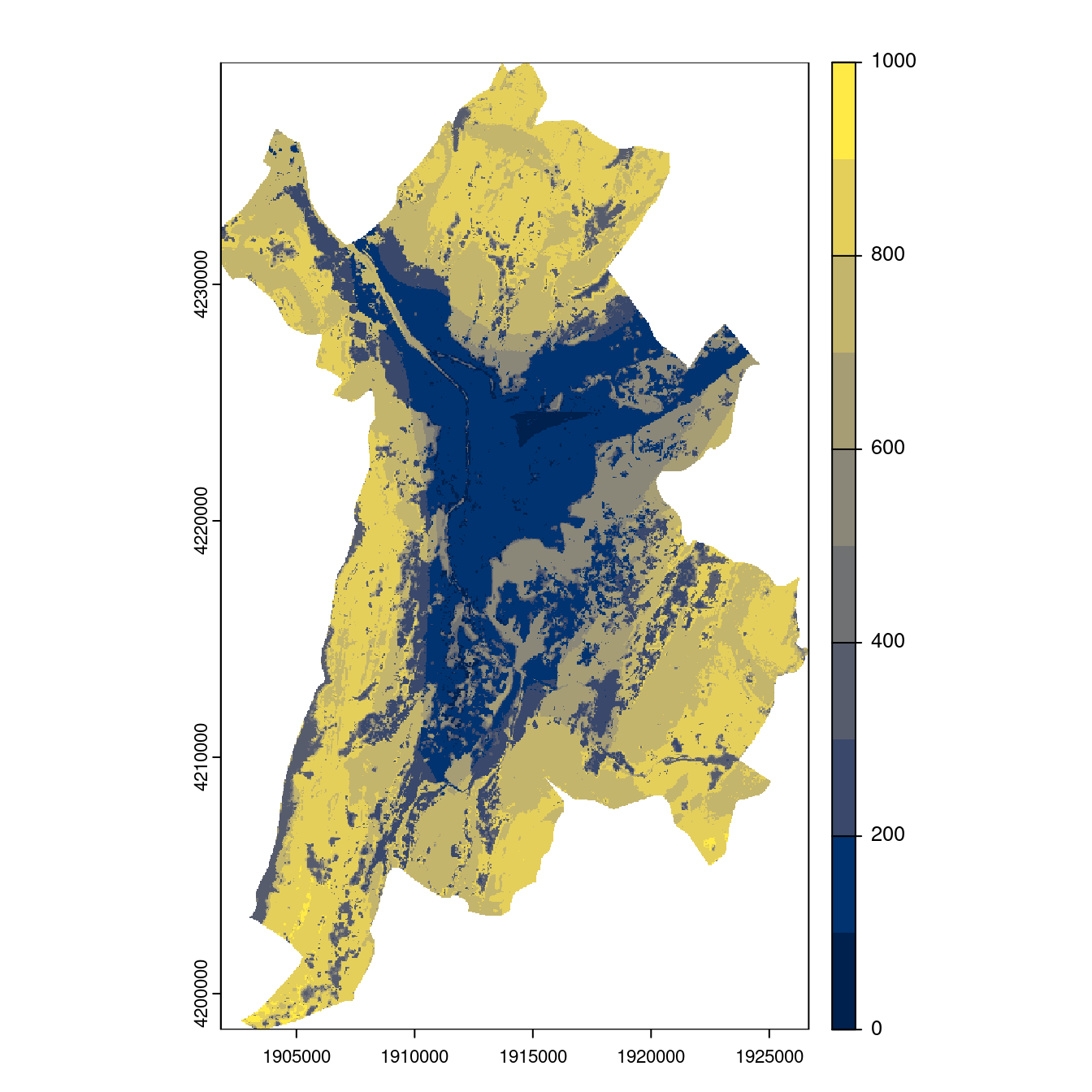
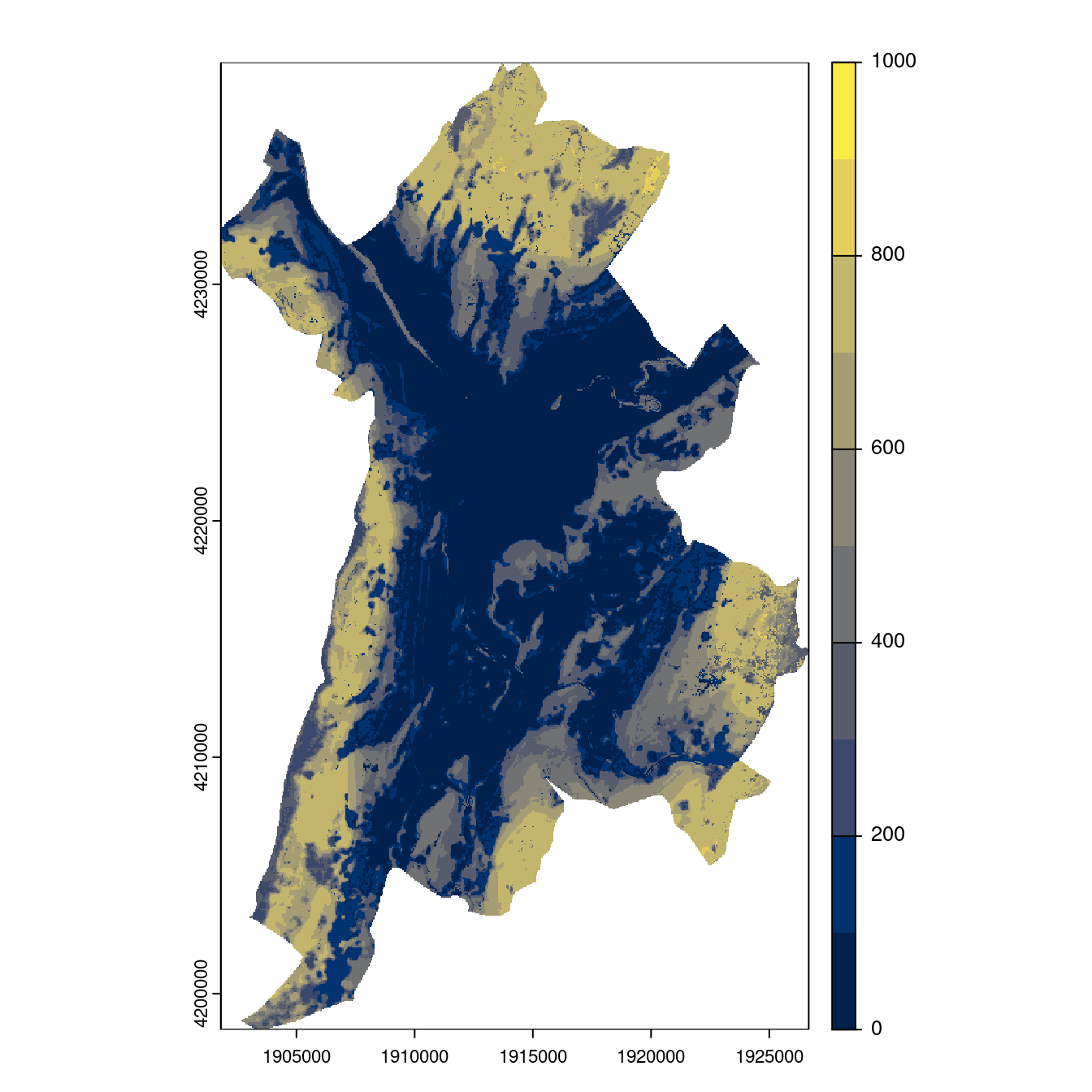
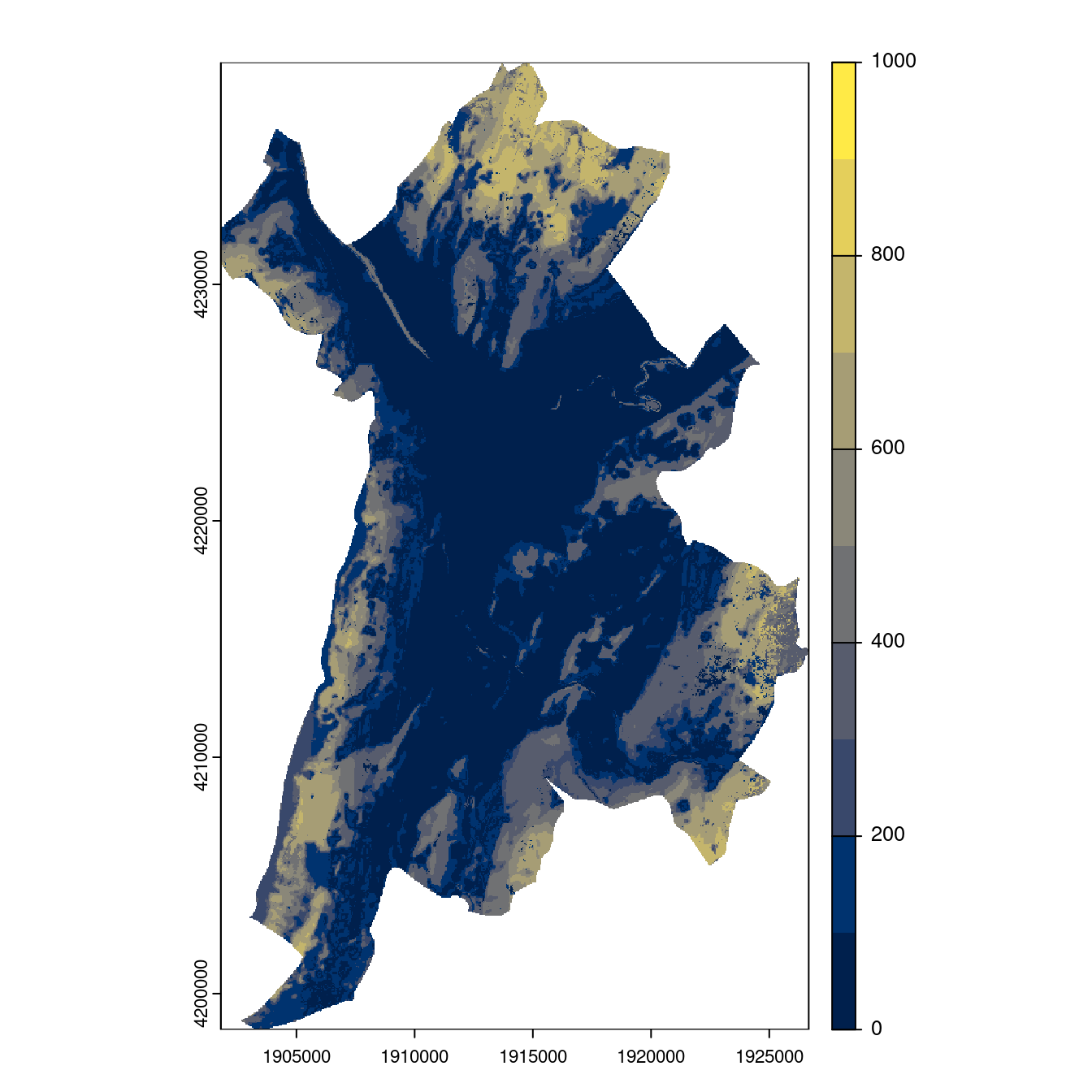
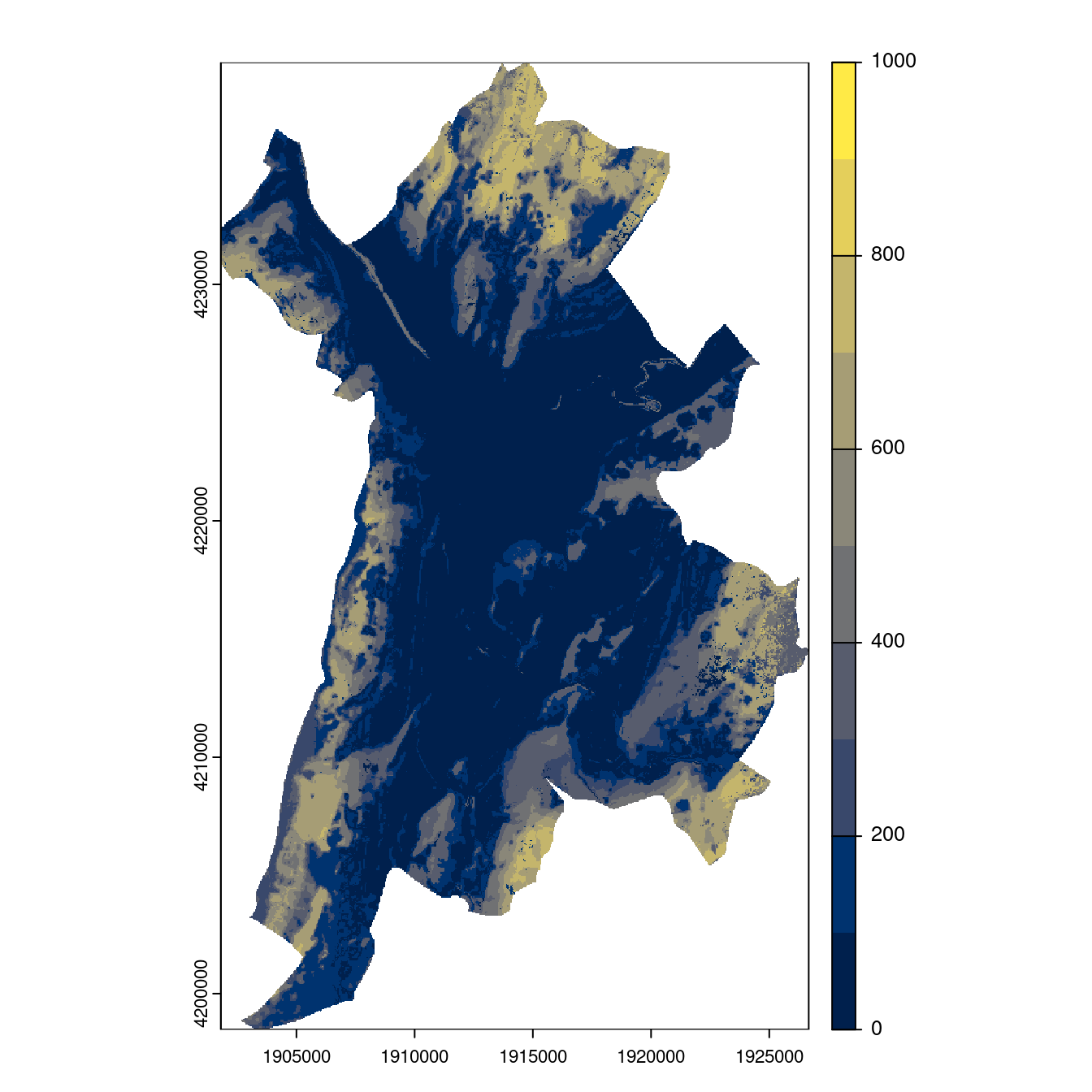
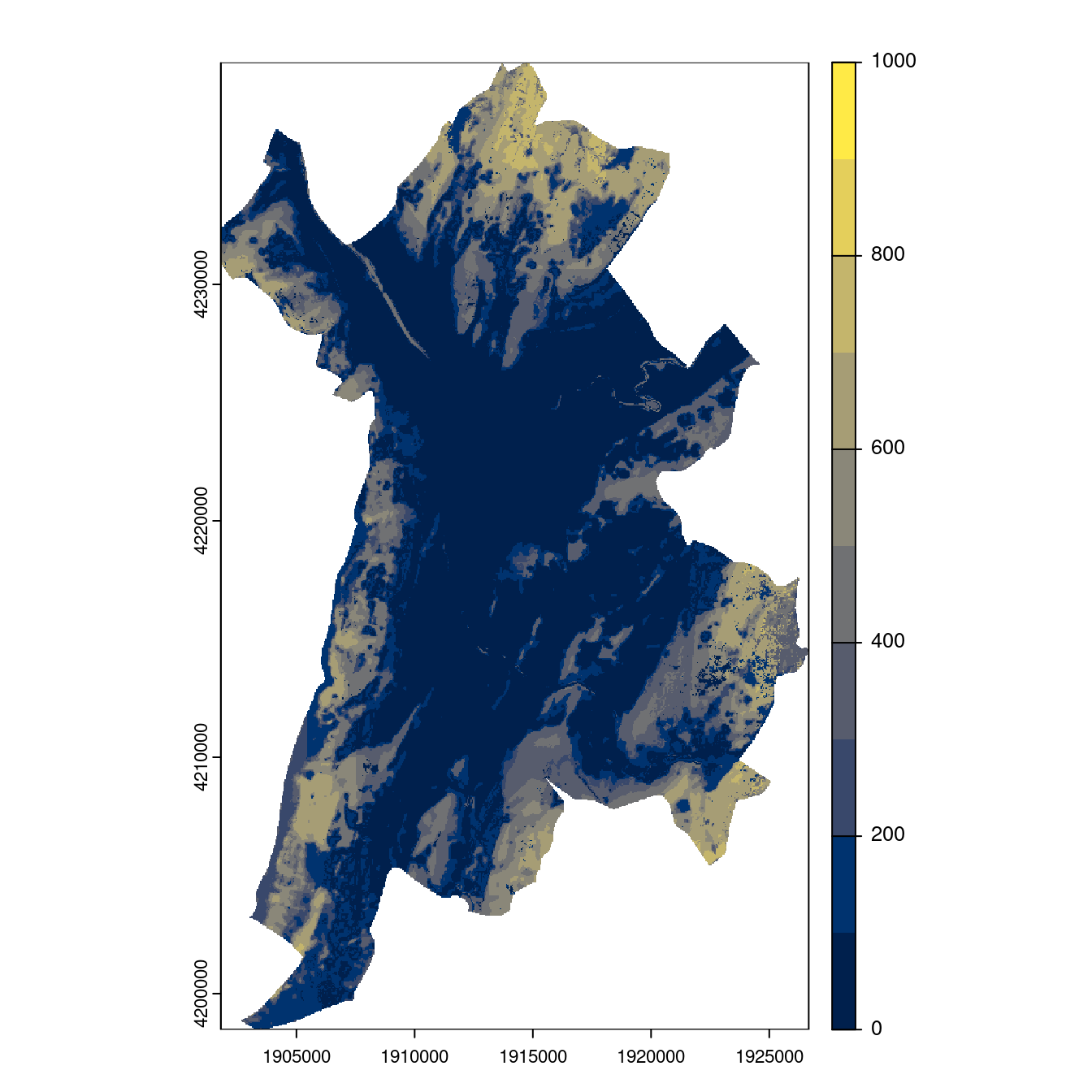
Figure 4.26: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP2-4.5

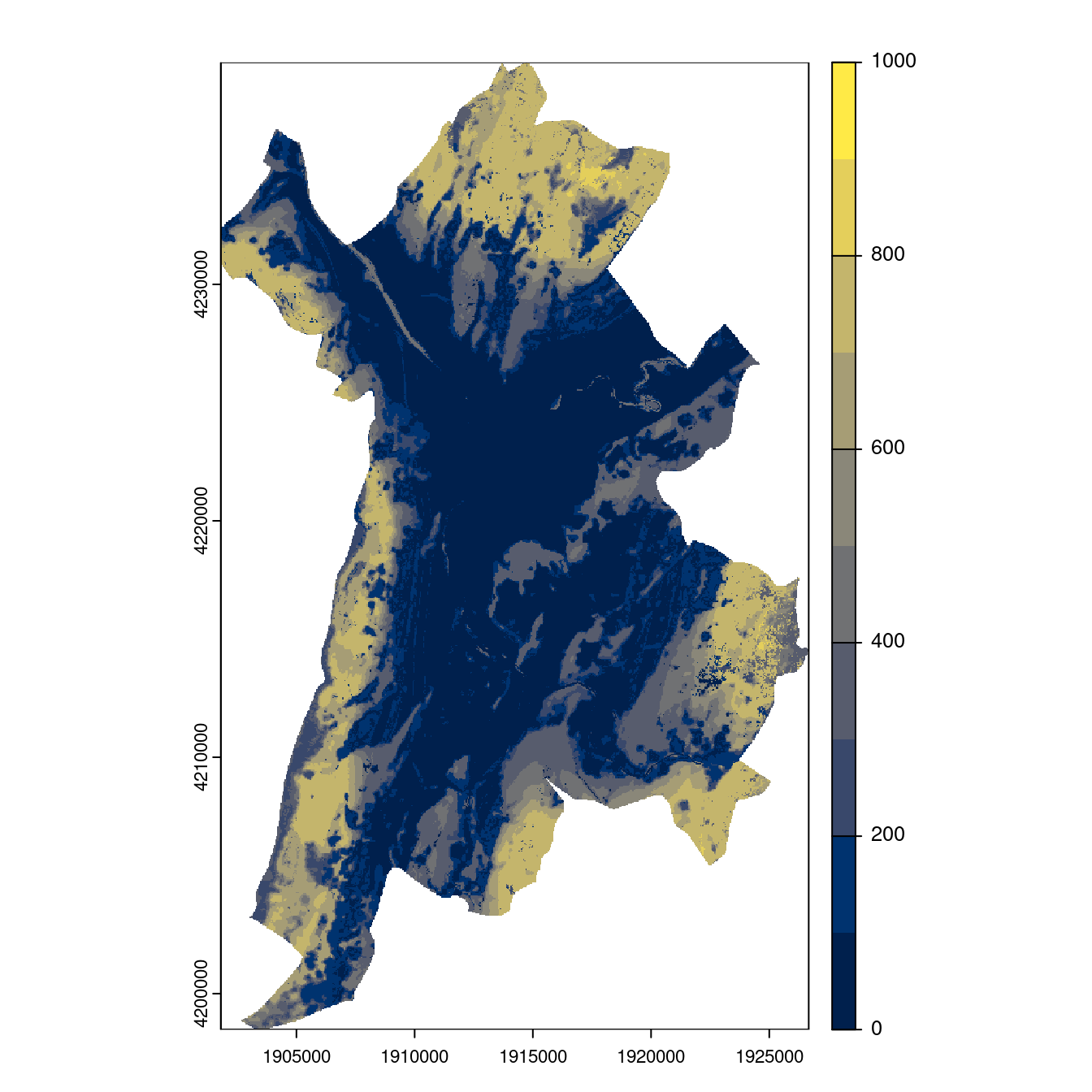


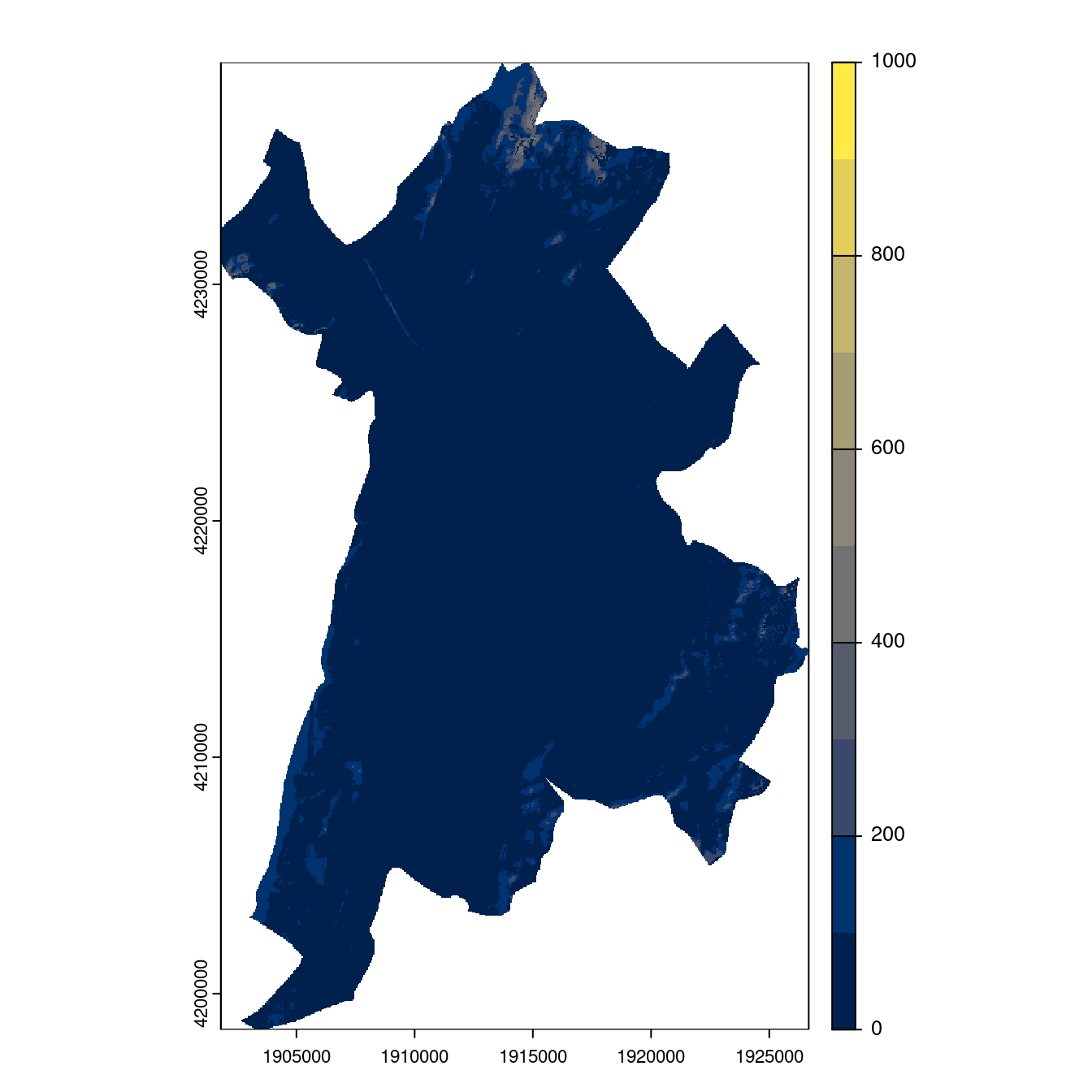
Figure 4.27: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP3-7.0
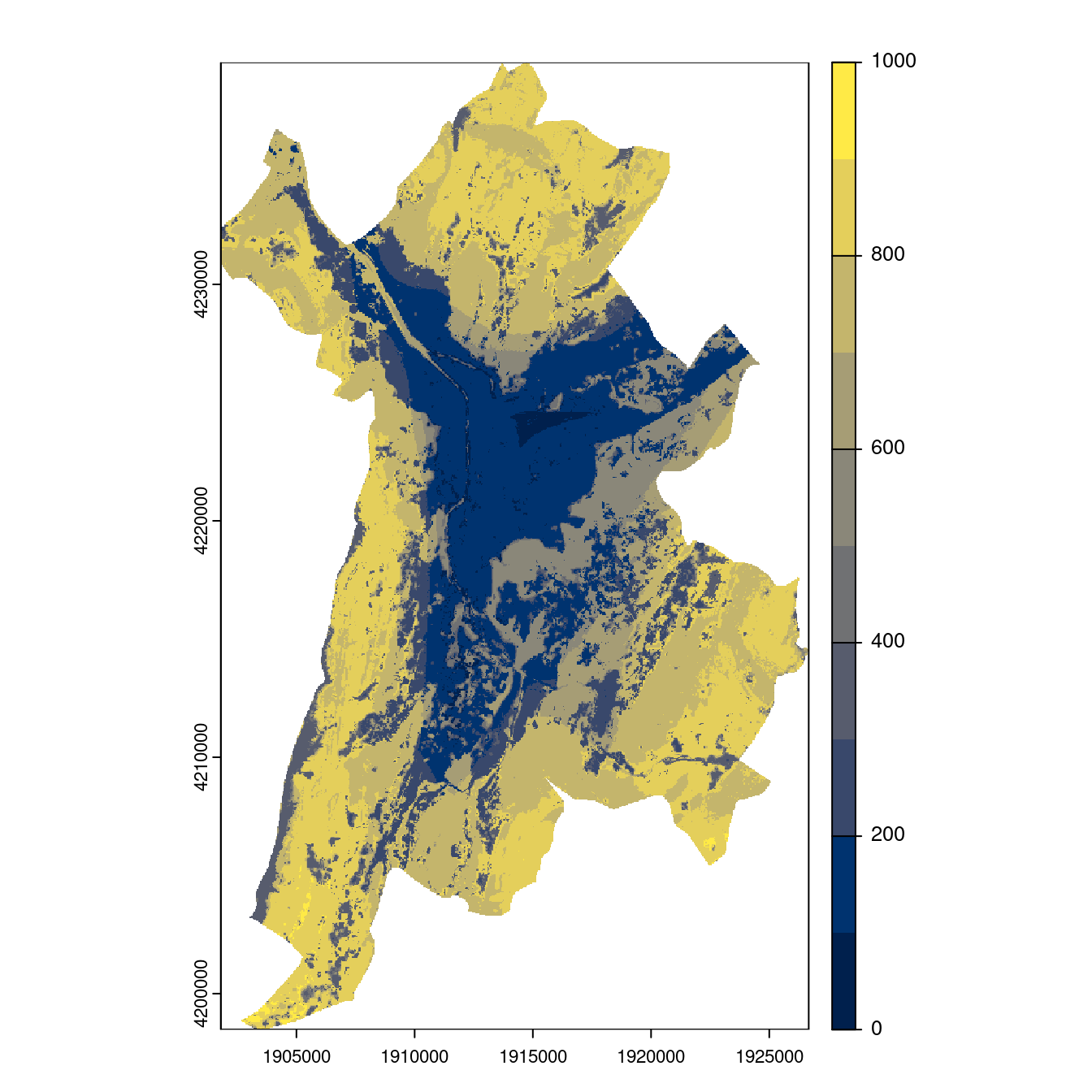
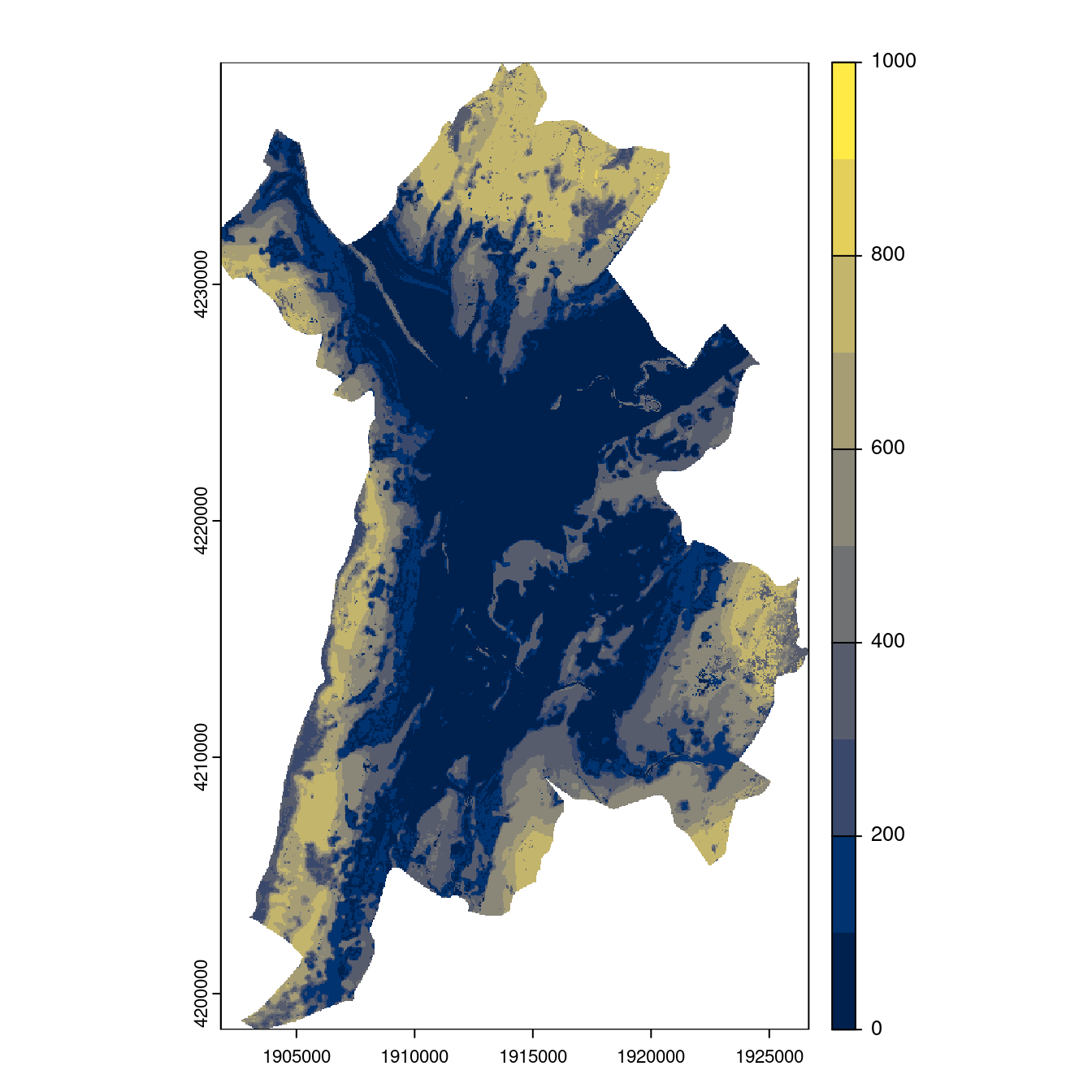
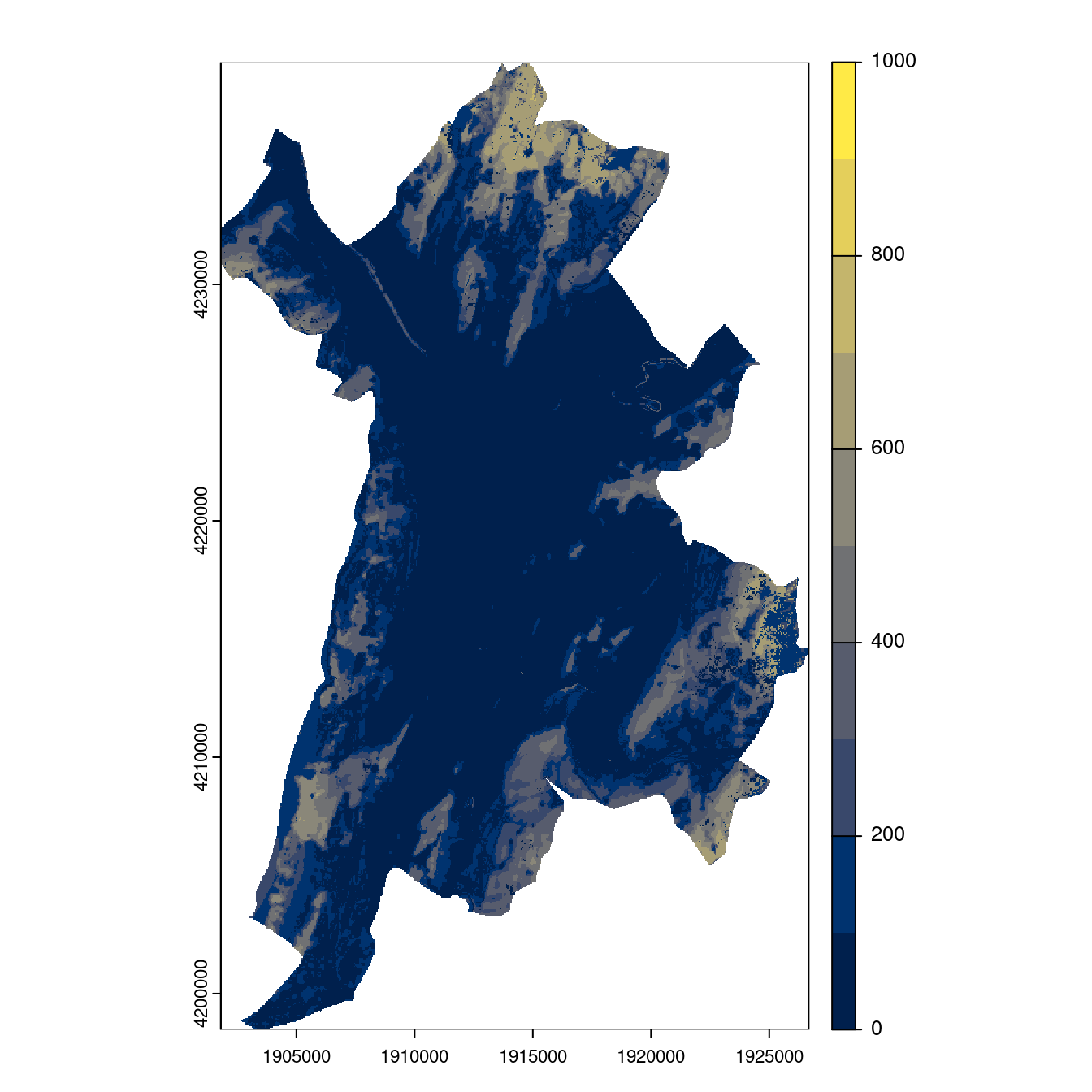
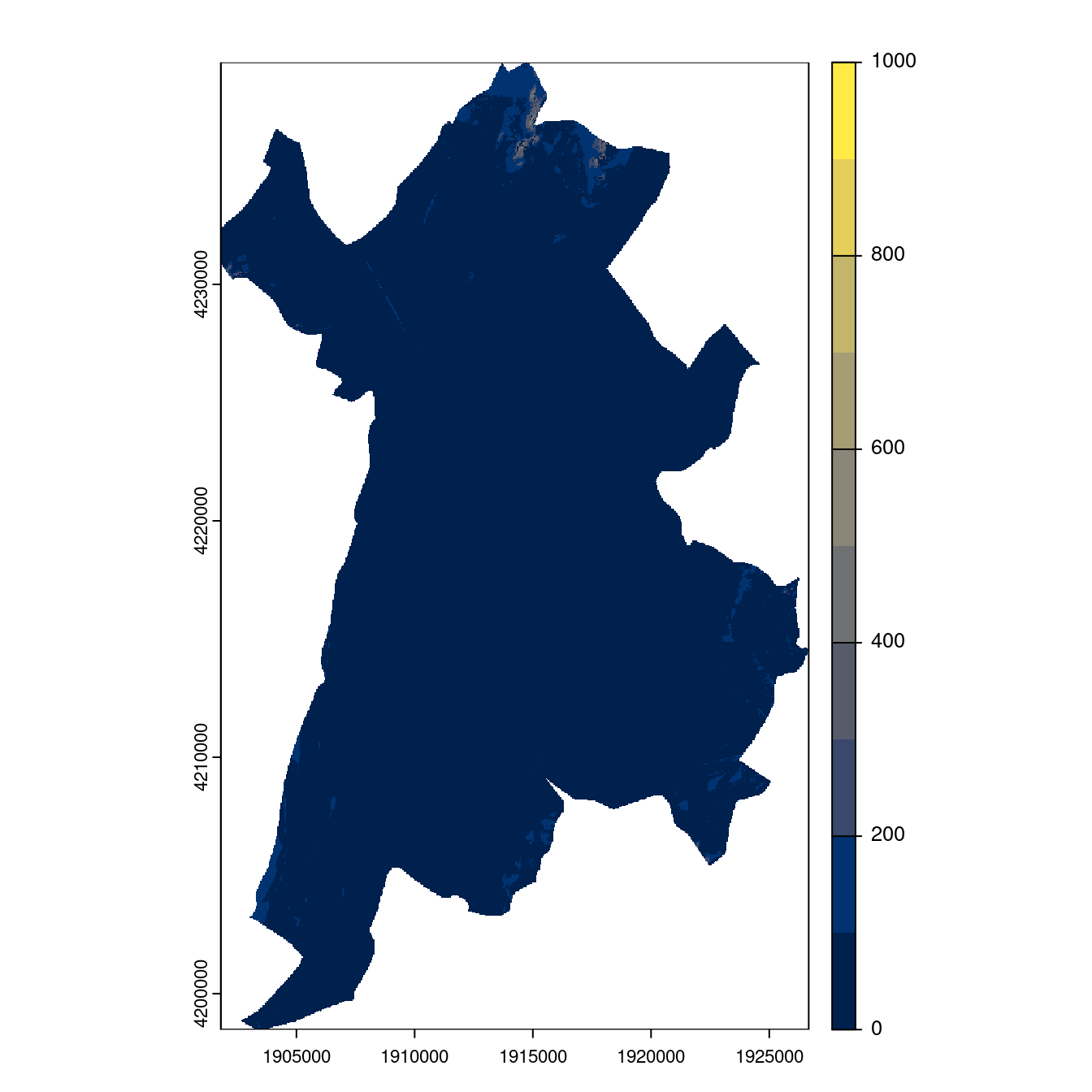
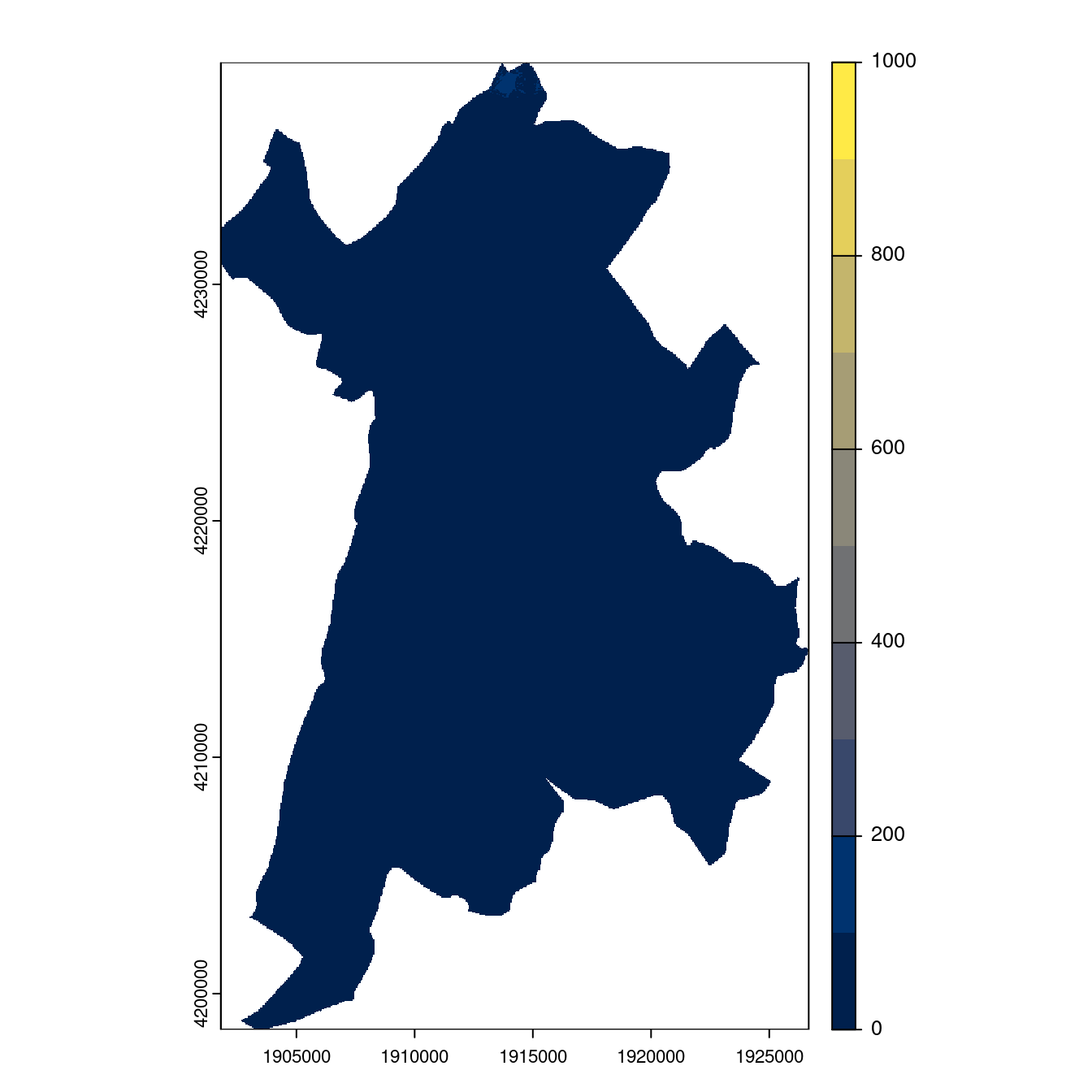
Figure 4.28: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP5-8.5

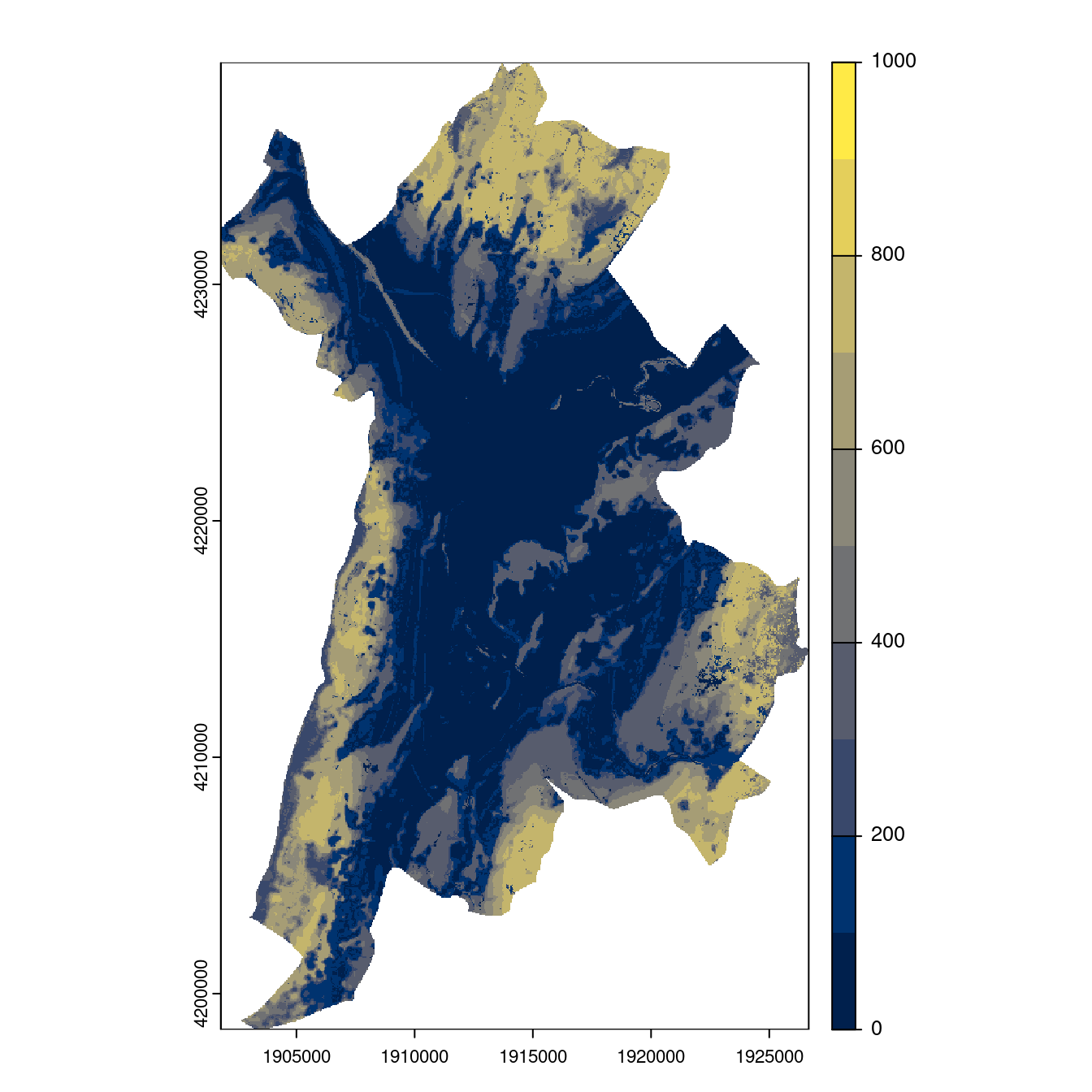
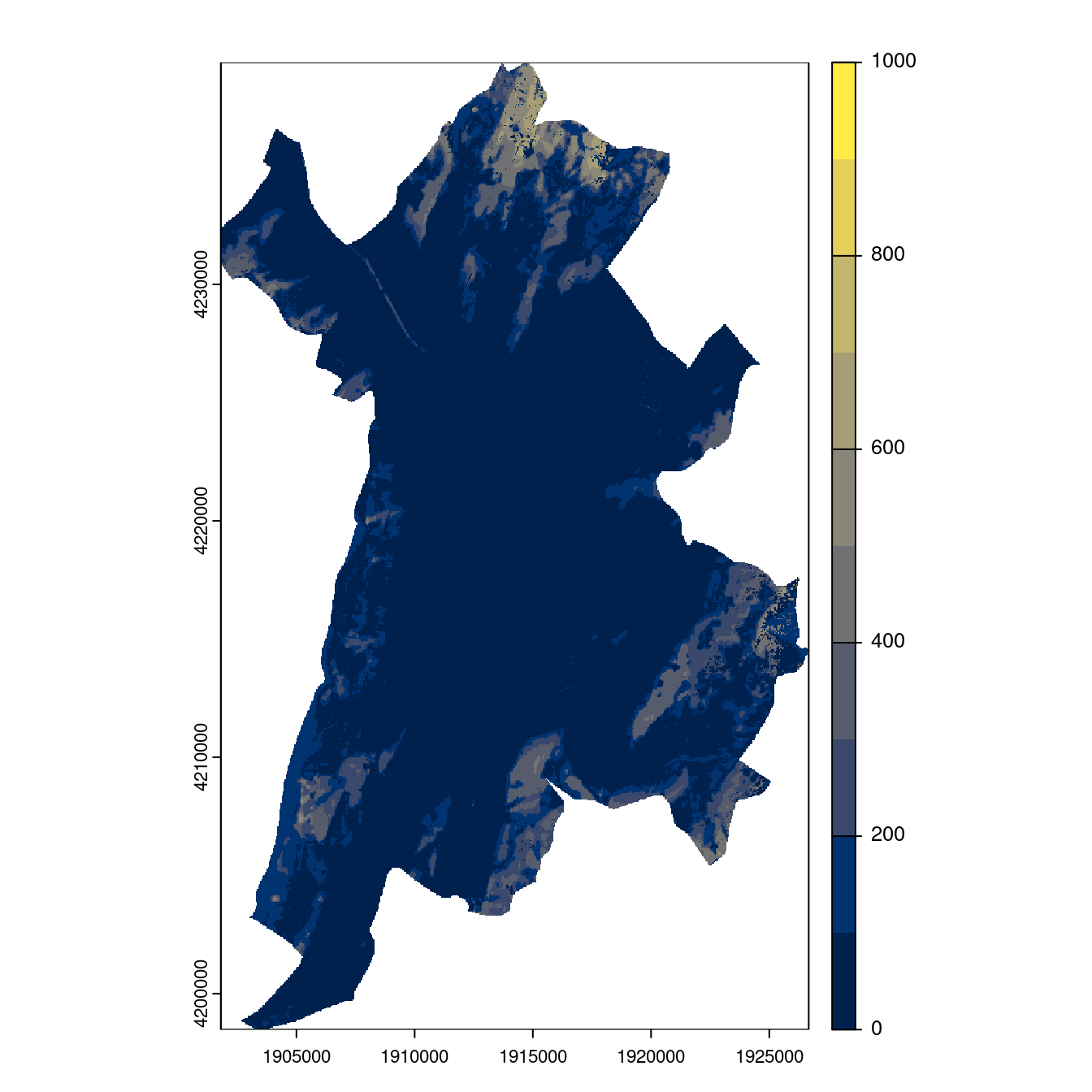
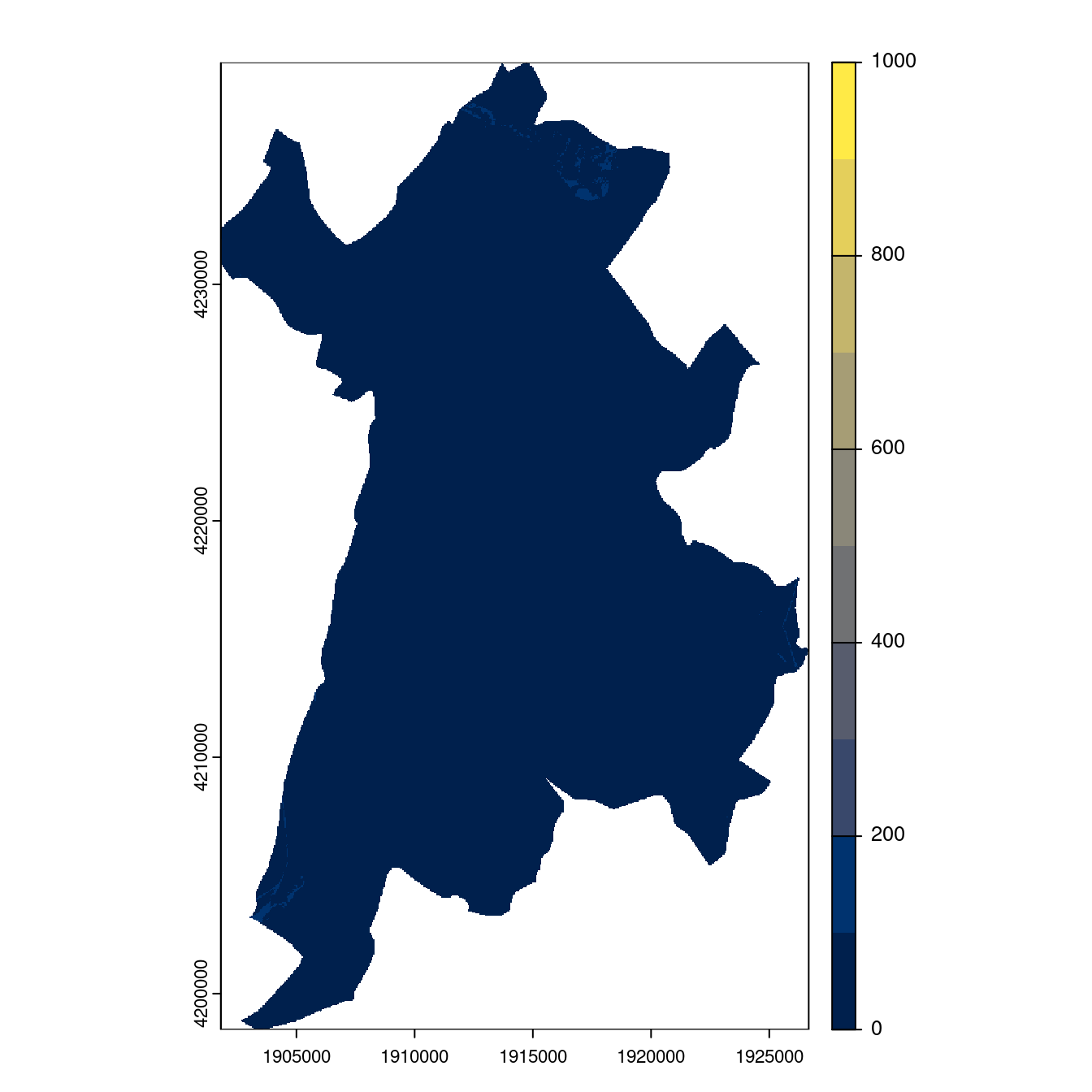
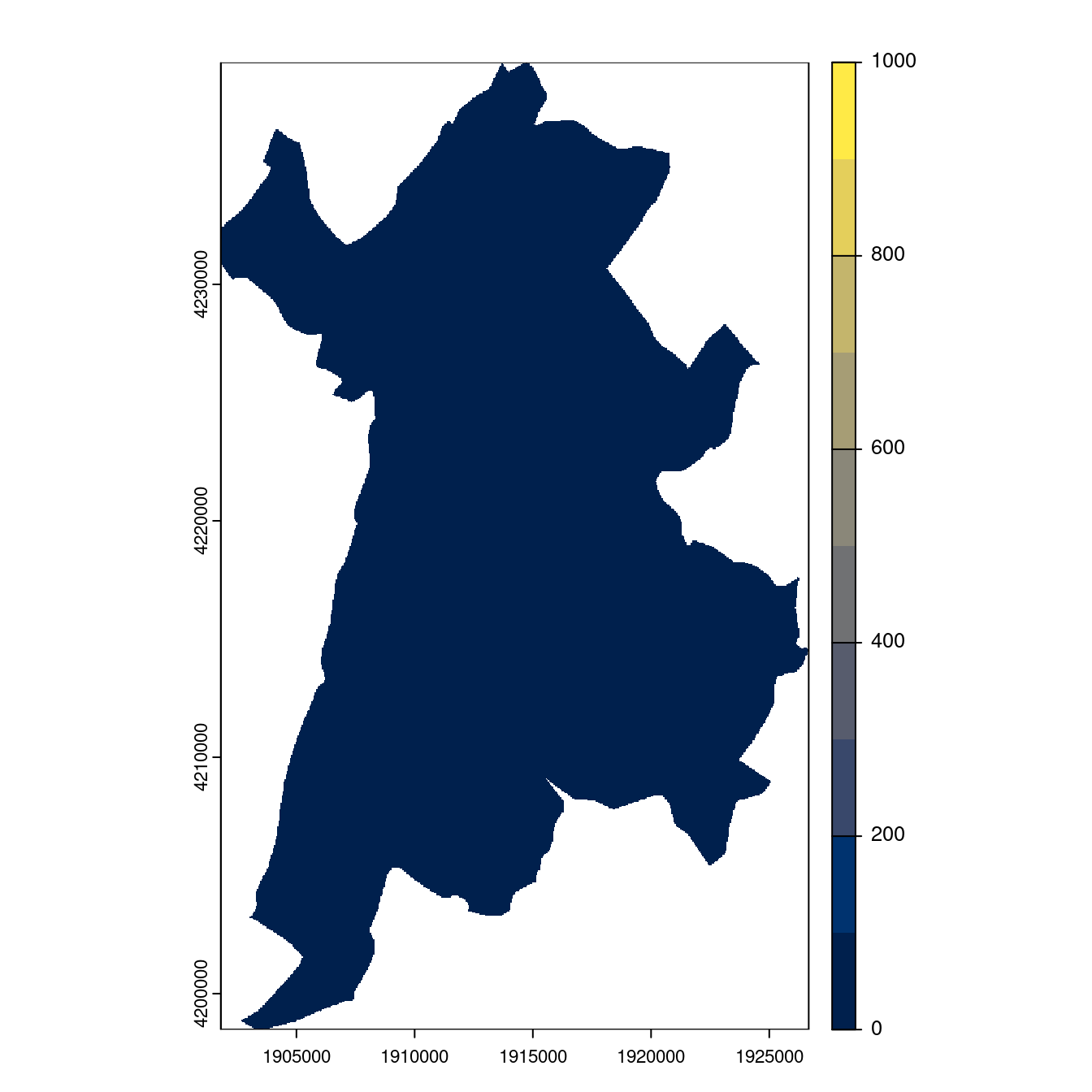
Figure 4.29: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
4.5.2 Évolution temporelle
# A tibble: 17 × 8
min q1 median mean q3 max ssp year
<table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <fct> <dbl>
1 75 260 724 564.39522881 801 918 0 2000
2 42 76 237 317.07329928 549 820 126 2040
3 41 64 104 223.65627700 365 778 126 2060
4 42 67 102 224.64448145 364 770 126 2080
5 40 66 102 222.33854693 384 777 126 2100
6 41 75 160 294.21558417 473 837 245 2040
7 41 61 80 161.04168501 219 731 245 2060
8 39 46 62 93.11846866 93 681 245 2080
9 38 43 48 60.86930541 64 511 245 2100
10 40 69 192 293.38635483 504 809 370 2040
11 38 53 76 149.48759861 180 725 370 2060
12 38 44 47 54.65729997 58 457 370 2080
13 36 42 45 46.54639595 48 108 370 2100
14 40 73 161 284.24752618 458 786 585 2040
15 39 44 58 99.53565812 97 671 585 2060
16 36 43 45 47.77426232 47 122 585 2080
17 30 38 41 41.54138210 45 65 585 2100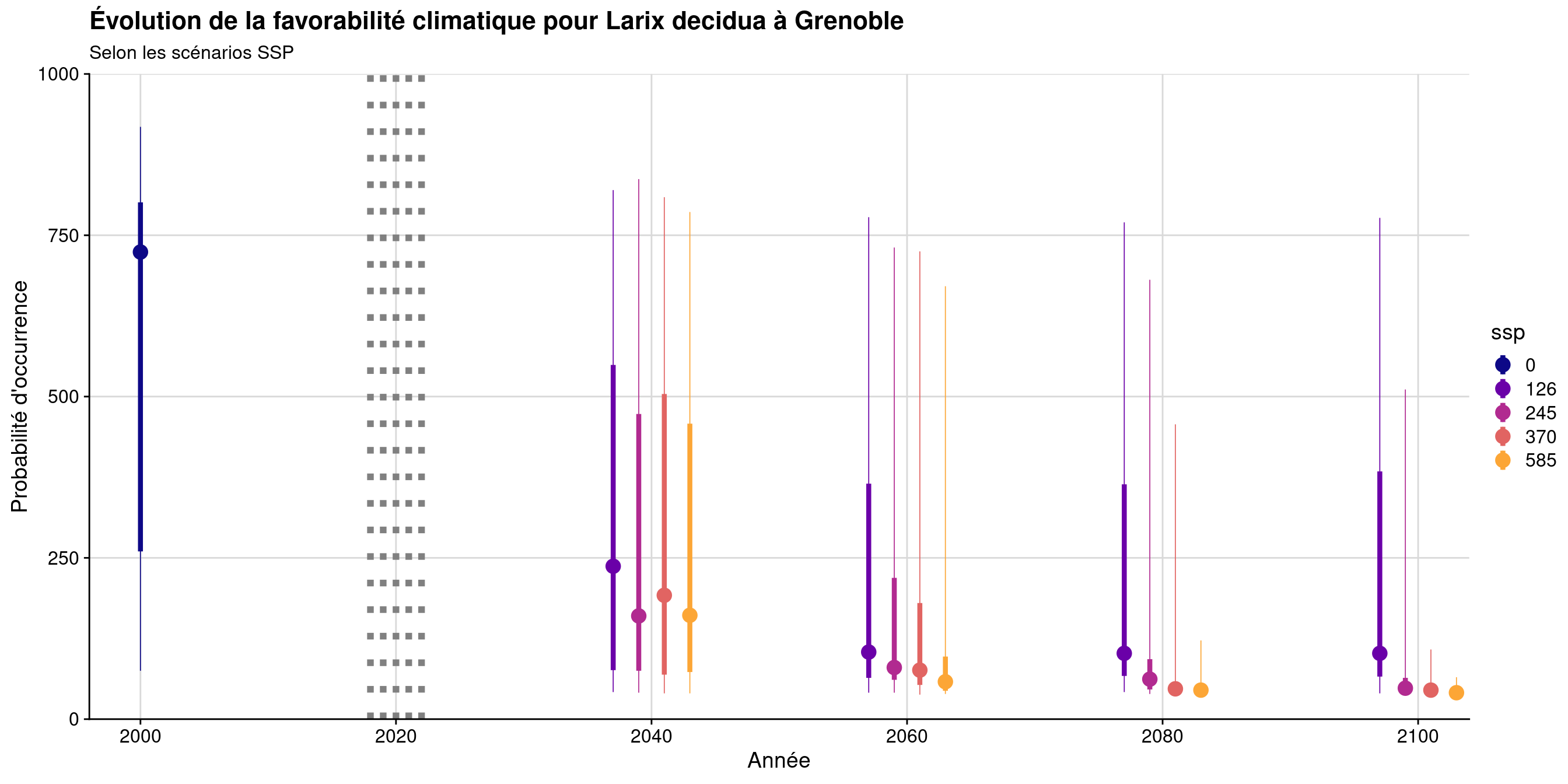
Figure 4.30: Évolution de la favorabilité climatique au cours du XXIème siècle. Le point représente la médiane sur l’étendue de la métropole (i.e. tous les pixels de la carte) ; le trait gras représente 50 % des données intermédiaires (espace inter-quartile) ; le trait fin représente le reste des données du minimum au maximum.