1 Collecte des données
Cette phase concerne toutes les données du projet qui ne sont pas les occurrences des espèces d’arbre, i.e. données « espèces » génériques, données géographiques et données climatiques. La plupart sont téléchargées sur des plateformes de données publiques.
Il est attendu un maximum de 50 GB de données environnementales téléchargées , dont les plus importantes sont :
- ~30–40 GB de données climatiques (250–300 MB × 127 TIFF) :
- variables bioclimatiques contemporaines (×19)
- variables bioclimatiques futures × 4 périodes × 3 GCMS × 4 scénarios = 48 TIFF
- la même chose pour les températures maximales (×12 + 16 × 3 GCMs)
- ~2 GB de données d’occurrences (10–20 MB × 200 espèces)
1.1 Données « espèces » génériques
1.1.1 Taxonomie : Liste d’espèces (essences) et résolution des noms d’espèces
50 essences prioritaires :
La résolution des noms d’espèces est un problème compliqué. On s’appuiera sur
l’ossature de taxonomie du GBIF (GBIF Backbone
Taxonomy),
qui propose une classification unique pour intégrer les données de plusieurs
sources de manière cohérente. L’interaction entre le GBIF et R se fait par le
package rgbif (on utilise également les outils de manipulation de données
présents dans les packages dplyr et tibble).
On peut récupérer des clés taxonomiques GBIF à partir d’un nom scientifique et vérifier la meilleure correspondance renvoyée par la fonction. On fait le test sur le premier nom de la liste fournie, qui est un nom correctement rempli :
# A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Zelkova serrata (Thunb.) Makino ACCEPTED FALSE Zelkova serrata 2984532 99 EXACT On vérifie le statut ($status) qui peut prendre les valeurs suivantes :
ACCEPTED: nom scientifique accepté ;DOUBTFUL: en pratique accepté, mais il subsiste des doutes sur l’exactitude de l’évaluation ;SYNONYM: Un synonyme dont le type exact est inconnu ;*_SYNONYM/MISAPPLIED: des sous-classes spécifiques deSYNONYM.
En cas de synonyme, on travaillera sur le nom scientifique accepté. Plus
précisément, la colonne $synonym (TRUE/FALSE) permet de tester si le nom
proposé est un synonyme ; la colonne $species renvoie directement le nom
scientifique accepté et la colonne $speciesKey le numéro de clé
correspondant (sur lequel on travaillera).
Finalement, $confidence et $matchType renseignent la correspondance trouvée
avec la taxonomie GBIF. (à noter que name_backbone() renvoie aussi toute
l’information sur les différents niveaux taxonomiques plus élevés).
Pour cet exemple, rien à signaler donc, c’est directement le nom scientifique accepté. On peut faire quelques tests sur la taxonomie (un nom hybride, un synonyme, un nom inconnu, des coquilles) :
# A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Platanus ×acerifolia (Aiton) Willd. SYNONYM TRUE Platanus hispanica 7400250 98 EXACT # A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Morus kagayamae Koidz. SYNONYM TRUE Morus australis 7262450 98 EXACT # A tibble: 1 × 5
confidence matchType synonym verbatim_name verbatim_rank
* <int> <chr> <lgl> <chr> <chr>
1 100 NONE FALSE Cladastris lutea species # A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Pterostyrax hispidus Siebold & Zucc. ACCEPTED FALSE Pterostyrax his… 3646556 96 FUZZY # A tibble: 1 × 7
scientificName status synonym species speciesKey confidence matchType
<chr> <chr> <lgl> <chr> <int> <int> <chr>
1 Chionanthus virginicus L. ACCEPTED FALSE Chionanthus virginicus 3172303 95 FUZZY Tous les tests sont concluants, sauf évidemment celui de l’essence inconnue.
On vérifie et corrige la taxonomie sur toutes les essences choisies à l’aide de
la taxonomie GBIF. Au passage, il faut corriger manuellement "Sorbus aria" en
"Sorbus aria L" afin que celui-ci soit reconnu au niveau de l’espèce. On
regarde ensuite la correspondance (exacte ou non) puis le niveau de confiance,
et enfin le statut (par exemple « synonyme »). Pour ce dernier point, on
visualise toutes les différences après correction :
EXACT
50 Min. 1st Qu. Median Mean 3rd Qu. Max.
97 97 98 98 99 100
ACCEPTED SYNONYM
44 6 # A tibble: 6 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Sophora japonica Styphnolobium japonicum SYNONYM
2 Platanus acerifolia Platanus hispanica SYNONYM
3 Catalpa bignonoides Catalpa bignonioides SYNONYM
4 Malus tschonoskii Docyniopsis tschonoskii SYNONYM
5 Morus kagayamae Morus australis SYNONYM
6 Sorbus aria L Aria edulis SYNONYMOn ajoute un nom de code abrégé qui reprend les 2 premières lettres du genre et les 2 premières lettres de l’espèce. On vérifie le nombre de doublon (en espérant qu’il reste à 0) :
[1] 0Finalement, on rajoute une colonne nom_commun (qui est pour l’instant une
copie du nom scientifique) qui servira plus tard pour présenter les fiches par
espèce, ainsi que des colonnes pour l’instant vide qui seront remplies au fur et
à mesure du traitement de chaque espèce :
$n_gbif: nombre total d’occurrences dans le GBIF ;$area: région d’endémisme (EUR, AMN, ASI)$n_area: nombre d’occurrences dans la région d’endémisme ;$n_libr: nombre d’occurrences libres d’utilisation dans la région d’endémisme (= utilisables) ;$ech: est-ce que le sous-échantillonnage a été effectué (TRUE/FALSE, ≥ 25k occurrences utilisables) ;$n_fin: nombre final d’occurrences utilisées (après potentiel sous-échantillonnage) ;$mod_ok: performance des modèles individuels suffisante (TRUE), limitée (FALSE) ou insuffisante (NA) ;$mod_n: nombre de modèles qui ont fonctionné, utilisés pour le modèle d’ensemble (soit TSS ≥ 0.8, soit TSS ≥ 0.75 si$mod_ok == FALSE) ;$mod_tss: TSS du modèle d’ensemble final ;$mod_cont_avg,$mod_cont_min,$mod_cont_max: valeurs projetées moyennes, minimales et maximales sur la métropole de Grenoble selon les conditions climatiques contemporaines ;$mod_s2_avg,$mod_s2_min,$mod_s2_max: valeurs projetées moyennes, minimales et maximales sur la métropole de Grenoble à l’horizon 2100 selon le SSP2-4.5 ;$mod_s5_avg,$mod_s5_min,$mod_s5_max: valeurs projetées moyennes, minimales et maximales sur la métropole de Grenoble à l’horizon 2100 selon les SSP5-8.5.
On visualise le tableau final d’essences :
# A tibble: 50 × 28
taxon_proposé scientificName status synonym species speciesKey confidence matchType nom_abr
<chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr>
1 Zelkova serrata Zelkova serra… ACCEP… FALSE Zelkov… 2984532 99 EXACT zese
2 Pyrus calleryana Pyrus callery… ACCEP… FALSE Pyrus … 5363119 98 EXACT pyca
3 Alnus spaethii Alnus ×spaeth… ACCEP… FALSE Alnus … 4097656 99 EXACT alsp
4 Celtis australis Celtis austra… ACCEP… FALSE Celtis… 2984492 97 EXACT ceau
5 Acer campestre Acer campestr… ACCEP… FALSE Acer c… 3189863 99 EXACT acca
6 Quercus cerris Quercus cerri… ACCEP… FALSE Quercu… 2880580 97 EXACT quce
7 Prunus serrulata Prunus serrul… ACCEP… FALSE Prunus… 3022609 98 EXACT prse
8 Prunus avium Prunus avium … ACCEP… FALSE Prunus… 3020791 97 EXACT prav
9 Liquidambar styraciflua Liquidambar s… ACCEP… FALSE Liquid… 3152824 99 EXACT list
10 Carpinus betulus Carpinus betu… ACCEP… FALSE Carpin… 2875818 98 EXACT cabe
# ℹ 40 more rows
# ℹ 19 more variables: nom_commun <lgl>, n_gbif <lgl>, area <lgl>, n_area <lgl>, n_libr <lgl>,
# ech <lgl>, n_fin <lgl>, mod_ok <lgl>, mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>,
# mod_cont_min <lgl>, mod_cont_max <lgl>, mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>,
# mod_s5_avg <lgl>, mod_s5_min <lgl>, mod_s5_max <lgl>On fait la même chose pour les 4 espèces prototypes :
EXACT
4 Min. 1st Qu. Median Mean 3rd Qu. Max.
99.0 99.0 99.5 99.5 100.0 100.0
ACCEPTED
4 # A tibble: 2 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Acer pseudoplatanus L Acer pseudoplatanus ACCEPTED
2 Aesculus hippocastanum L Aesculus hippocastanum ACCEPTED[1] 0# A tibble: 4 × 28
taxon_proposé nom_commun scientificName status synonym species speciesKey confidence matchType nom_abr
<chr> <chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr>
1 Acer pseudop… érable sy… Acer pseudopl… ACCEP… FALSE Acer p… 3189870 100 EXACT acps
2 Acer monspes… érable de… Acer monspess… ACCEP… FALSE Acer m… 7262958 99 EXACT acmo
3 Larix decidua mélèze d'… Larix decidua… ACCEP… FALSE Larix … 2686212 99 EXACT lade
4 Aesculus hip… marronnie… Aesculus hipp… ACCEP… FALSE Aescul… 3189815 100 EXACT aehi
# ℹ 18 more variables: n_gbif <lgl>, area <lgl>, n_area <lgl>, n_libr <lgl>, ech <lgl>, n_fin <lgl>,
# mod_ok <lgl>, mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>, mod_cont_min <lgl>,
# mod_cont_max <lgl>, mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>, mod_s5_avg <lgl>,
# mod_s5_min <lgl>, mod_s5_max <lgl>On complète avec le deuxième lot d’essences :
EXACT
47 Min. 1st Qu. Median Mean 3rd Qu. Max.
97.00000 97.00000 99.00000 98.17021 99.00000 99.00000
ACCEPTED DOUBTFUL SYNONYM
45 1 1 # A tibble: 2 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Prunus X schmittii Prunus schmittii DOUBTFUL
2 Malus floribunda Sinomalus sieboldii SYNONYM [1] 17# A tibble: 47 × 27
taxon_proposé scientificName status synonym species speciesKey confidence matchType nom_abr n_gbif
<chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr> <lgl>
1 Acer buergerian… Acer buergeri… ACCEP… FALSE Acer b… 7100404 99 EXACT acbu NA
2 Celtis julianae Celtis julian… ACCEP… FALSE Celtis… 4159571 99 EXACT ceju NA
3 Betula pendula Betula pendul… ACCEP… FALSE Betula… 5331916 99 EXACT bepe NA
4 Ailanthus altis… Ailanthus alt… ACCEP… FALSE Ailant… 3190653 99 EXACT aial NA
5 Albizia julibri… Albizia julib… ACCEP… FALSE Albizi… 2972983 97 EXACT alju NA
6 Cladrastis kent… Cladrastis ke… ACCEP… FALSE Cladra… 2945513 97 EXACT clke NA
7 Pteroceltis tat… Pteroceltis t… ACCEP… FALSE Pteroc… 4159853 99 EXACT ptta NA
8 Pistacia chinen… Pistacia chin… ACCEP… FALSE Pistac… 3190586 99 EXACT pich NA
9 Salix viminalis Salix viminal… ACCEP… FALSE Salix … 5372933 97 EXACT savi NA
10 Pinus sylvestris Pinus sylvest… ACCEP… FALSE Pinus … 5285637 98 EXACT pisy NA
# ℹ 37 more rows
# ℹ 17 more variables: area <lgl>, n_area <lgl>, n_libr <lgl>, ech <lgl>, n_fin <lgl>, mod_ok <lgl>,
# mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>, mod_cont_min <lgl>, mod_cont_max <lgl>,
# mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>, mod_s5_avg <lgl>, mod_s5_min <lgl>,
# mod_s5_max <lgl>On complète avec le troisième lot d’essences :
EXACT
85 Min. 1st Qu. Median Mean 3rd Qu. Max.
97.0 97.0 99.0 98.2 99.0 100.0
ACCEPTED SYNONYM
77 8 # A tibble: 8 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Cedrela sinensis Toona sinensis SYNONYM
2 Malus trilobata Eriolobus trilobatus SYNONYM
3 Pinus griffithii McClell Pinus wallichiana SYNONYM
4 Prunus amygdalus Prunus dulcis SYNONYM
5 Pterocarya fraxinifolia Pterocarya pterocarpa SYNONYM
6 Sorbus domestica Cormus domestica SYNONYM
7 Sorbus intermedia Scandosorbus intermedia SYNONYM
8 Sorbus torminalis Torminalis glaberrima SYNONYM[1] 17# A tibble: 85 × 27
taxon_proposé scientificName status synonym species speciesKey confidence matchType nom_abr n_gbif
<chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr> <lgl>
1 Abies alba Abies alba Mi… ACCEP… FALSE Abies … 2685484 97 EXACT abal NA
2 Abies koreana Abies koreana… ACCEP… FALSE Abies … 2685681 99 EXACT abko NA
3 Abies pinsapo Abies pinsapo… ACCEP… FALSE Abies … 2685464 99 EXACT abpi NA
4 Acer capillipes Acer capillip… ACCEP… FALSE Acer c… 7495204 97 EXACT accl NA
5 Acer griseum Acer griseum … ACCEP… FALSE Acer g… 3768452 99 EXACT acgr NA
6 Acer heldreichii Acer heldreic… ACCEP… FALSE Acer h… 7375236 98 EXACT ache NA
7 Acer negundo Acer negundo … ACCEP… FALSE Acer n… 3189866 99 EXACT acne NA
8 Acer opalus Acer opalus M… ACCEP… FALSE Acer o… 7100278 99 EXACT acop NA
9 Alniaria alnifo… Alniaria alni… ACCEP… FALSE Alniar… 10825869 99 EXACT alal NA
10 Alnus incana Alnus incana … ACCEP… FALSE Alnus … 9148577 97 EXACT alin NA
# ℹ 75 more rows
# ℹ 17 more variables: area <lgl>, n_area <lgl>, n_libr <lgl>, ech <lgl>, n_fin <lgl>, mod_ok <lgl>,
# mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>, mod_cont_min <lgl>, mod_cont_max <lgl>,
# mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>, mod_s5_avg <lgl>, mod_s5_min <lgl>,
# mod_s5_max <lgl>On complète avec le quatrième lot d’essences :
EXACT FUZZY
46 3 Min. 1st Qu. Median Mean 3rd Qu. Max.
94.00000 97.00000 98.00000 97.93878 99.00000 99.00000
ACCEPTED SYNONYM
47 2 # A tibble: 5 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Celtis caucasica Celtis australis SYNONYM
2 Eriobotrya deflexa Rhaphiolepis deflexa SYNONYM
3 Koelreuteria bippinata Koelreuteria bipinnata ACCEPTED
4 Ostrya virgniana Ostrya virginiana ACCEPTED
5 Pterocarya stepnoptera Pterocarya stenoptera ACCEPTED taxon_proposé scientificName status synonym species speciesKey
1 Celtis caucasica Celtis caucasica Willd. SYNONYM TRUE Celtis australis 2984492
2 Pinus pinaster Pinus pinaster Aiton ACCEPTED FALSE Pinus pinaster 5285565
3 Quercus canariensis Quercus canariensis Willd. ACCEPTED FALSE Quercus canariensis 2880808
4 Quercus faginea Quercus faginea Lam. ACCEPTED FALSE Quercus faginea 2881480
5 Quercus ithaburensis Quercus ithaburensis Decne. ACCEPTED FALSE Quercus ithaburensis 2878323
6 Quercus rotundifolia Quercus rotundifolia Lam. ACCEPTED FALSE Quercus rotundifolia 2881550
confidence matchType nom_abr
1 97 EXACT ceau
2 98 EXACT pipi
3 99 EXACT quca
4 97 EXACT qufa
5 99 EXACT quit
6 99 EXACT quro[1] 0# A tibble: 48 × 27
taxon_proposé scientificName status synonym species speciesKey confidence matchType nom_abr n_gbif
<chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr> <lgl>
1 Abies concolor Abies concolo… ACCEP… FALSE Abies … 2685580 98 EXACT abco NA
2 Acer hyrcanum Acer hyrcanum… ACCEP… FALSE Acer h… 7262979 99 EXACT achy NA
3 Arbutus andrach… Arbutus andra… ACCEP… FALSE Arbutu… 4170901 99 EXACT aran NA
4 Arbutus unedo Arbutus unedo… ACCEP… FALSE Arbutu… 2882803 99 EXACT arun NA
5 Banksia integri… Banksia integ… ACCEP… FALSE Banksi… 7124338 97 EXACT bain NA
6 Carpinus orient… Carpinus orie… ACCEP… FALSE Carpin… 2875780 99 EXACT caor NA
7 Carpinus turcza… Carpinus turc… ACCEP… FALSE Carpin… 2875945 99 EXACT catu NA
8 Carya illinoine… Carya illinoi… ACCEP… FALSE Carya … 3054289 99 EXACT cail NA
9 Casuarina cunni… Casuarina cun… ACCEP… FALSE Casuar… 2891932 99 EXACT cacu NA
10 Cercidiphyllum … Cercidiphyllu… ACCEP… FALSE Cercid… 8060423 98 EXACT ceja NA
# ℹ 38 more rows
# ℹ 17 more variables: area <lgl>, n_area <lgl>, n_libr <lgl>, ech <lgl>, n_fin <lgl>, mod_ok <lgl>,
# mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>, mod_cont_min <lgl>, mod_cont_max <lgl>,
# mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>, mod_s5_avg <lgl>, mod_s5_min <lgl>,
# mod_s5_max <lgl>Puis le cinquième (et dernier) lot d’essences :
EXACT
71 Min. 1st Qu. Median Mean 3rd Qu. Max.
83.00000 97.00000 98.00000 97.78873 99.00000 99.00000
ACCEPTED DOUBTFUL SYNONYM
63 1 7 # A tibble: 8 × 3
taxon_proposé species status
<chr> <chr> <chr>
1 Carya tomentosa Carya alba SYNONYM
2 Halesia monticola Halesia tetraptera SYNONYM
3 Maackia chinensis Maackia hupehensis SYNONYM
4 Eriobotrya deflexa Rhaphiolepis deflexa SYNONYM
5 Sapindus saponaria l. Sapindus saponaria ACCEPTED
6 Senegalia visco Parasenegalia visco SYNONYM
7 Abies bornmuelleriana Abies nordmanniana SYNONYM
8 Acer cappadocicum subsp. lobelii Acer lobelii SYNONYM [1] 6[1] 0# A tibble: 68 × 27
taxon_proposé scientificName status synonym species speciesKey confidence matchType nom_abr n_gbif
<chr> <chr> <chr> <lgl> <chr> <int> <int> <chr> <chr> <lgl>
1 Abies nordmanni… Abies nordman… ACCEP… FALSE Abies … 2685552 99 EXACT abno NA
2 Abies numidica Abies numidic… ACCEP… FALSE Abies … 2685546 97 EXACT abnu NA
3 Aesculus flava Aesculus flav… ACCEP… FALSE Aescul… 3189824 97 EXACT aefl NA
4 Aesculus indica Aesculus indi… ACCEP… FALSE Aescul… 8566446 97 EXACT aein NA
5 Carya cordiform… Carya cordifo… ACCEP… FALSE Carya … 3054299 98 EXACT caco NA
6 Carya tomentosa Carya tomento… SYNON… TRUE Carya … 3054302 98 EXACT caal NA
7 Catalpa speciosa Catalpa speci… ACCEP… FALSE Catalp… 3172612 97 EXACT casp NA
8 Celtis bungeana Celtis bungea… ACCEP… FALSE Celtis… 7141664 99 EXACT cebu NA
9 Cercis canadens… Cercis canade… ACCEP… FALSE Cercis… 5353583 98 EXACT ceca NA
10 Corylus colurna Corylus colur… ACCEP… FALSE Corylu… 2875968 99 EXACT cocl NA
# ℹ 58 more rows
# ℹ 17 more variables: area <lgl>, n_area <lgl>, n_libr <lgl>, ech <lgl>, n_fin <lgl>, mod_ok <lgl>,
# mod_n <lgl>, mod_tss <lgl>, mod_cont_avg <lgl>, mod_cont_min <lgl>, mod_cont_max <lgl>,
# mod_s2_avg <lgl>, mod_s2_min <lgl>, mod_s2_max <lgl>, mod_s5_avg <lgl>, mod_s5_min <lgl>,
# mod_s5_max <lgl>1.1.2 Carte de référence des espèces en France (IFN)
Pour valider les données de chaque espèce, on comparera avec la distribution française issue de l’Inventaire Forestier National, carte préparée par Delphine Legron. Les colonnes avec des NAs sont importées en tant que caractères, pas en tant que nombres. On corrige donc cela.
Reading layer `SER_EssenceV1' from data source
`/home/mathieu/Travail/Arbres Grenoble/Data/SER_Placette/SER_EssenceV1.shp' using driver `ESRI Shapefile'
Simple feature collection with 85 features and 159 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 99038.3984 ymin: 6046555.789 xmax: 1242435.544 ymax: 7110523.769
Projected CRS: RGF93 v1 / Lambert-93Simple feature collection with 10 features and 12 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: 99038.3984 ymin: 6150166.078 xmax: 880525.8197 ymax: 7110523.769
Projected CRS: RGF93 v1 / Lambert-93
codeser NomSER CODE_SER Surface_FR Surface_AU SURFACE_TO Taux_Boise
1 A11 Ouest-Bretagne et Nord-Cotentin A11 221 1230 1451 0.15230875258
2 A12 Pays de Saint-Malo A12 80 496 576 0.13888888889
3 A13 Bocage normand et Pays de Fougères A13 139 1171 1310 0.10610687023
4 A21 Bretagne méridionale A21 100 427 527 0.18975332068
5 A22 Bocage armoricain A22 93 969 1062 0.08757062147
6 A30 Bocage vendéen A30 82 949 1031 0.07953443259
7 B10 Côtes et plateaux de la Manche B10 204 1419 1623 0.12569316081
8 B21 Flandres B21 32 332 364 0.08791208791
9 B22 Plaine picarde B22 43 704 747 0.05756358768
10 B23 Mosan, Thiérache et Hainaut B23 51 285 336 0.15178571429
CODE_SER_1 ailanthus Liriodendr Nb_total Nb_Placett geometry
1 A11 NA NA 5734 383 MULTIPOLYGON (((176022.0722...
2 A12 NA NA 2094 125 MULTIPOLYGON (((617421.5988...
3 A13 NA NA 2887 229 MULTIPOLYGON (((367861.8486...
4 A21 NA NA 2022 154 MULTIPOLYGON (((232292.7348...
5 A22 NA NA 2309 179 MULTIPOLYGON (((365978.3768...
6 A30 NA NA 2059 166 MULTIPOLYGON (((404545.8662...
7 B10 NA NA 4433 385 MULTIPOLYGON (((599795.0914...
8 B21 NA NA 798 50 MULTIPOLYGON (((719475.0944...
9 B22 NA NA 1082 91 MULTIPOLYGON (((705413.3238...
10 B23 NA NA 1127 84 MULTIPOLYGON (((747176.4614...Le jeu de données comporte les informations de surface (Surface_FR = surface
de forêt ; Surface_AU = surface autre ; Surface_TO = surface totale), le
taux boisé, le nombre d’observation (par espèce et total) et le nombre de
placettes pour 85 sylvo-écorégions sur le territoire français. Chaque essence
est listée dans une colonne dédiée avec le nom scientifique. Problème, le format
Shapefile tronque les noms de colonnes à 10 caractères ; on croise donc ce
tableau avec la liste préparée en amont par Margot Ahr. On importe uniquement
l’information pertinente, dans les lignes 4 (nom usuel) et 5 (nom
scientifique). Lorsque le nom scientifique est absent, on utilise le nom usuel ;
on rajoute également une majuscule au début :
On termine par cartographier le nombre de placette par écorégion, à l’aide du
package ggplot2 (on charge aussi cowplot pour les thèmes et viridis pour
les palettes de couleurs) :
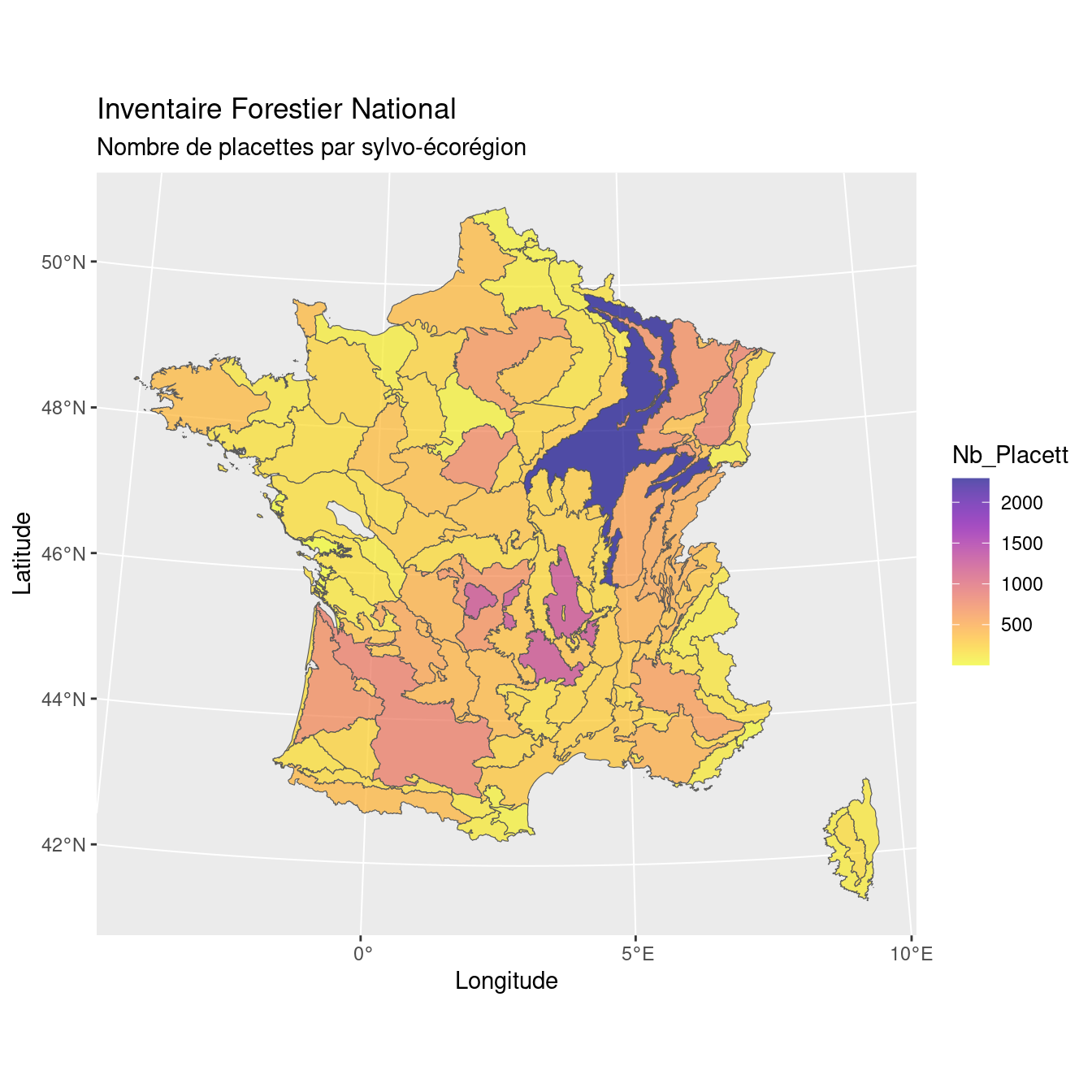
Figure 1.1: Nombre de placettes par sylvo-écorégion de l’Inventaire Forestier National.
On peut y voir une grosse variabilité du nombre de placettes selon l’écorégion. Voyons ce que ça donne pour l’érable de Montpellier (densité par écorégion) :

Figure 1.2: Densité d’observations de l’érable de Montpellier par sylvo-écorégion de l’Inventaire Forestier National.
1.2 Données géographiques
1.2.1 Fond de carte mondiale et masques régionaux
On prépare une carte du monde en résolution moyenne avec le package
rnaturalearth (et les données dans rnaturalearthdata) :
On créé au passage trois masques (rectangulaires) en WGS 84 (EPSG : 4326) qui nous serviront plus tard pour évaluer l’endémisme des essences, et pour sous-échantillonner les données d’occurrences et de climat :
- masque Europe : longitude 12°W–47°E, latitude 29–71°N
- masque Amérique du Nord : longitude 50–130°W, latitude 13–55°N
- masque Asie : longitude 68–149°W, latitude 3–57°N
On visualise les 3 masques sur une carte du monde (note : les côtés verticaux des rectangles sont ajustés à la projection de Robinson pour suivre les méridiens de manière courbe) :
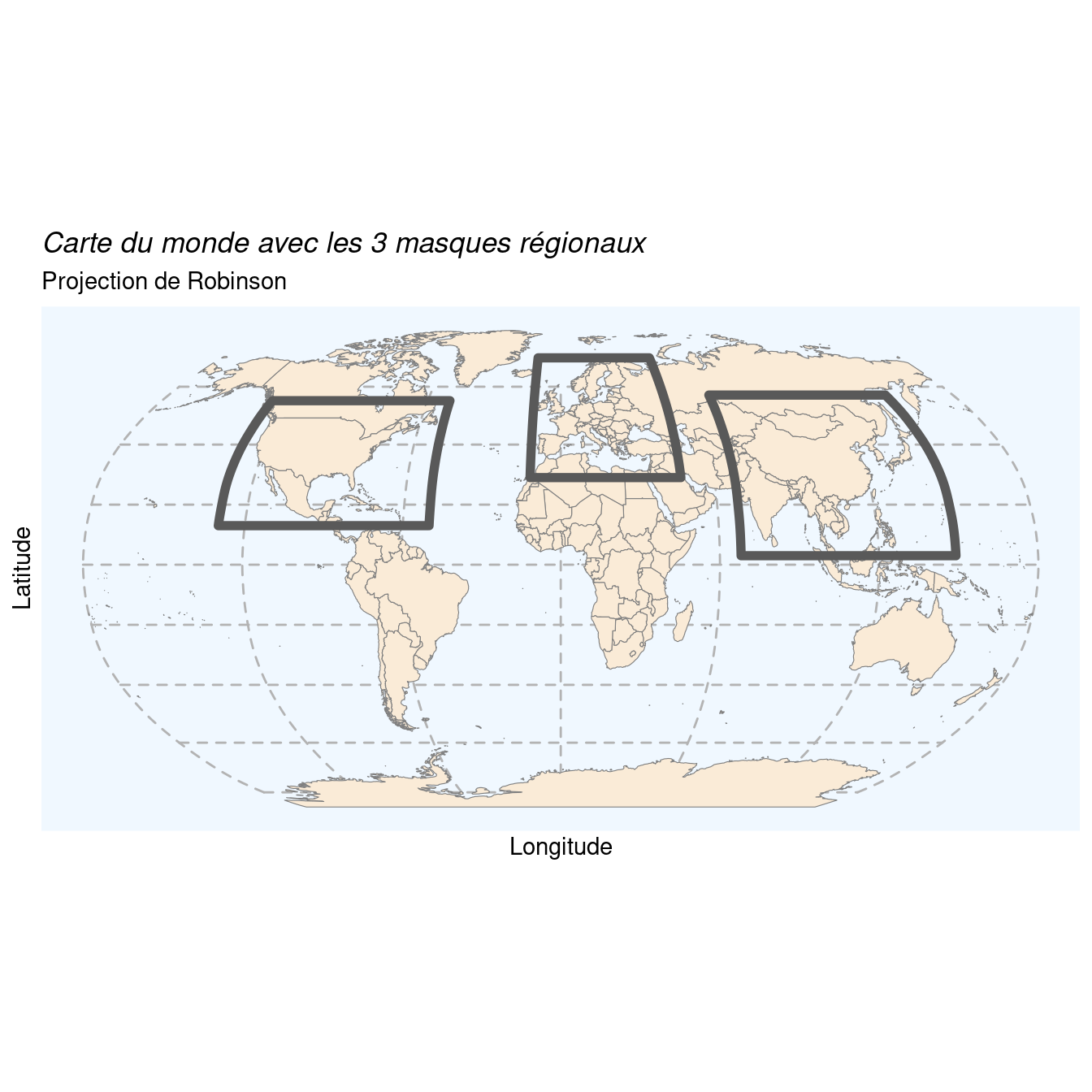
Figure 1.3: Carte du monde avec les 3 masques régionaux
Pour les cartes régionales, on utilisera les projections de référence suivantes :
- Europe : ETRS89 Lambert Azimuthal Equal Area (EPSG : 3035), le système de référence pour les cartes à l’échelle européenne ;
- Amérique du Nord : NAD27 US National Atlas Equal Area (EPSG : 2163), le système de référence pour les États-Unis ;
- Asie : China Geodetic Coordinate System 2000 (EPSG : 4480), le système de référence pour la Chine.
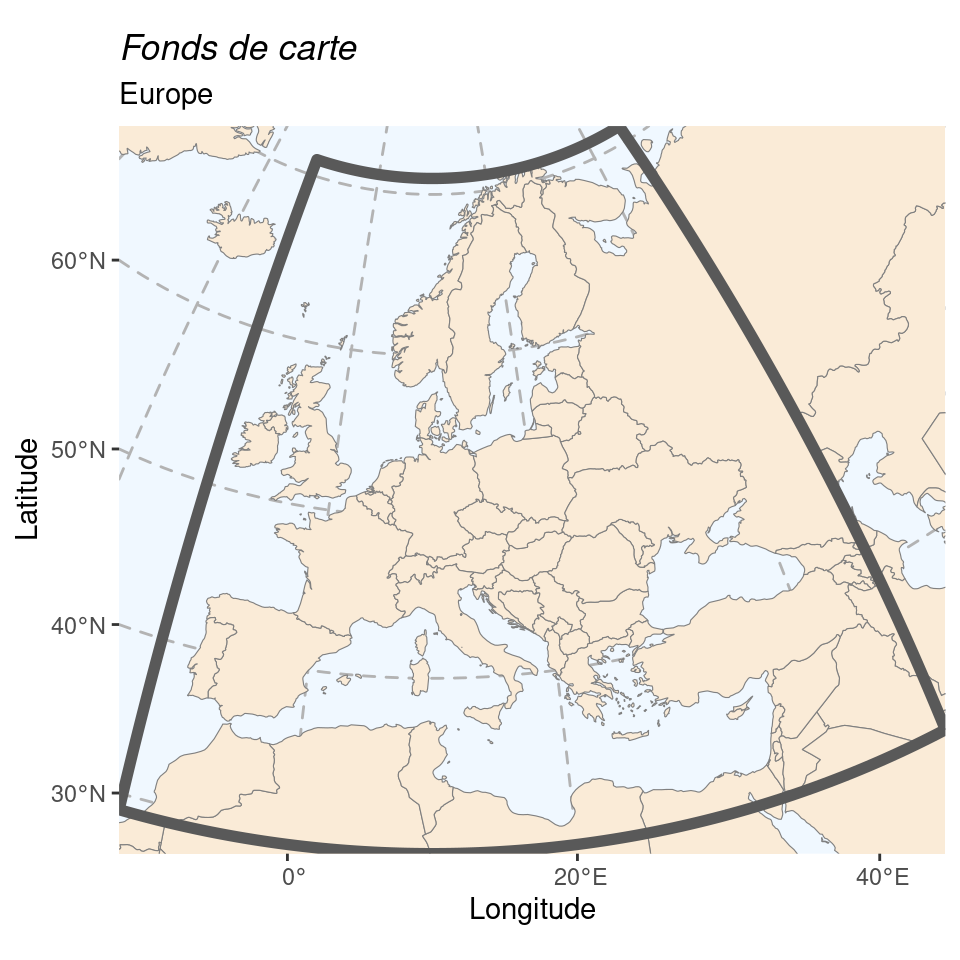
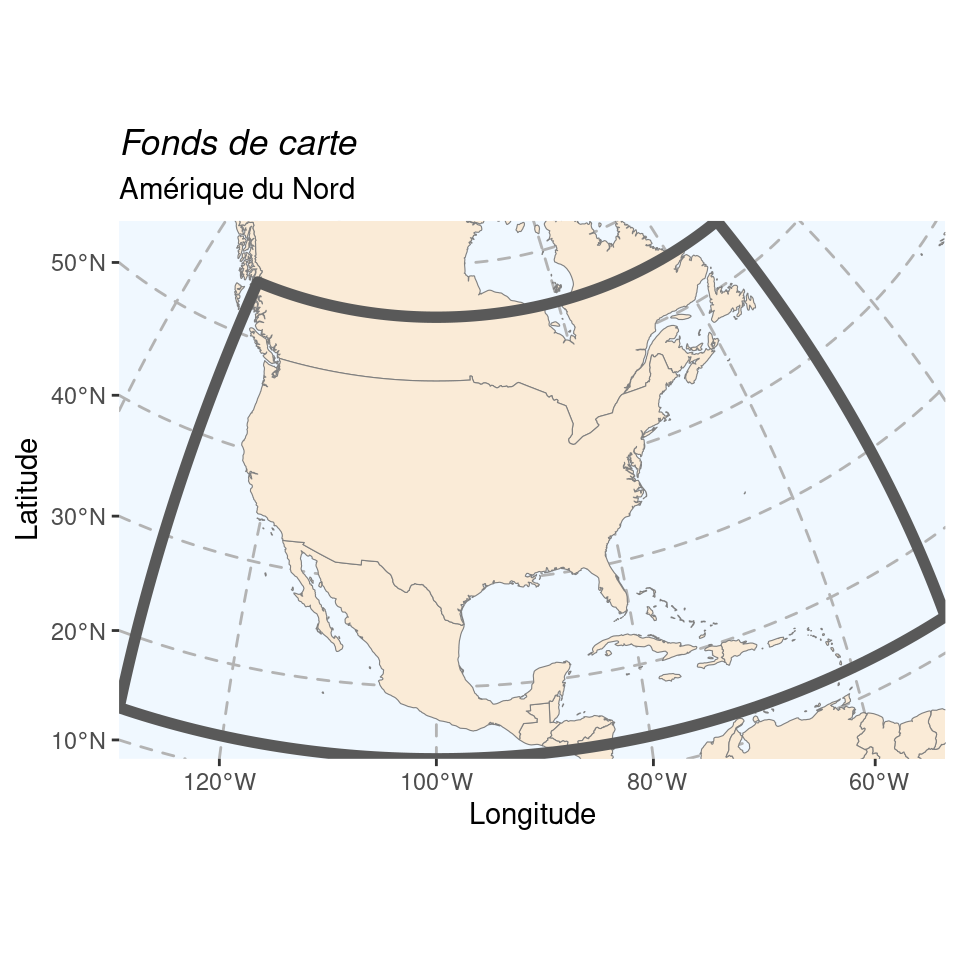
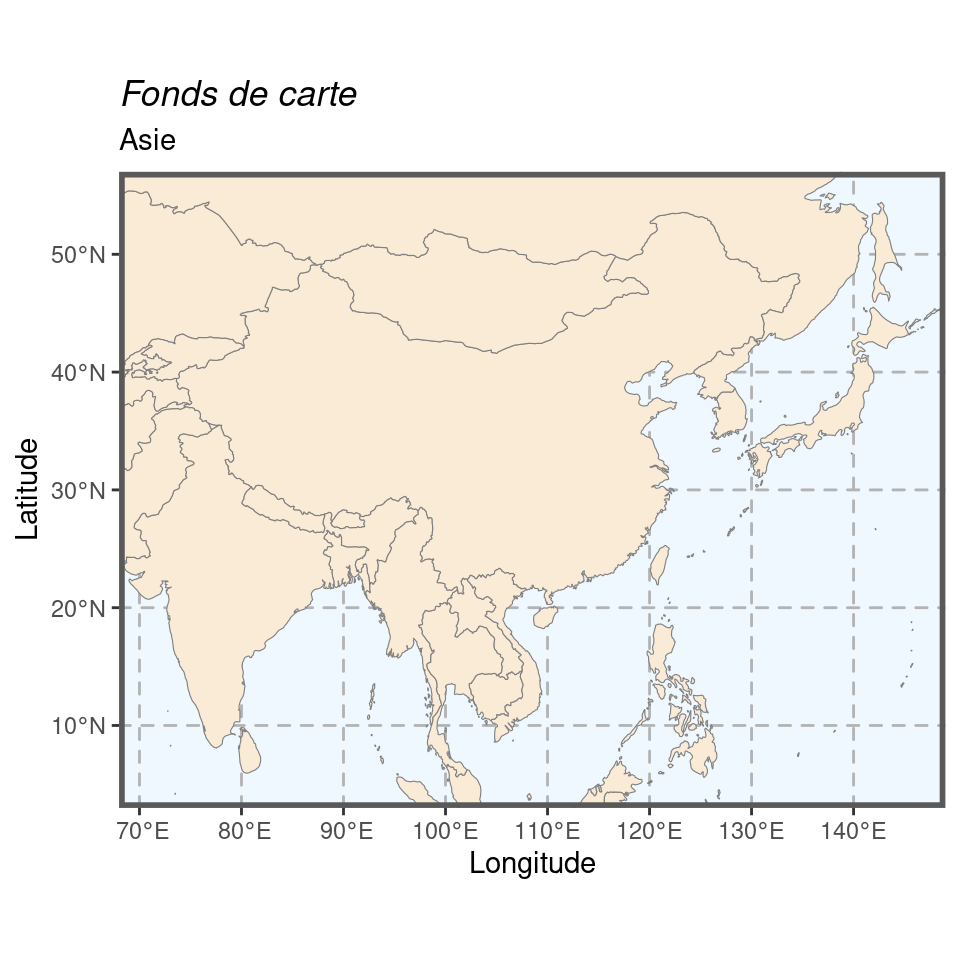
Figure 1.4: Fonds de cartes pour les 3 cartes régionales.
1.2.2 Données climatiques contemporaines
Pour toutes les données climatiques, nous nous appuierons sur les données fournies par WorldClim (version 2.1), un jeu de données global au format raster pour la période contemporaine (1970–2000) et pour des projections futures sur des périodes de 20 ans (2021-2040, 2041-2060, 2061-2080, 2081-2100). WorldClim interpole les données ponctuelles de stations météo pour offrir une couverture globale jusqu’à une résolution spatiale de 30 secondes (environ 1 km²) pour les données contemporaines, mais seulement 2,5 minutes (environ 20 km² par pixel) pour les données futures. Pour des raisons de cohérence et de comparabilité, nous utiliserons donc la résolution maximale possible pour les deux jeux de données, à savoir 2,5 minutes.
Les deux jeux de données présentent les températures (valeurs mensuelles moyennes, minimales et maximales) et la précipitation (valeurs mensuelles), ainsi que 19 variables bioclimatiques dérivées représentant des tendances annuelles, la saisonnalité, ou les conditions extrêmes ou limitantes :
- BIO1 = Température annuelle moyenne [“temp_year”]
- BIO2 = Amplitude de température diurne moyenne (moyenne des valeurs mensuelles (temp max - temp min)) [“temp_diurnal_range”]
- BIO3 = Isothermalité (BIO2/BIO7 ×100) [“temp_iso”]
- BIO4 = Saisonnalité de température (écart type ×100) [“temp_season”]
- BIO5 = Température maximale du mois le plus chaud [“temp_max”]
- BIO6 = Température minimale du mois le plus froid [“temp_min”]
- BIO7 = Amplitude annuelle de température (BIO5-BIO6) [“temp_year_range”]
- BIO8 = Température moyenne du trimestre le plus humide [“temp_wet_quart”]
- BIO9 = Température moyenne du trimestre le plus sec [“temp_dry_quart”]
- BIO10 = Température moyenne du trimestre le plus chaud [“temp_mean_hot”]
- BIO11 = Température moyenne du trimestre le plus froid [“temp_mean_cold”]
- BIO12 = Précipitations annuelles [“prec_year”]
- BIO13 = Précipitations du mois le plus humide [“prec_wet_month”]
- BIO14 = Précipitations du mois le plus sec [“prec_dry_month”]
- BIO15 = Saisonnalité de précipitation (coefficient de variation) [“prec_season”]
- BIO16 = Précipitations du trimestre le plus humide [“prec_wet_quart”]
- BIO17 = Précipitations du trimestre le plus sec [“prec_dry_quart”]
- BIO18 = Précipitations du trimestre le plus chaud [“prec_hot_quart”]
- BIO19 = Précipitations du trimestre le plus froid [“prec_cold_quart”]
Les variables bioclimatiques présentent des paramètres importants pour la physiologie des plantes, notamment pour leur croissance et leur distribution, et sont de ce fait plus pertinentes que les variables de base (température et précipitation). Pour cette étude, nous utiliserons les variables bioclimatiques qui ne sont pas trop corrélées à l’échelle européenne (coefficient de corrélation de Pearson \(r < 0,7\)), ainsi que la température au mois d’août afin de pouvoir affiner ensuite avec les températures de surface de l’agglomération de Grenoble.
On commence par charger le package geodata qui sert à télécharger des données
globales, comme celles de WorldClim.
Le package geodata permet de récupérer en un seul raster de classe
SpatRaster (terra) les 19 variables bioclimatiques. On précise la
résolution, et on récupère les données mondiales de
WorldClim, qui seront
stockées dans Data-dl :
class : SpatRaster
dimensions : 4320, 8640, 19 (nrow, ncol, nlyr)
resolution : 0.04166666667, 0.04166666667 (x, y)
extent : -180, 180, -90, 90 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
sources : wc2.1_2.5m_bio_1.tif
wc2.1_2.5m_bio_2.tif
wc2.1_2.5m_bio_3.tif
... and 16 more source(s)
names : temp_year, temp_~range, temp_iso, temp_season, temp_max, temp_min, ...
min values : -54.75916672, 1.00000000, 9.063088417, 0.000000, -30.76000023, -72.50399780, ...
max values : 31.16666603, 21.97299957, 100.000000000, 2377.624023, 48.45999908, 26.45000076, ... On télécharge également la température maximale au mois d’août, que l’on ajoute au raster existant :
class : SpatRaster
dimensions : 4320, 8640, 20 (nrow, ncol, nlyr)
resolution : 0.04166666667, 0.04166666667 (x, y)
extent : -180, 180, -90, 90 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
sources : wc2.1_2.5m_tmax_08.tif
wc2.1_2.5m_bio_1.tif
wc2.1_2.5m_bio_2.tif
... and 17 more source(s)
names : temp_~ugust, temp_year, temp_~range, temp_iso, temp_season, temp_max, ...
min values : -62.09999847, -54.75916672, 1.00000000, 9.063088417, 0.000000, -30.76000023, ...
max values : 46.72399979, 31.16666603, 21.97299957, 100.000000000, 2377.624023, 48.45999908, ... On prépare le masque pour les 3 régions d’endémisme au format terra :
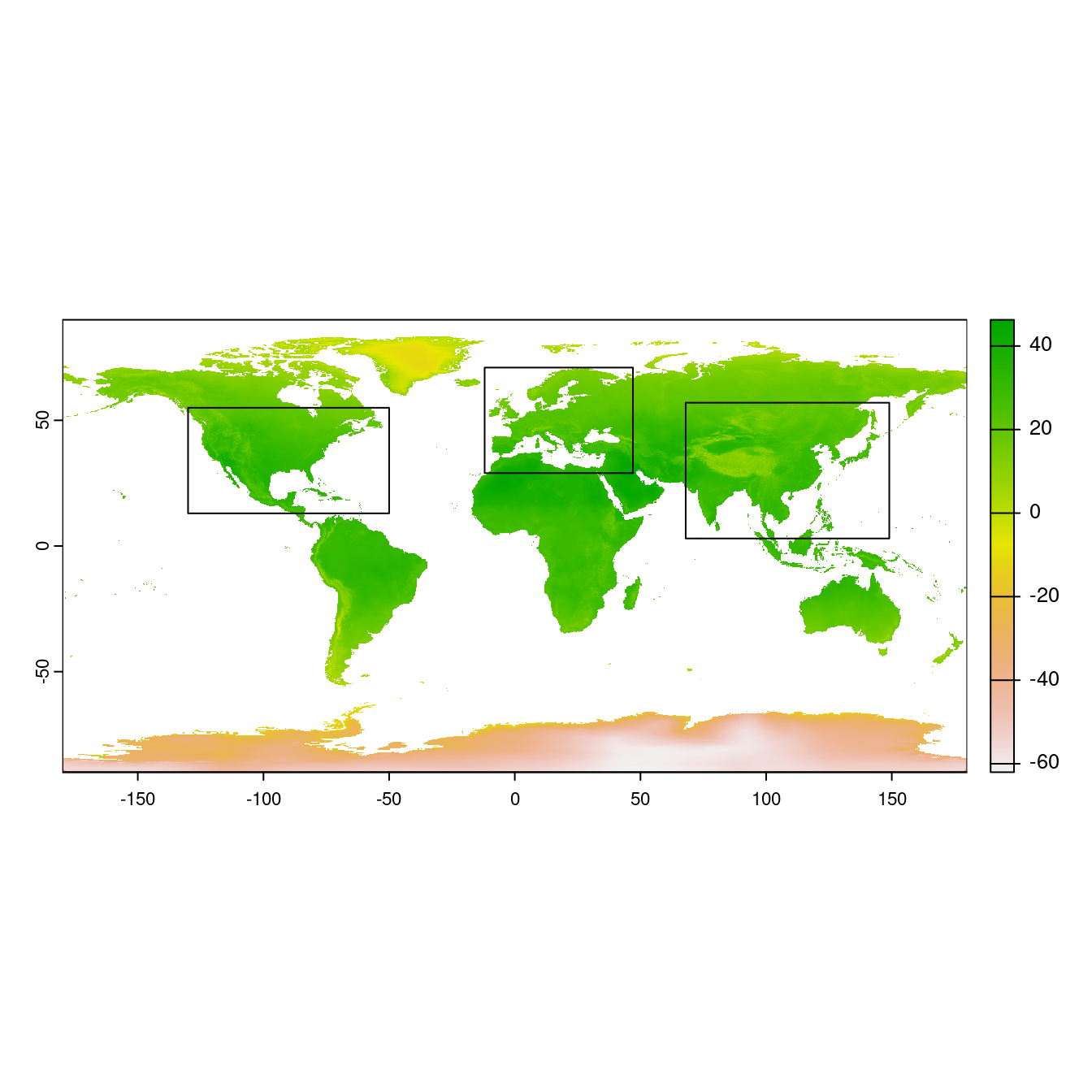
Figure 1.5: Carte du monde avec les 3 zones d’intérêt.
Puis on l’utilise pour découper les données climatiques mondiales au niveau des 3 zones :
On souhaite conserver un nombre restreint de variables peu corrélées ; on
enlèvera notamment les variables avec un coefficient de corrélation \(r > 0.75\). Pour cela, on s’appuie sur les données climatiques européennes ; on
utilisera la même sélection de données pour les deux autres régions. On examine
les structures de corrélation à l’aide d’une Analyse en composantes principales
(ACP) en utilisant le package ade4 :


Figure 1.6: A) Diagramme en barre des valeurs propres de l’ACP sur les données climatiques contemporaines. B) Cercle des corrélations des variables climatiques contemporaines.
Deux composantes principales ressortent visiblement, et représentent une large part de la variabilité climatique. Sur ces deux axes, il ressort plusieurs groupes de variables corrélées, dont un grand nombre avec des valeurs très élevées de corrélation. On cherche les variables qui sont le moins corrélées, incluant les températures maximales en août et une variable de température hivernale. On retient 6 variables (4 de température et 2 de précipitation) :
temp_max_augusttemp_mintemp_wet_quarttemp_seasonprec_wet_quartprec_season
class : SpatRaster
dimensions : 1008, 1416, 6 (nrow, ncol, nlyr)
resolution : 0.04166666667, 0.04166666667 (x, y)
extent : -12, 47, 29, 71 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
source(s) : memory
names : temp_~ugust, temp_min, temp_~quart, temp_season, prec_~quart, prec_season
min values : 0.08399999887, -24.58399963, -14.85666656, 0.00000, 3, 4.495999813
max values : 46.10400009155, 12.39999962, 26.40666580, 1395.26355, 1264, 124.089523315 Et on vérifie les corrélations :
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1.00 0.74 0.17 -0.03 -0.53 0.61
[2,] 0.74 1.00 -0.10 -0.67 -0.15 0.46
[3,] 0.17 -0.10 1.00 0.43 -0.38 -0.10
[4,] -0.03 -0.67 0.43 1.00 -0.32 -0.06
[5,] -0.53 -0.15 -0.38 -0.32 1.00 -0.27
[6,] 0.61 0.46 -0.10 -0.06 -0.27 1.00Les seules corrélations résiduelles supérieurs à 0.5 sont entre la température maximale en août et 1) la température minimum (\(r = 0.74\)), 2) les précipitations du trimestre le plus humide (\(r = -0.53\)), 3) la saisonnalité des précipitations (\(r = 0.61\)), et entre la saisonnalité des températures et la température minimum (\(r = -0.67\)). On est toutefois en-dessous du seuil établi, et les autres corrélations ne sont pas un problème.
Et on vérifie sur la carte ce que ça donne pour deux variables de température et une de précipitation :
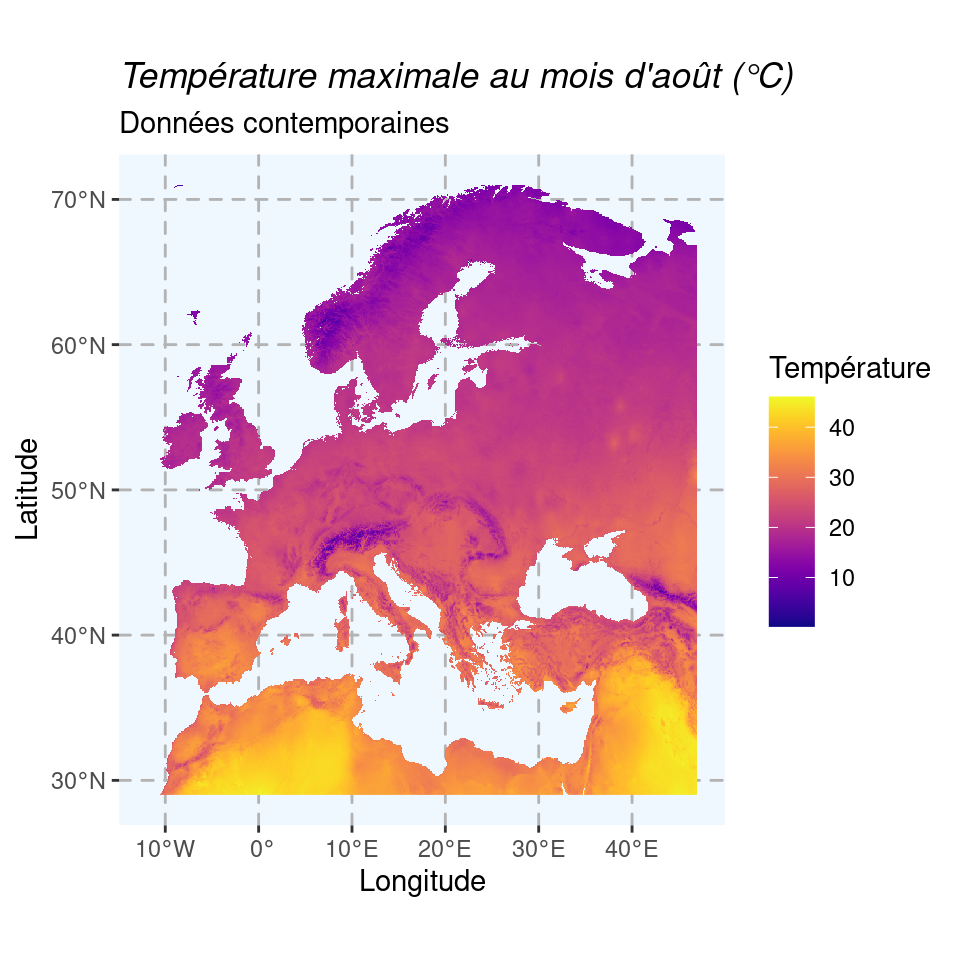
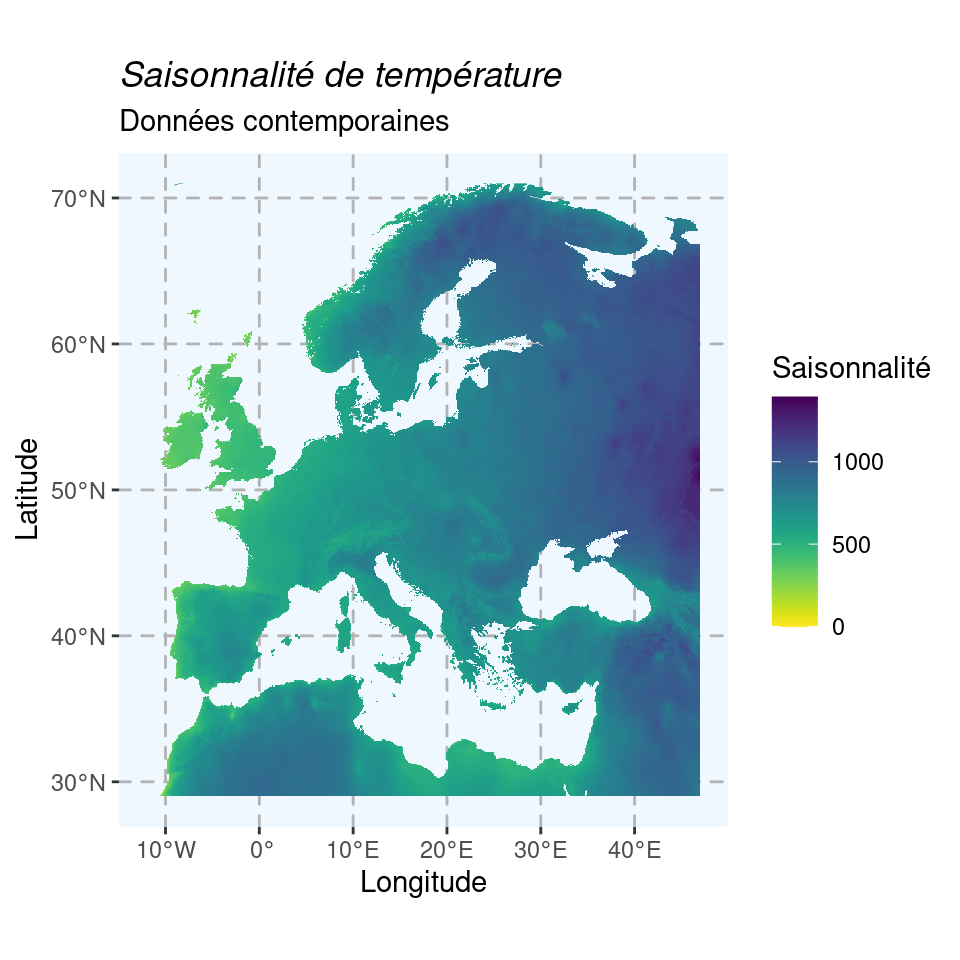
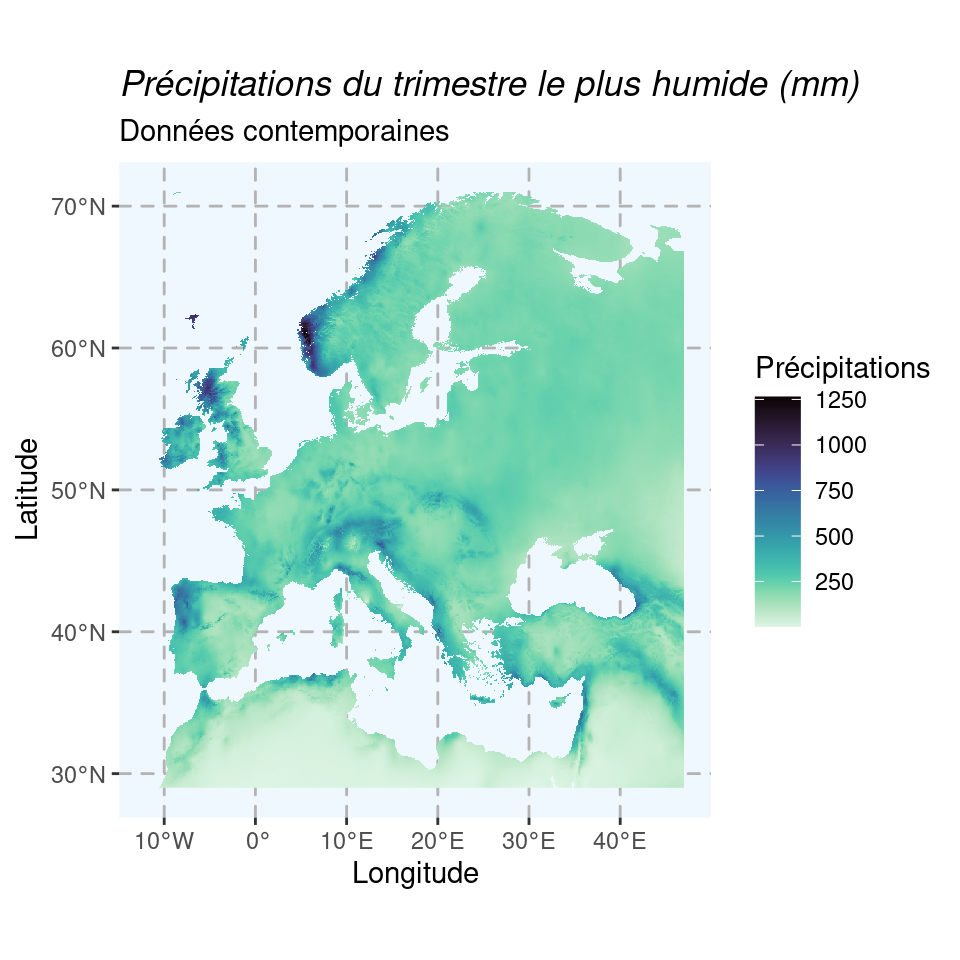
Figure 1.7: Cartes climatiques contemporaines en Europe (1970–2000).
Plus globalement, voici toutes les couches climatiques dans les 3 zones d’intérêt :
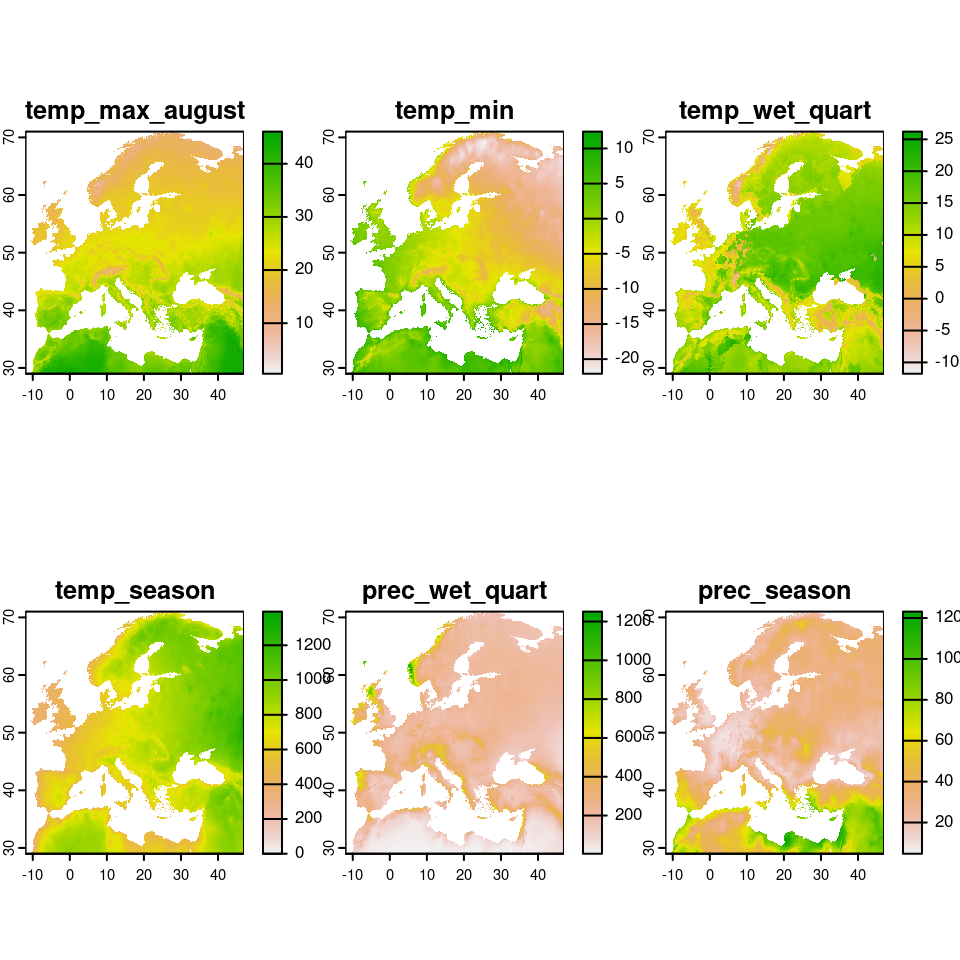
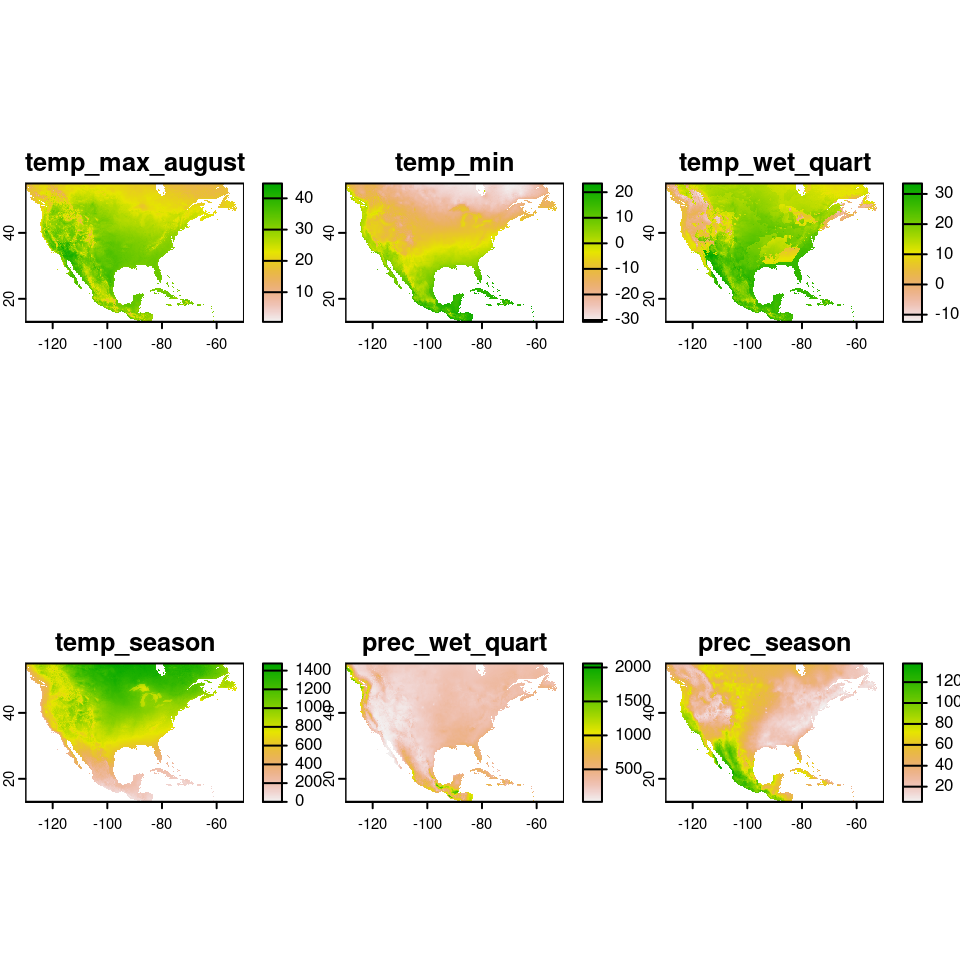
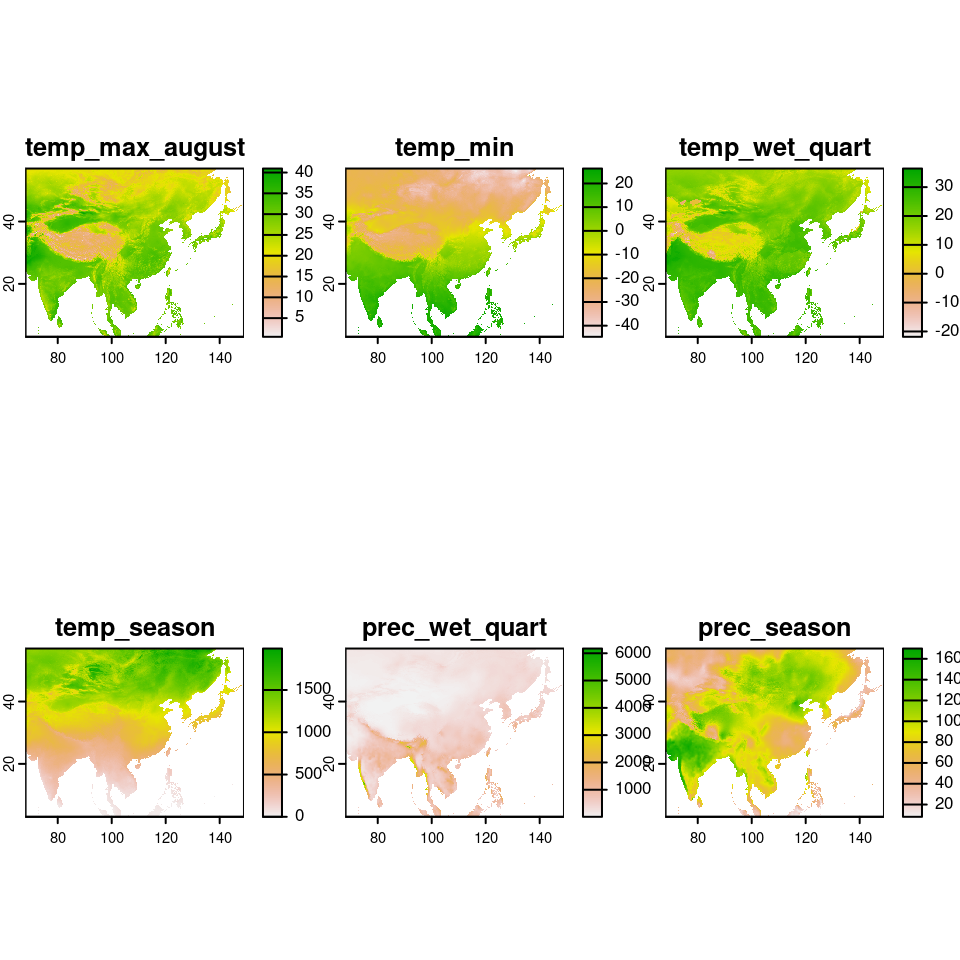
Figure 1.8: Conditions climatiques contemporaines dans les 3 régions d’endémisme données par WorldClim.
1.2.3 Données climatiques futures
Les données disponibles proviennent du Coupled Model Intercomparison Project Phase 6 (CMIP6) en lien avec le Sixième rapport d’évaluation du GIEC (IPCC AR6, 2021–2022). Les données climatiques futures ont été mises à l’échelle, corrigées pour leur biais, et couvrent les 4 scénarios retenus (trajectoires socio-économiques partagées, en anglais shared socio-economic pathways, SSPs) :
- un scénario « vert » à +1,8 °C projetés à l’horizon 2100 (SSP1-2.6) ;
- un scénario optimiste à +2,7 °C (SSP2-4.5) ;
- un scénario moyen à +3,6 °C (SSP3-7.0) ;
- un scénario du pire à +4,4 °C (SSP5-8.5).
Les conditions générales d’utilisation des modèles du CMIP6 sont disponibles en ligne. Pour résumer :
- les modèles sont sous licence Creative Commons Attribution-ShareAlike 4.0 (CC BY-SA) ;
- lien pour citer les modèles du CMIP6
1.2.3.1 GCM « IPSL-CM6A-LR »
On commence par les données issues du modèle de l’Institut Pierre-Simon Laplace
version 6A à faible résolution (IPSL-CM6A-LR), pour les 4 scénarios envisagés
et les 4 périodes de temps futures.
On télécharge également les températures maximales en août.
Puis l’on sélectionne et combine les 6 variables d’intérêt (température maximale en août, température minimale annuelle, température du trimestre le plus humide, saisonnalité de température, précipitations du trimestre le plus humide, saisonnalité de précipitation).
On utilise ensuite le masque pour l’Europe.
Carte pour les mêmes variables de température et de précipitation, pour la période 2080–2100 du scénario SSP5-8.5 et le modèle IPSL-CM6A-LR :
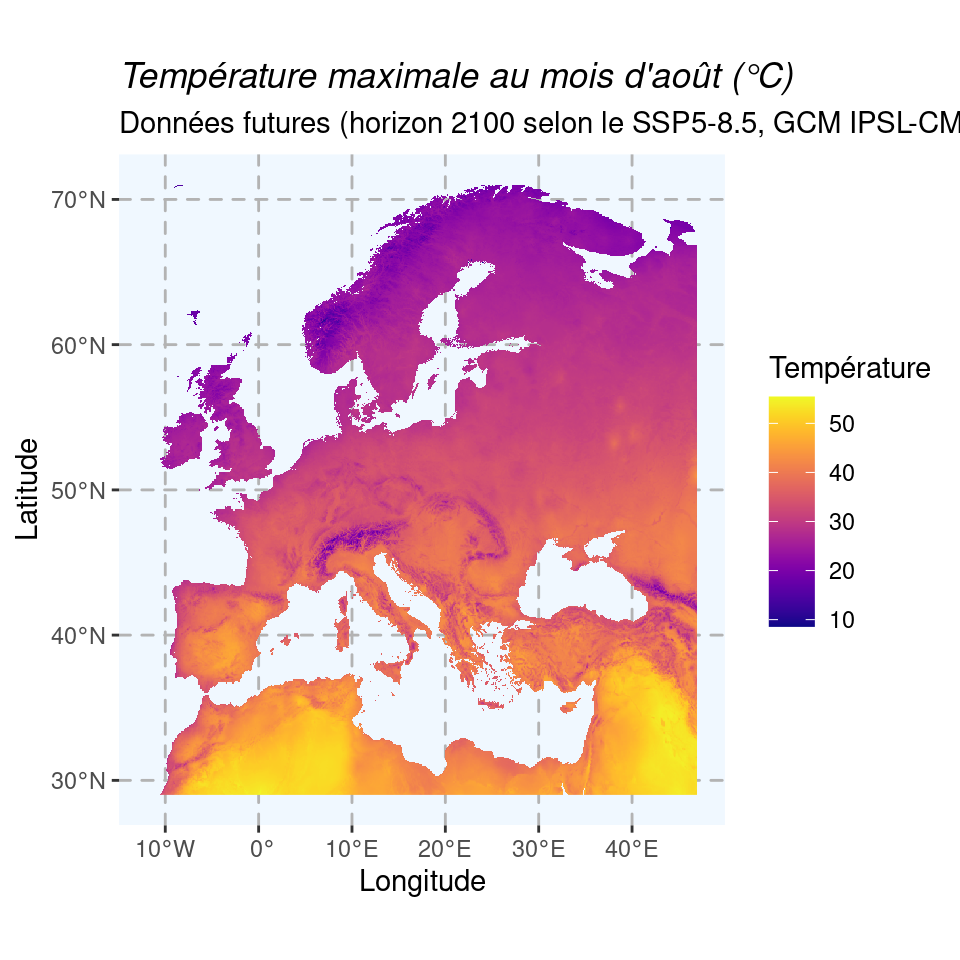
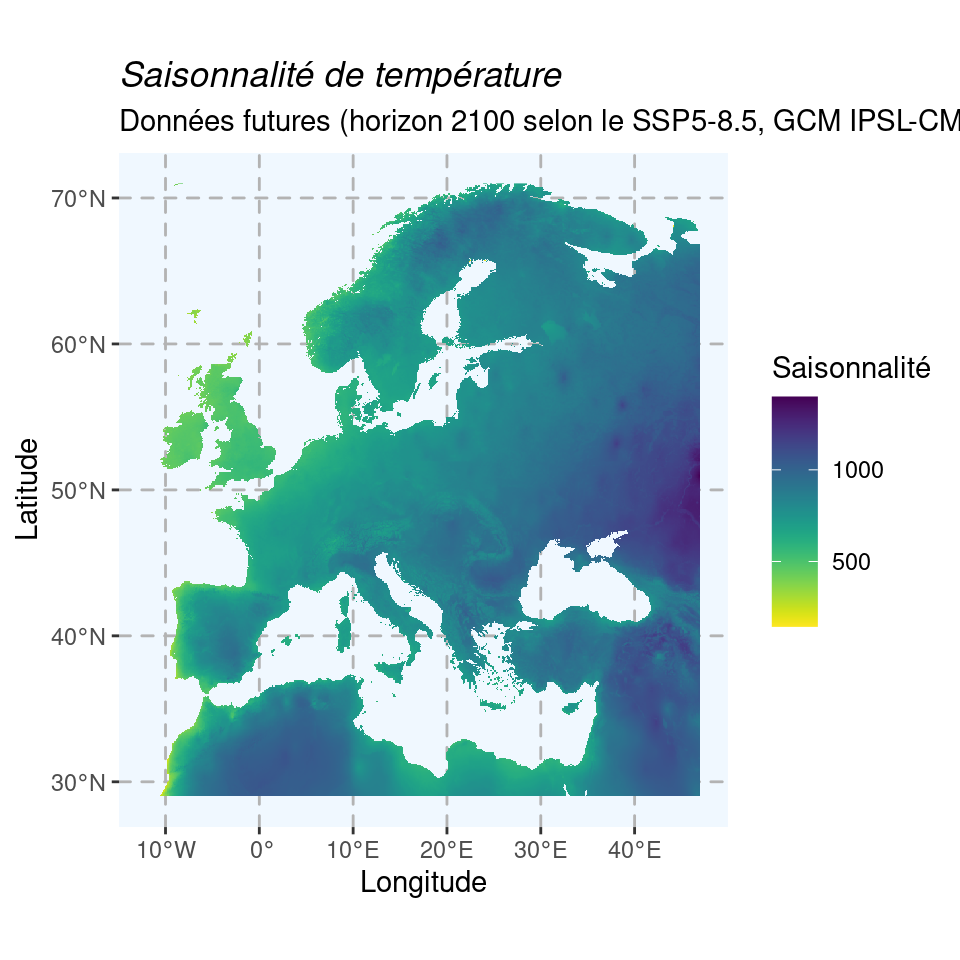
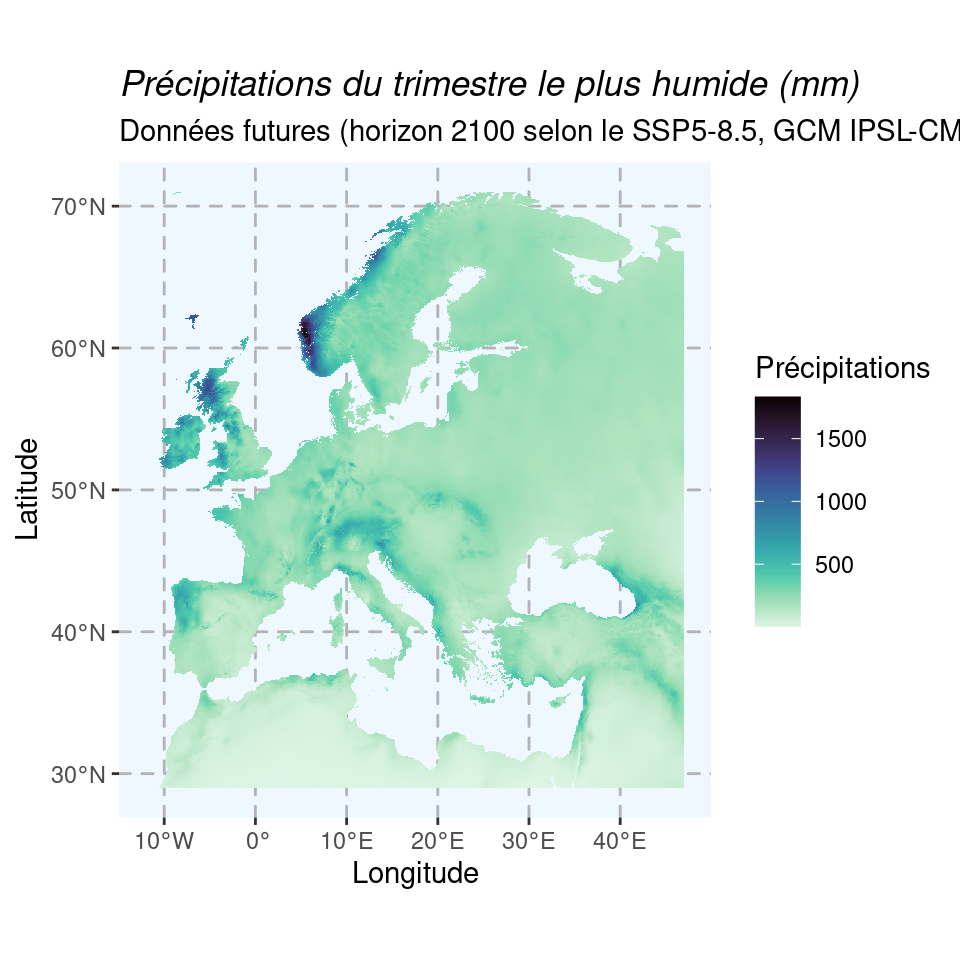
Figure 1.9: Cartes du climat futur en 2080–2100 (SSP5-8.5) selon le modèle IPSL-CM6A-LR.
1.2.3.2 GCM « CanESM5 »
On refait la même chose avec le modèle du Canadian earth system version 5
(CanESM5).
On télécharge également les températures maximales en août.
Puis l’on sélectionne et combine les 6 variables d’intérêt.
On utilise ensuite le masque pour l’Europe.
Carte pour les mêmes variables de température et de précipitation, pour la période 2080–2100 du scénario SSP5-8.5 et le modèle CanESM5 :
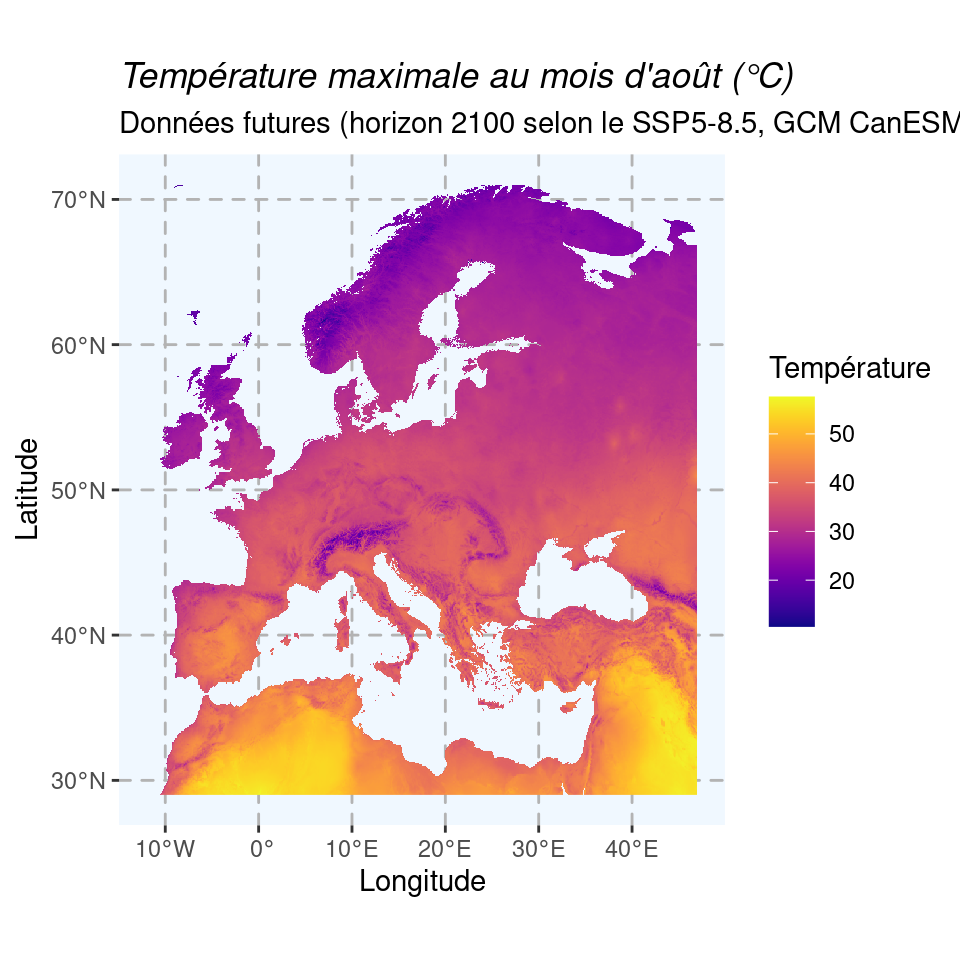
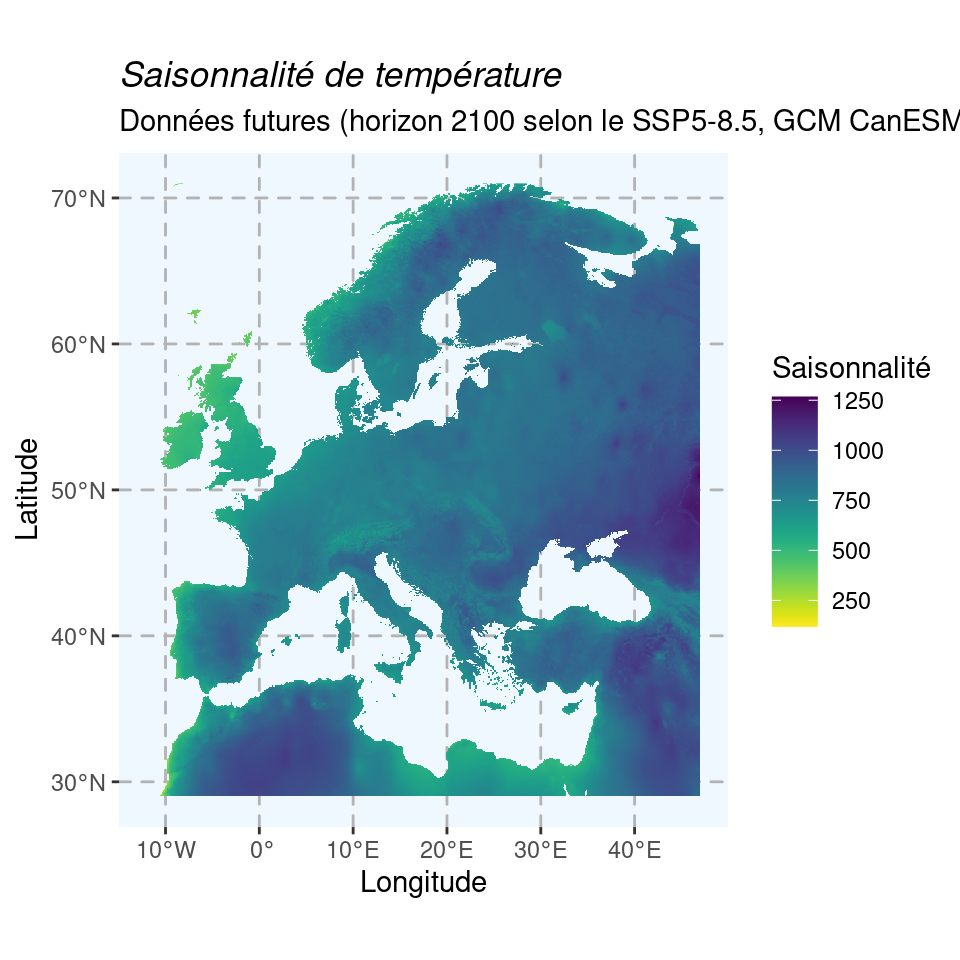
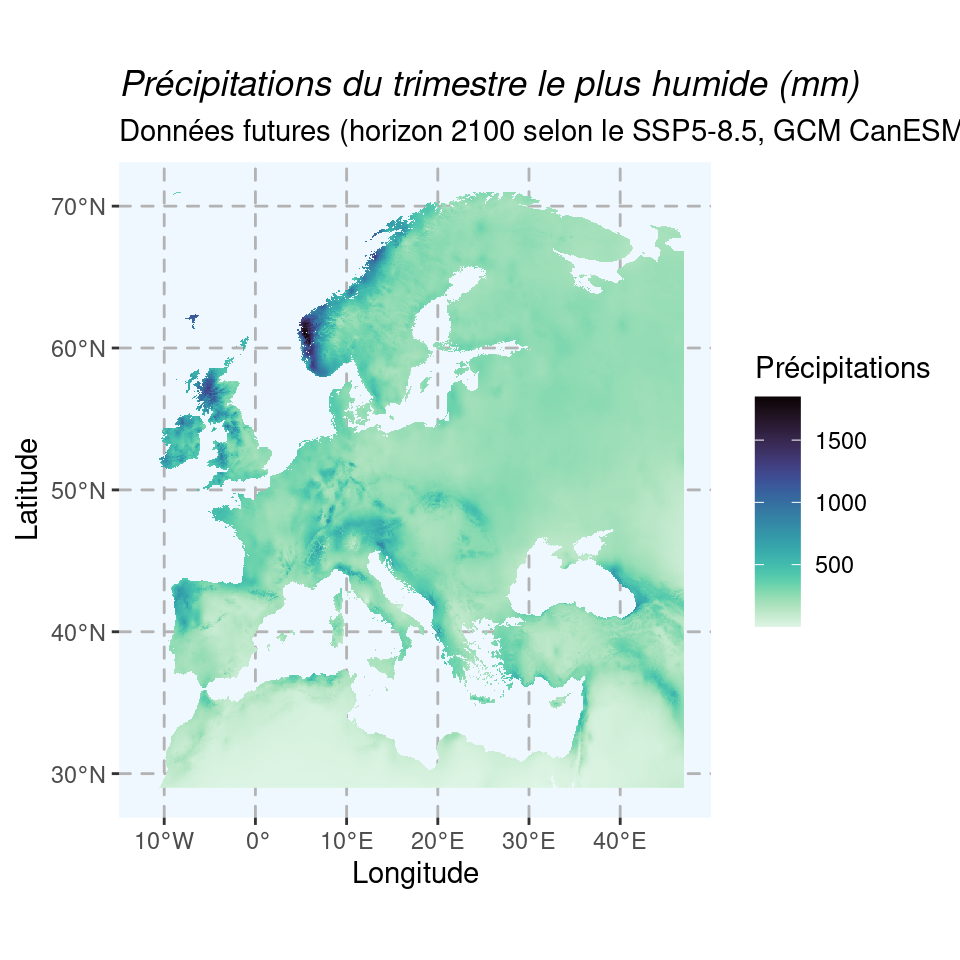
Figure 1.10: Cartes du climat futur en 2080–2100 (SSP5-8.5) selon le modèle CanESM5.
1.2.3.3 GCM « MIROC6 »
On refait la même chose avec le modèle pour la recherche interdisciplinaire sur
le climat version 6 (MIROC6).
On télécharge également les températures maximales en août.
Puis l’on sélectionne et combine les 6 variables d’intérêt.
On utilise ensuite le masque pour l’Europe.
Carte pour les mêmes variables de température et de précipitation, pour la période 2080–2100 du scénario SSP5-8.5 et le modèle MIROC6.
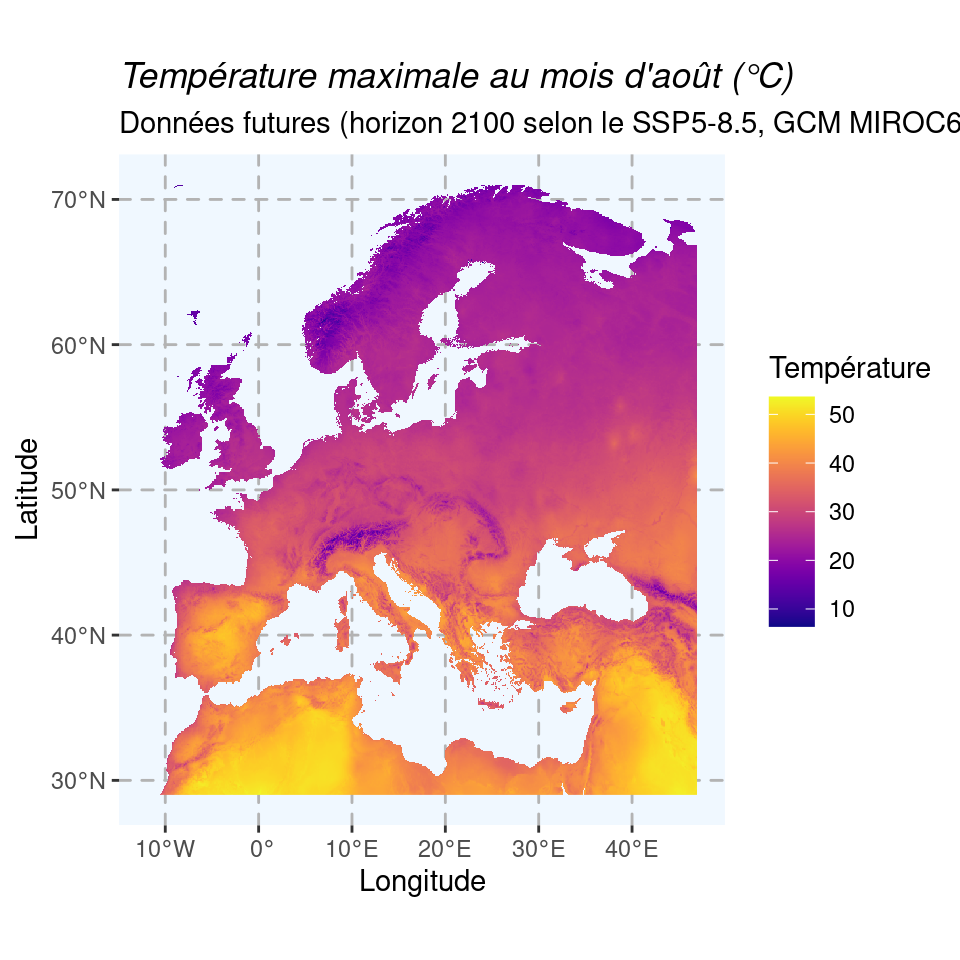
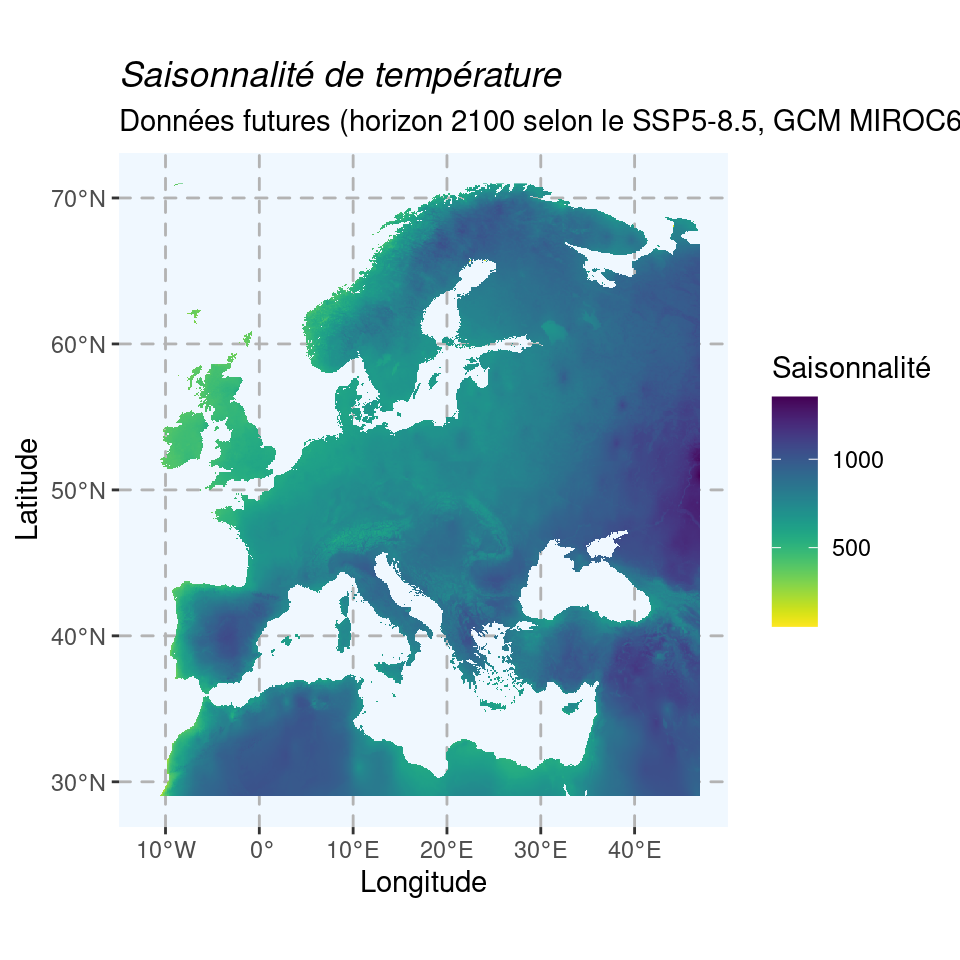
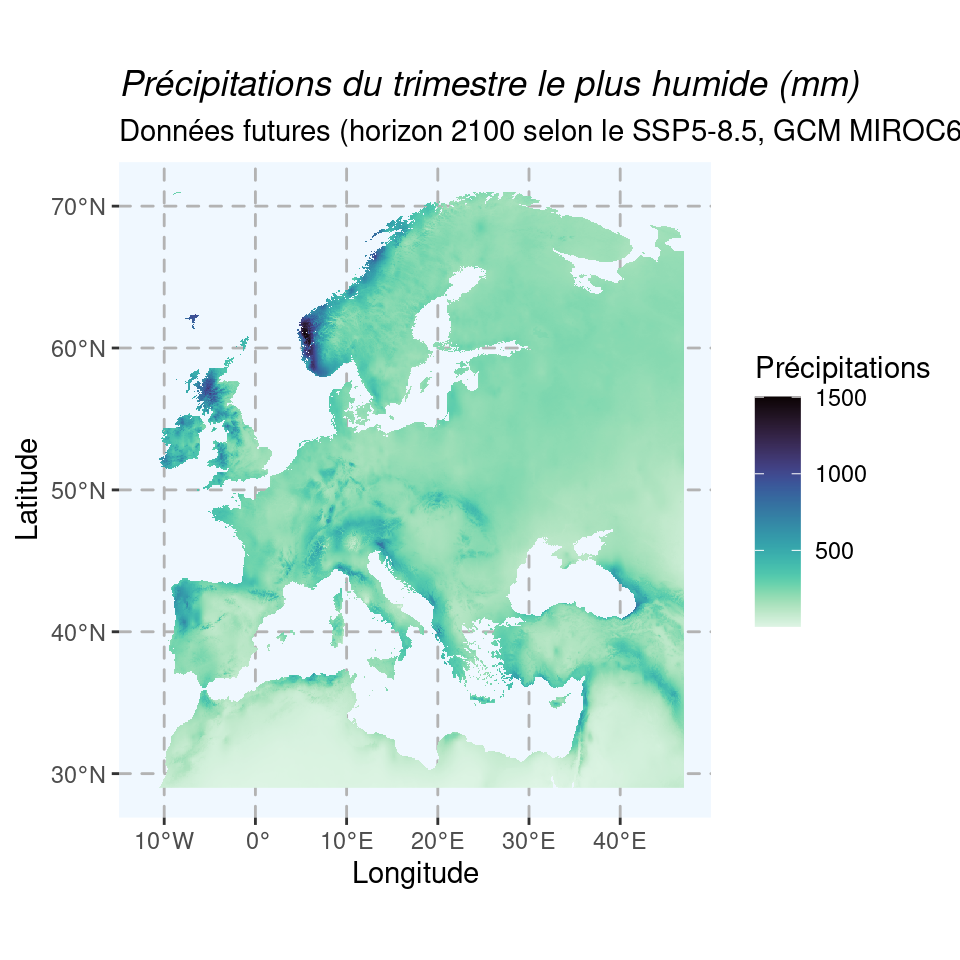
Figure 1.11: Cartes du climat futur en 2080–2100 (SSP5-8.5) selon le modèle MIROC6.
1.2.3.4 Comparaison des GCMs
Pour comparer les GCMs, on regarde sur la fenêtre temporelle 2080–2100, c’est-à-dire la plus éloignée (permettant ainsi des divergences maximales), et sur deux scénarios opposés : le SSP1-2.6 et le SSP5-8.5.
Commençons par le SSP1-2.6.
[,1] [,2] [,3]
[1,] 1 1 1
[2,] 1 1 1
[3,] 1 1 1 [,1] [,2] [,3]
[1,] 1.00 0.99 0.99
[2,] 0.99 1.00 0.99
[3,] 0.99 0.99 1.00 [,1] [,2] [,3]
[1,] 1.00 0.89 0.93
[2,] 0.89 1.00 0.91
[3,] 0.93 0.91 1.00 [,1] [,2] [,3]
[1,] 1.00 0.99 0.99
[2,] 0.99 1.00 0.99
[3,] 0.99 0.99 1.00 [,1] [,2] [,3]
[1,] 1 1.00 1.00
[2,] 1 1.00 0.99
[3,] 1 0.99 1.00 [,1] [,2] [,3]
[1,] 1.00 0.99 0.99
[2,] 0.99 1.00 1.00
[3,] 0.99 1.00 1.00Dans ce premier cas, toutes les corrélations sont extrêmement proches de 1, avec seulement des déviations concernant la température du trimestre le plus humide (corrélation de 0.89 minimum toutefois).
La même chose avec le SSP5-8.5 :
[,1] [,2] [,3]
[1,] 1.00 0.99 0.99
[2,] 0.99 1.00 0.98
[3,] 0.99 0.98 1.00 [,1] [,2] [,3]
[1,] 1.00 0.96 0.98
[2,] 0.96 1.00 0.98
[3,] 0.98 0.98 1.00 [,1] [,2] [,3]
[1,] 1.00 0.69 0.77
[2,] 0.69 1.00 0.78
[3,] 0.77 0.78 1.00 [,1] [,2] [,3]
[1,] 1.00 0.95 0.95
[2,] 0.95 1.00 0.95
[3,] 0.95 0.95 1.00 [,1] [,2] [,3]
[1,] 1.00 0.99 0.98
[2,] 0.99 1.00 0.99
[3,] 0.98 0.99 1.00 [,1] [,2] [,3]
[1,] 1.00 0.98 0.99
[2,] 0.98 1.00 0.98
[3,] 0.99 0.98 1.00Pour toutes les variables sauf la température du trimestre le plus humide, les corrélations sont à nouveau extrêmement proches de 1. Pour la température du trimestre le plus humide, les déviations sont un peu plus fortes, avec toutefois des corrélations supérieures à 0.69 dans tous les cas (ces déviations pourraient tout simplement être le résultat de définitions différentes du trimestre le plus froid).
En conclusion, il est inutile de considérer plusieurs GCMs. À l’échelle de
l’étude, le surcoût d’analyse ne justifie pas d’intégrer les différences minimes
entre modèles. On considérera donc uniquement le modèle de l’Institut
Pierre-Simon Laplace version 6A à faible résolution (IPSL-CM6A-LR).
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 3651755 195.1 7156917 382.3 7156917 382.3
Vcells 6152280 47.0 12255594 93.6 9048502 69.11.2.4 Données Grenoble
1.2.4.1 Températures de surface de la métropole de Grenoble (août 2019)
On commence par charger les deux packages qui nous serviront pour la
préparation des données raster : terra (évolution du package raster, mais
qui n’est pas encore utilisé par tous les autres packages) et tidyterra
(intégration de terra à dplyr).
On récupère les données de températures de surface de la métropole de Grenoble (août 2019), fournies par Grenoble-Alpes Métropole. Il s’agit d’un raster en projection RGF93 / CC45, la projection de référence pour la France (Lambert conique conforme) au niveau de Grenoble. On utilisera plus tard les coordonnées de la ville de Grenoble comme point de référence (latitude : 45.188529°N, longitude : 5.724524°E).
class : SpatRaster
dimensions : 1364, 829, 1 (nrow, ncol, nlyr)
resolution : 29.99287724, 29.99287724 (x, y)
extent : 1901804.347, 1926668.442, 4198479.105, 4239389.389 (xmin, xmax, ymin, ymax)
coord. ref. : RGF93 v1 / CC45 (EPSG:3945)
source(s) : memory
name : temp_surf_3945_clip
min value : 23.42581177
max value : 53.57958984 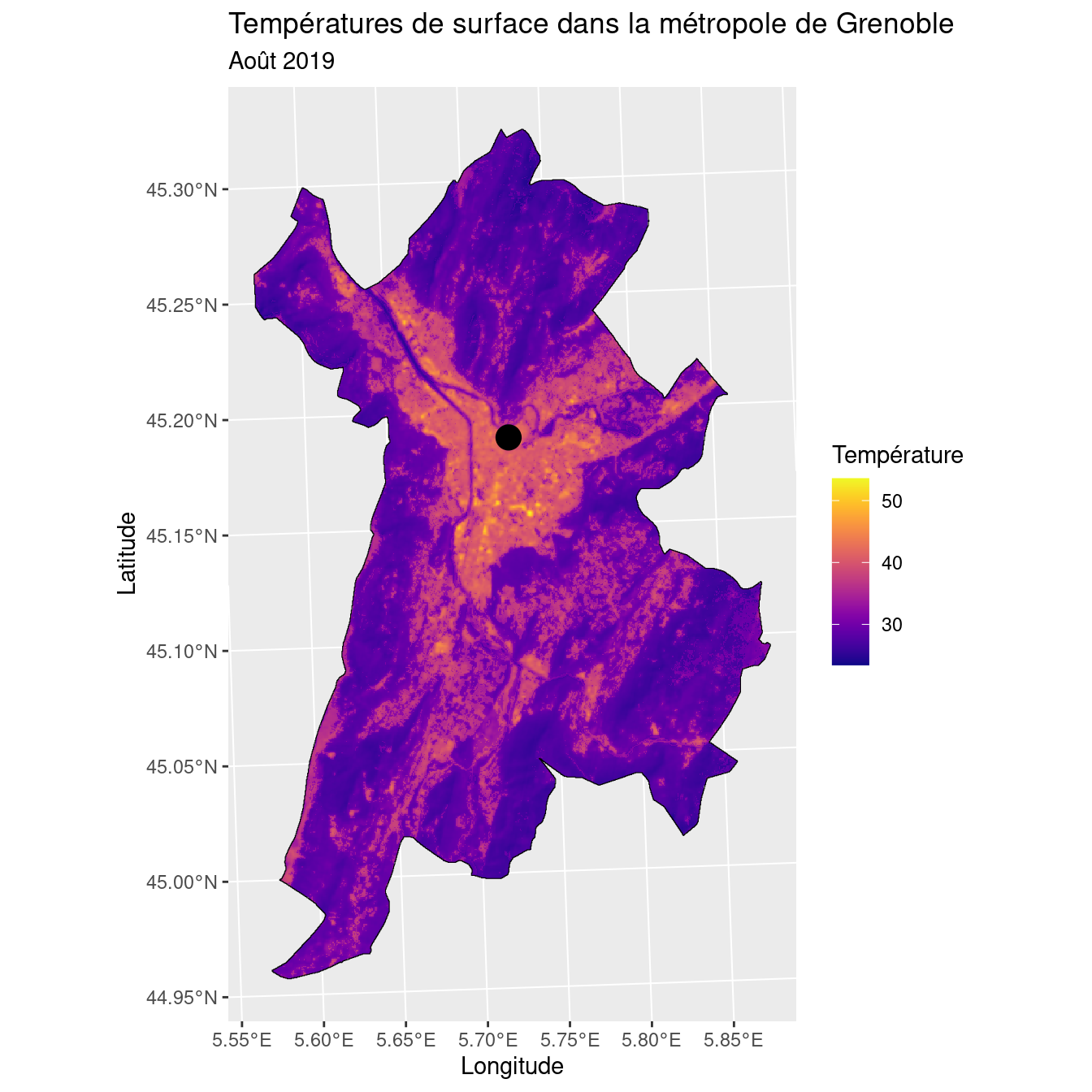
Figure 1.12: Carte des températures de surface dans la métropole de Grenoble, août 2019. Le point représente le centre ville de Grenoble comme référence.
1.2.4.2 Interpolation à l’échelle de Grenoble : données climatiques contemporaines
- Croisement avec températures de surface 2019 (fournie par la métropole)
WorldClim sur Grenoble représente à peu près 7 pixels × 9 pixels ; on reprojette sur le raster des températures d’août 2019, ce qui induit une interpolation bilinéaire pour reconstruire à la bonne résolution. On découpe ensuite le résultat pour coller au raster original de Grenoble :
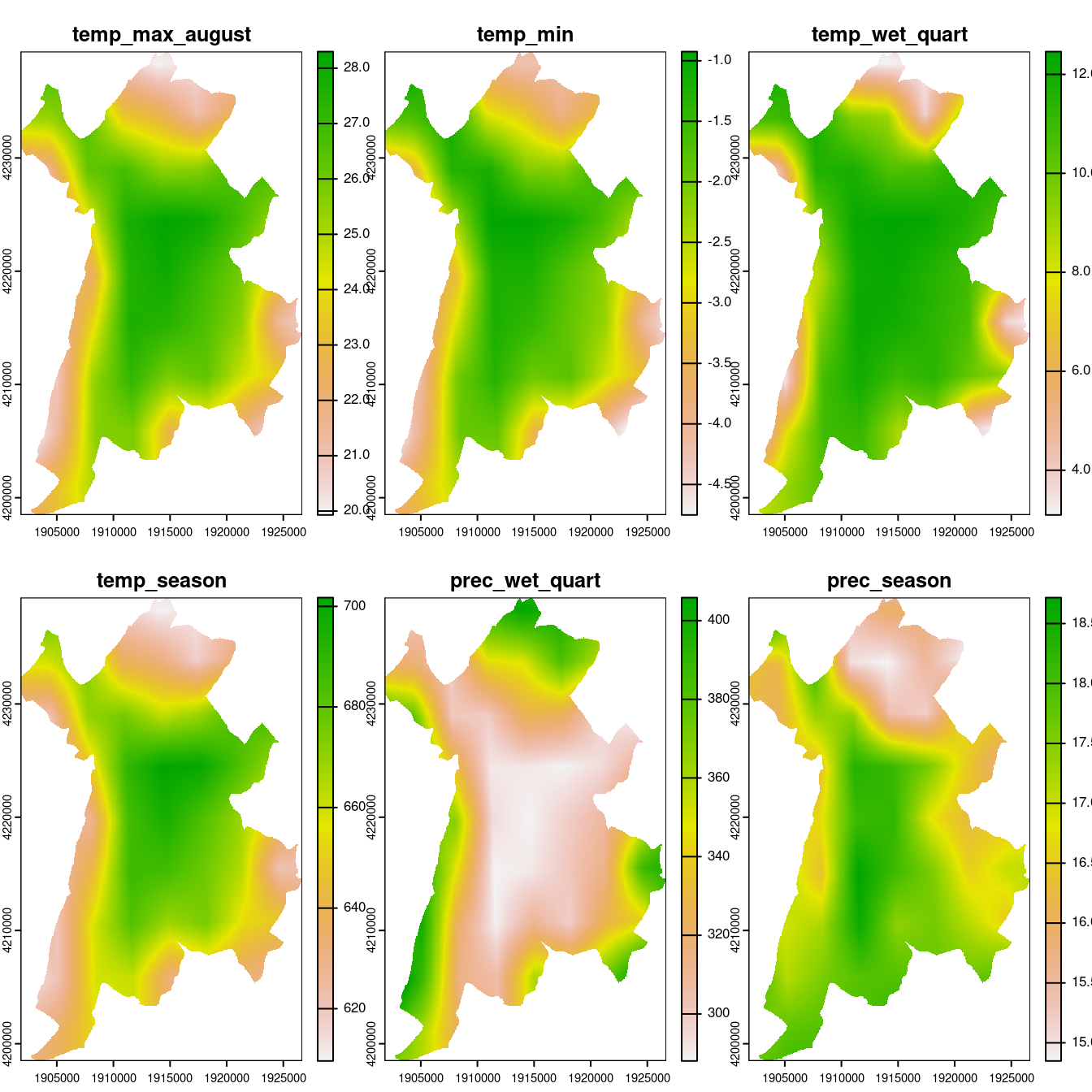
Figure 1.13: Conditions climatiques contemporaines (WorldClim) projetées sur l’emprise et la résolution du raster de Grenoble.
On calcule la température moyenne sur la métropole de Grenoble dans le raster WorldClim des conditions climatiques contemporaines, ainsi que dans le raster d’août 2019. On recentre alors le raster d’août 2019 en utilisant la moyenne WorldClim.
mean
temp_max_august 25.09620887 mean
temp_surf_3945_clip 31.42647347 mean
-6.330264607 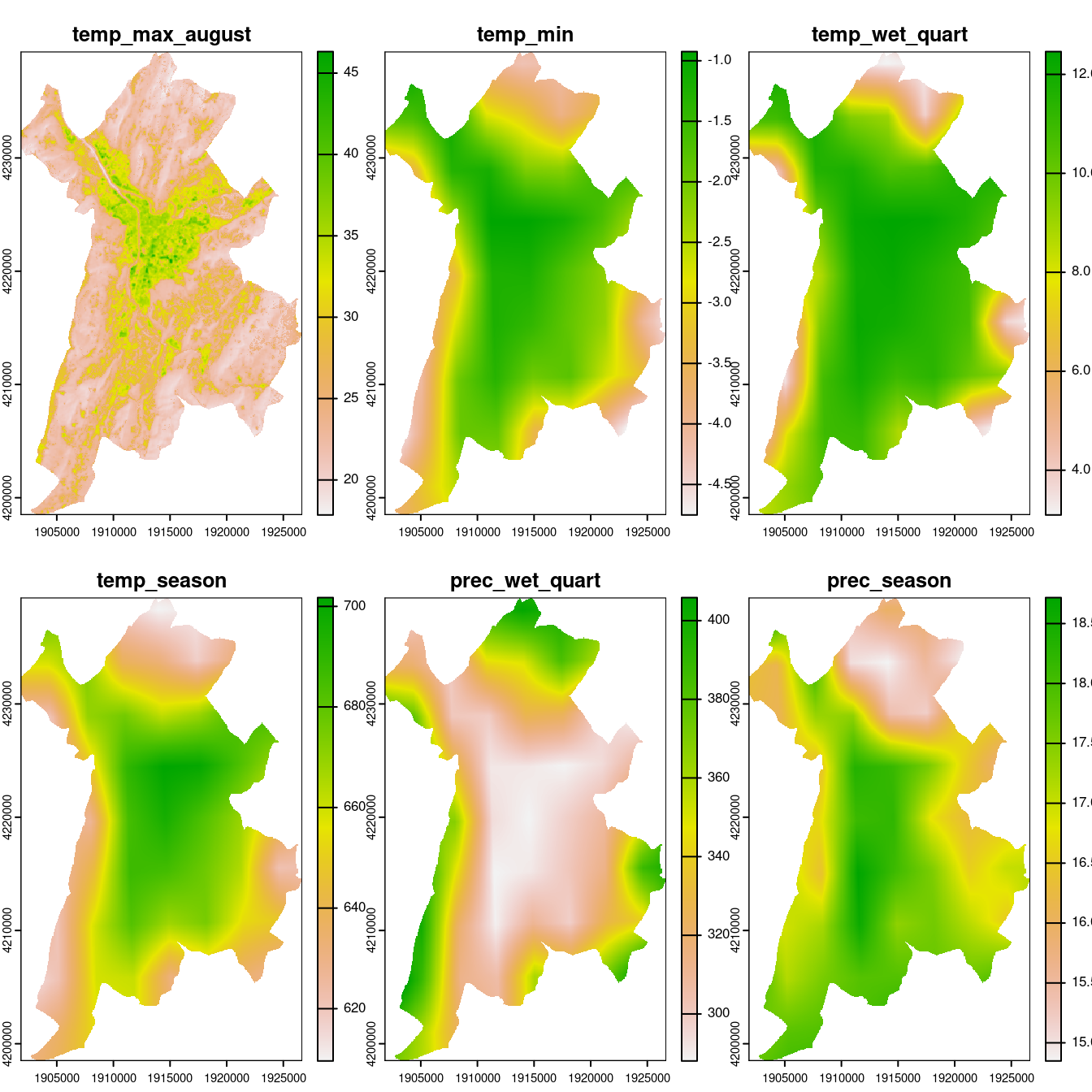
Figure 1.14: Conditions climatiques contemporaines (WorldClim) en prenant en compte la température locale à Grenoble.
On vérifie qu’on a bien la même moyenne que celle donnée par WorldClim initialement :
mean
temp_max_august 25.096208861.2.4.3 Interpolation à l’échelle de Grenoble : données climatiques futures (GCM IPSL-CM6A-LR)
mean
temp_max_august 27.70720895 mean
temp_surf_3945_clip 31.42647347 mean
-3.719264521 mean
temp_max_august 28.49844428 mean
temp_surf_3945_clip 31.42647347 mean
-2.928029192 mean
temp_max_august 28.69823569 mean
temp_surf_3945_clip 31.42647347 mean
-2.728237779 mean
temp_max_august 28.5617606 mean
temp_surf_3945_clip 31.42647347 mean
-2.864712871 mean
temp_max_august 27.70720895 mean
temp_max_august 28.49844428 mean
temp_max_august 28.69823569 mean
temp_max_august 28.5617606 mean
temp_max_august 27.99383706 mean
temp_surf_3945_clip 31.42647347 mean
-3.432636413 mean
temp_max_august 29.21383694 mean
temp_surf_3945_clip 31.42647347 mean
-2.21263653 mean
temp_max_august 30.21118186 mean
temp_surf_3945_clip 31.42647347 mean
-1.215291615 mean
temp_max_august 30.9147084 mean
temp_surf_3945_clip 31.42647347 mean
-0.5117650676 mean
temp_max_august 27.99383706 mean
temp_max_august 29.21383694 mean
temp_max_august 30.21118186 mean
temp_max_august 30.9147084 mean
temp_max_august 27.79549425 mean
temp_surf_3945_clip 31.42647347 mean
-3.630979227 mean
temp_max_august 29.23922327 mean
temp_surf_3945_clip 31.42647347 mean
-2.187250199 mean
temp_max_august 31.51140698 mean
temp_surf_3945_clip 31.42647347 mean
0.08493350884 mean
temp_max_august 33.93622401 mean
temp_surf_3945_clip 31.42647347 mean
2.509750535 mean
temp_max_august 27.79549425 mean
temp_max_august 29.23922327 mean
temp_max_august 31.51140698 mean
temp_max_august 33.93622401 mean
temp_max_august 27.98252124 mean
temp_surf_3945_clip 31.42647347 mean
-3.443952237 mean
temp_max_august 29.81140786 mean
temp_surf_3945_clip 31.42647347 mean
-1.615065613 mean
temp_max_august 33.11298093 mean
temp_surf_3945_clip 31.42647347 mean
1.686507453 mean
temp_max_august 36.59007449 mean
temp_surf_3945_clip 31.42647347 mean
5.163601021 mean
temp_max_august 27.98252124 mean
temp_max_august 29.81140786 mean
temp_max_august 33.11298092 mean
temp_max_august 36.59007449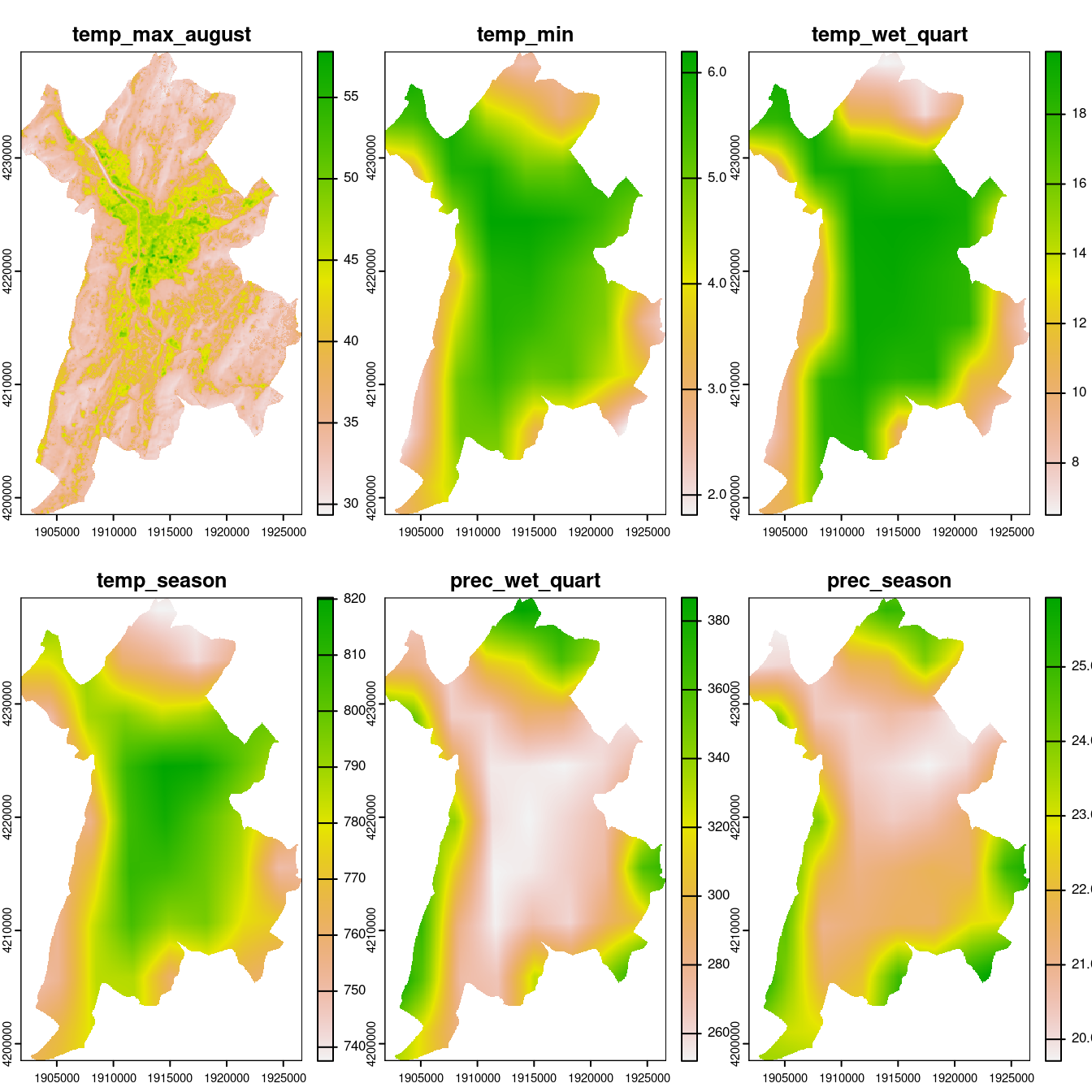
Figure 1.15: Conditions climatiques à l’horizon 2100 (WorldClim, SSP5-8.5, GCM IPSL-CM6A-LR) en prenant en compte la température locale à Grenoble.
used (Mb) gc trigger (Mb) max used (Mb)
Ncells 3659452 195.5 7156917 382.3 7156917 382.3
Vcells 6160475 47.1 12255594 93.6 9048502 69.1