240 Carya cordiformis
240.1 Données d’occurrences GBIF
Collecte des données du GBIF :
<<gbif download metadata>>
Status: SUCCEEDED
DOI: 10.15468/dl.k3dyrm
Format: SIMPLE_CSV
Download key: 0011944-230828120925497
Created: 2023-09-11T11:35:08.043+00:00
Modified: 2023-09-11T11:36:03.893+00:00
Download link: https://api.gbif.org/v1/occurrence/download/request/0011944-230828120925497.zip
Total records: 2958Conversion en objets géographiques (format Simple feature de sf) et
exploration rapide des données (classe du jeu de données et premières lignes du
tableau) :
Simple feature collection with 2958 features and 50 fields
Attribute-geometry relationships: constant (50)
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -123.25 ymin: 29.499967 xmax: 25.947515 ymax: 56.684089
Geodetic CRS: WGS 84
# A tibble: 2,958 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
2 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
3 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
4 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
5 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
6 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
7 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
8 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
9 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
10 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
# ℹ 2,948 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <lgl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <lgl>, depthAccuracy <lgl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …Il y a 2 958 occurrences dans le jeu de données. On les affiche sur la carte du monde :
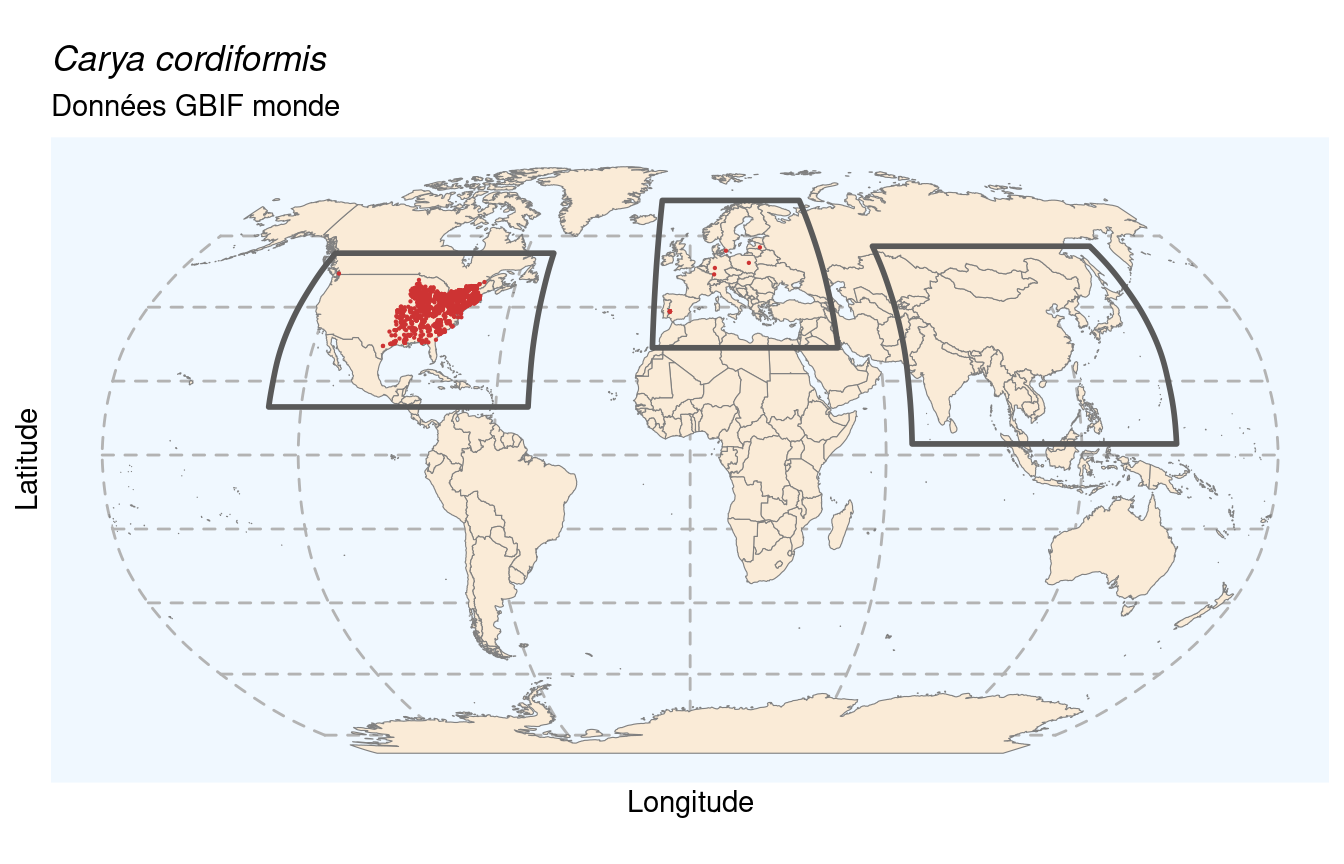
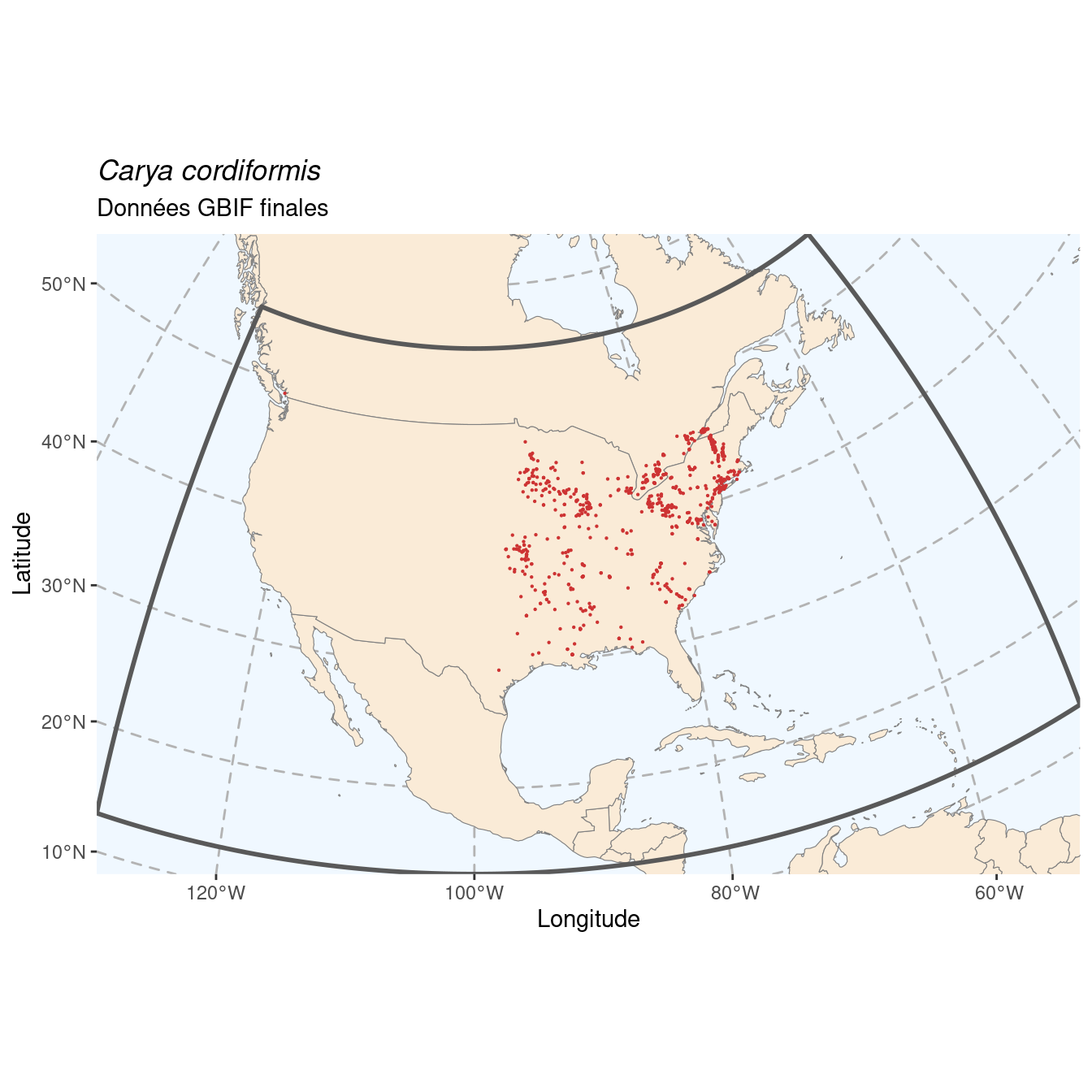
Figure 240.1: Occurrences de Carya cordiformis dans le monde.
240.1.1 Données par région
On évalue la proportion d’occurrences dans chaque région, en utilisant les 3 masques créés précédemment. Si on a au moins 50 % des occurrences dans une région, on peut continuer en travaillant uniquement sur ces données. Sinon, on vérifie qu’au moins 67 % des données tombent dans l’ensemble des régions, et on prend la région la plus représentée. Au final, s’il s’agit d’une espèce endémique d’Europe, on n’a pas besoin de manipulation supplémentaire.
[1] 0.005409060176[1] 0.9945909398[1] 0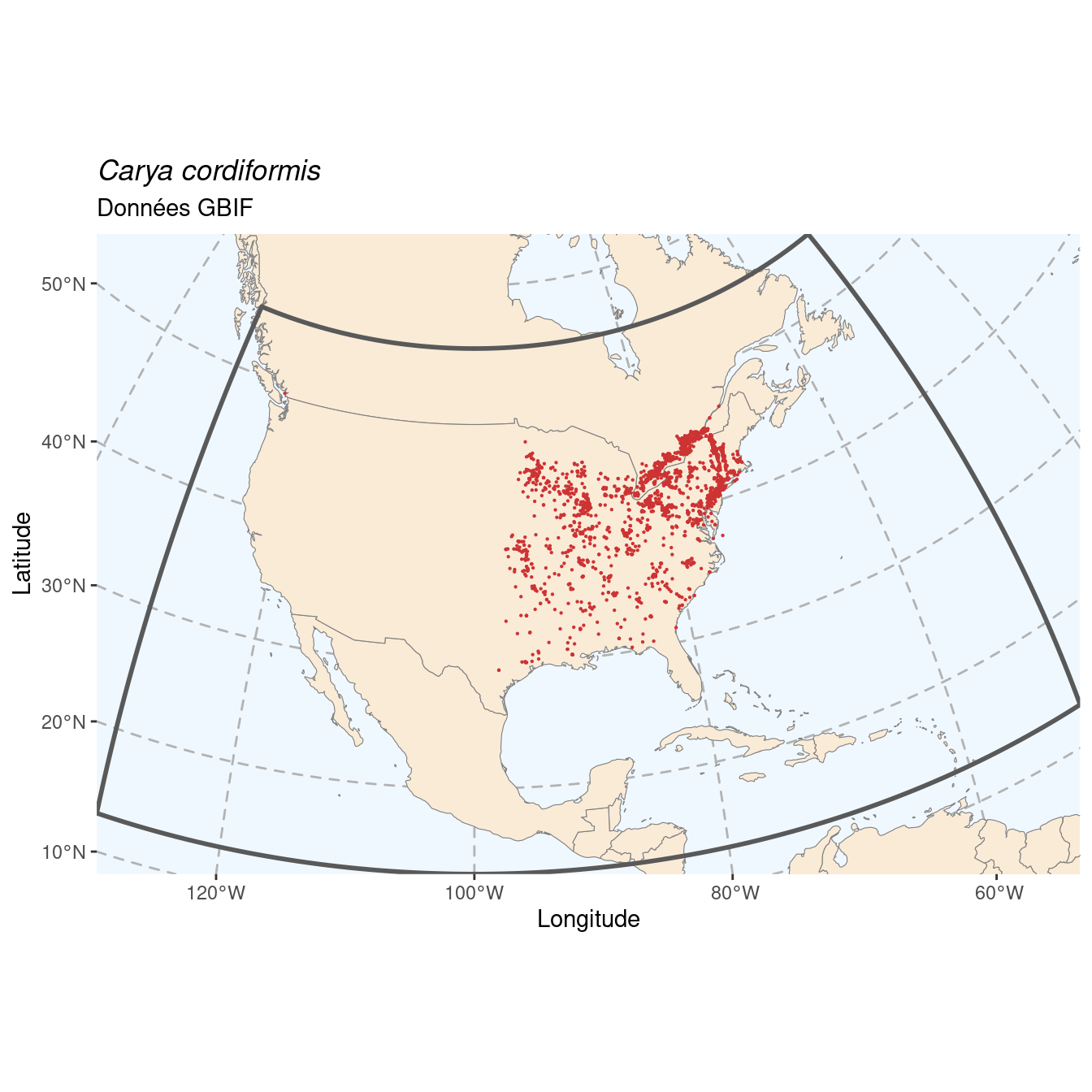
Figure 240.2: Occurrence de Carya cordiformis dans la région d’endémisme.
240.1.2 Licences
On s’intéresse ici au problème particulier des licences :
CC_BY_4_0 CC_BY_NC_4_0 CC0_1_0
407 2086 449 [1] 29.09585316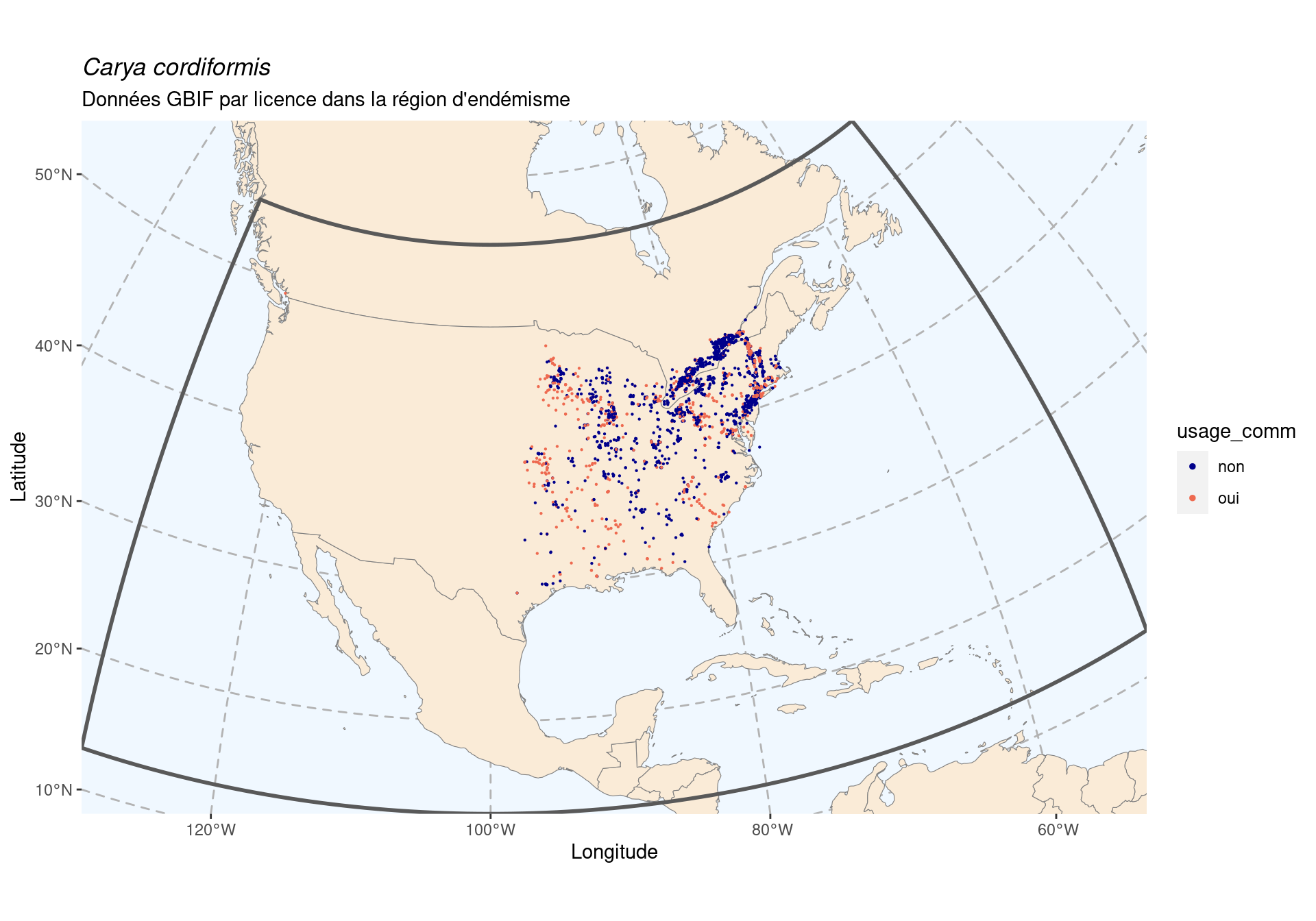
Figure 240.3: Occurrence de Carya cordiformis dans la région d’endémisme, selon la licence : CC BY-NC en bleu ; CC0 et CC BY en orange.
Idéalement, on aimerait avoir au moins 50 % de données vraiment libres
d’utilisation (c’est-à-dire sans la clause -NC). On continue en supprimant les
données en CC BY-NC.
[1] 856240.1.3 Sous-échantillonnage
Si le jeu de données contient 25 000 occurrences ou plus, on procède au sous-échantillonnage aléatoire pour en conserver un maximum de 25 000.
Simple feature collection with 856 features and 50 fields
Attribute-geometry relationships: constant (50)
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -123.25 ymin: 29.49997 xmax: -71.122465 ymax: 49.25
Geodetic CRS: WGS 84
# A tibble: 856 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
<int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
2 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
3 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
4 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
5 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
6 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
7 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
8 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
9 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
10 9 e8 0096dfc0-… http://bioi… Plantae Trach… Magn… Faga… Jugla… Carya Carya … ""
# ℹ 846 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <lgl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <lgl>, depthAccuracy <lgl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …240.2 Modélisation de la niche climatique
240.2.1 Préparer les données
Organisation des données d’entrées (occurrences et données environnementales) et des données produites de pseudo-absences :
class : SpatVector
geometry : points
dimensions : 856, 0 (geometries, attributes)
extent : -123.25, -71.122465, 29.49997, 49.25 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= caco Data Formating -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
! Response variable is considered as only presences... Is it really what you want?
! No data has been set aside for modeling evaluation
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
Checking Pseudo-absence selection arguments...
> random pseudo absences selection
> Pseudo absences are selected in explanatory variables
! Some NAs have been automatically removed from your data
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.formated.data -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
dir.name = Output
sp.name = caco
854 presences, 0 true absences and 2568 undefined points in dataset
6 explanatory variables
temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart
Min. :13.62800 Min. :-30.596001 Min. :-10.23467 Min. : 68.35162 Min. : 26.0000
1st Qu.:24.28000 1st Qu.:-15.168000 1st Qu.: 12.27933 1st Qu.: 768.90381 1st Qu.: 229.0000
Median :27.34200 Median : -8.888000 Median : 17.36800 Median : 911.54163 Median : 298.0000
Mean :27.32604 Mean : -8.742956 Mean : 16.11399 Mean : 897.54314 Mean : 311.2525
3rd Qu.:31.09500 3rd Qu.: -2.748000 3rd Qu.: 20.94983 3rd Qu.:1082.02689 3rd Qu.: 341.0000
Max. :42.60000 Max. : 21.700001 Max. : 33.14667 Max. :1458.26929 Max. :1740.0000
prec_season
Min. : 5.816862
1st Qu.: 18.297724
Median : 32.757786
Mean : 38.842631
3rd Qu.: 55.149600
Max. :132.901352
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=On visualise les pseudo-absences :
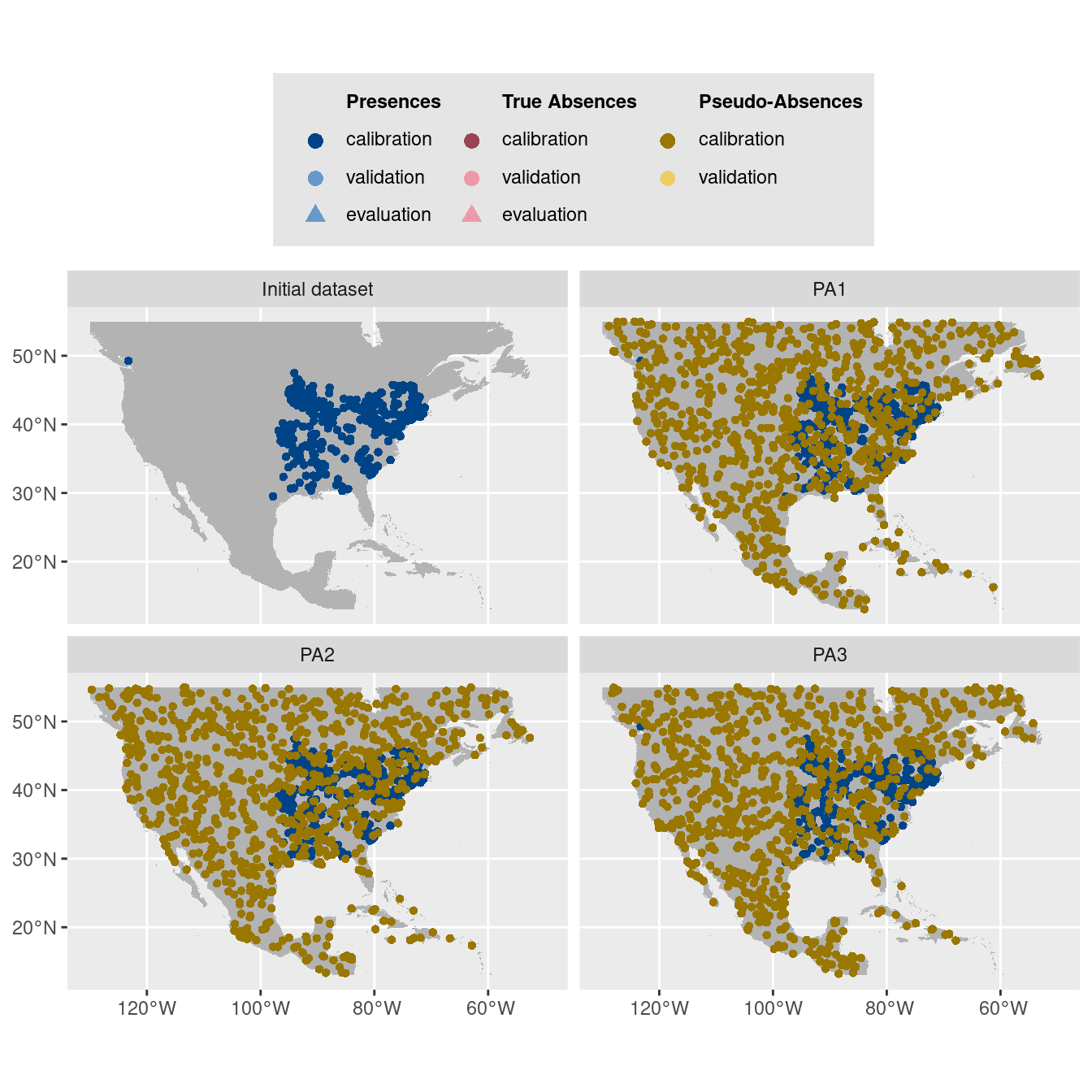
Figure 240.5: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
$data.vect
class : SpatVector
geometry : points
dimensions : 5984, 2 (geometries, attributes)
extent : -129.6875, -52.64583333, 13.0625, 54.97916667 (xmin, xmax, ymin, ymax)
coord. ref. :
names : resp dataset
type : <num> <chr>
values : 10 Initial dataset
10 Initial dataset
10 Initial dataset
$data.label
9 10 11
"**Presences**" "Presences (calibration)" "Presences (validation)"
12 19 20
"Presences (evaluation)" "**True Absences**" "True Absences (calibration)"
21 22 29
"True Absences (validation)" "True Absences (evaluation)" "**Pseudo-Absences**"
30 31 1
"Pseudo-Absences (calibration)" "Pseudo-Absences (validation)" "Background"
$data.plot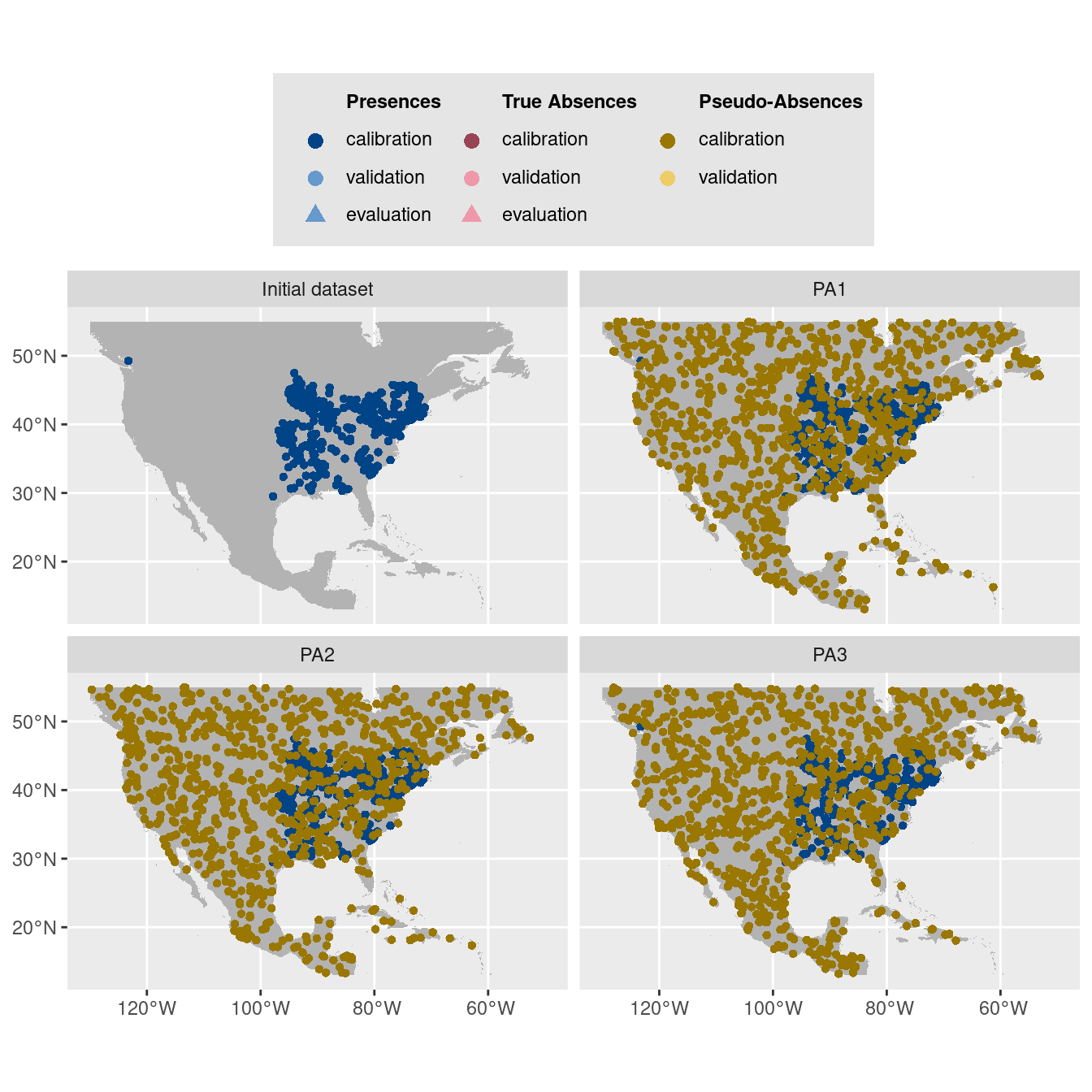
Figure 240.6: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
240.2.2 Modèles individuels de niche
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Build Single Models -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Checking Models arguments...
Creating suitable Workdir...
! Weights where automatically defined for caco_PA1 to rise a 0.5 prevalence !
! Weights where automatically defined for caco_PA2 to rise a 0.5 prevalence !
! Weights where automatically defined for caco_PA3 to rise a 0.5 prevalence !
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= caco Modeling Summary -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
6 environmental variables ( temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season )
Number of evaluation repetitions : 1
Models selected : GAM MARS MAXNET GBM ANN RF
Total number of model runs: 18
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=- Run : caco_PA1
-=-=-=--=-=-=- caco_PA1_RUN1
-=-=-=- Run : caco_PA2
-=-=-=--=-=-=- caco_PA2_RUN1
-=-=-=- Run : caco_PA3
-=-=-=--=-=-=- caco_PA3_RUN1
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=240.2.2.1 Évaluation de la performance
full.name PA run algo metric.eval cutoff sensitivity specificity calibration
1 caco_PA1_RUN1_GAM PA1 RUN1 GAM TSS 717.0 95.900 71.387 0.673
2 caco_PA1_RUN1_GAM PA1 RUN1 GAM ROC 717.5 95.900 71.387 0.801
3 caco_PA1_RUN1_MARS PA1 RUN1 MARS TSS 401.0 97.511 79.270 0.768
4 caco_PA1_RUN1_MARS PA1 RUN1 MARS ROC 398.0 97.657 79.124 0.942
5 caco_PA1_RUN1_MAXNET PA1 RUN1 MAXNET TSS 373.0 97.804 79.416 0.771
6 caco_PA1_RUN1_MAXNET PA1 RUN1 MAXNET ROC 371.5 97.804 79.416 0.936
validation evaluation
1 0.649 NA
2 0.765 NA
3 0.772 NA
4 0.920 NA
5 0.743 NA
6 0.925 NA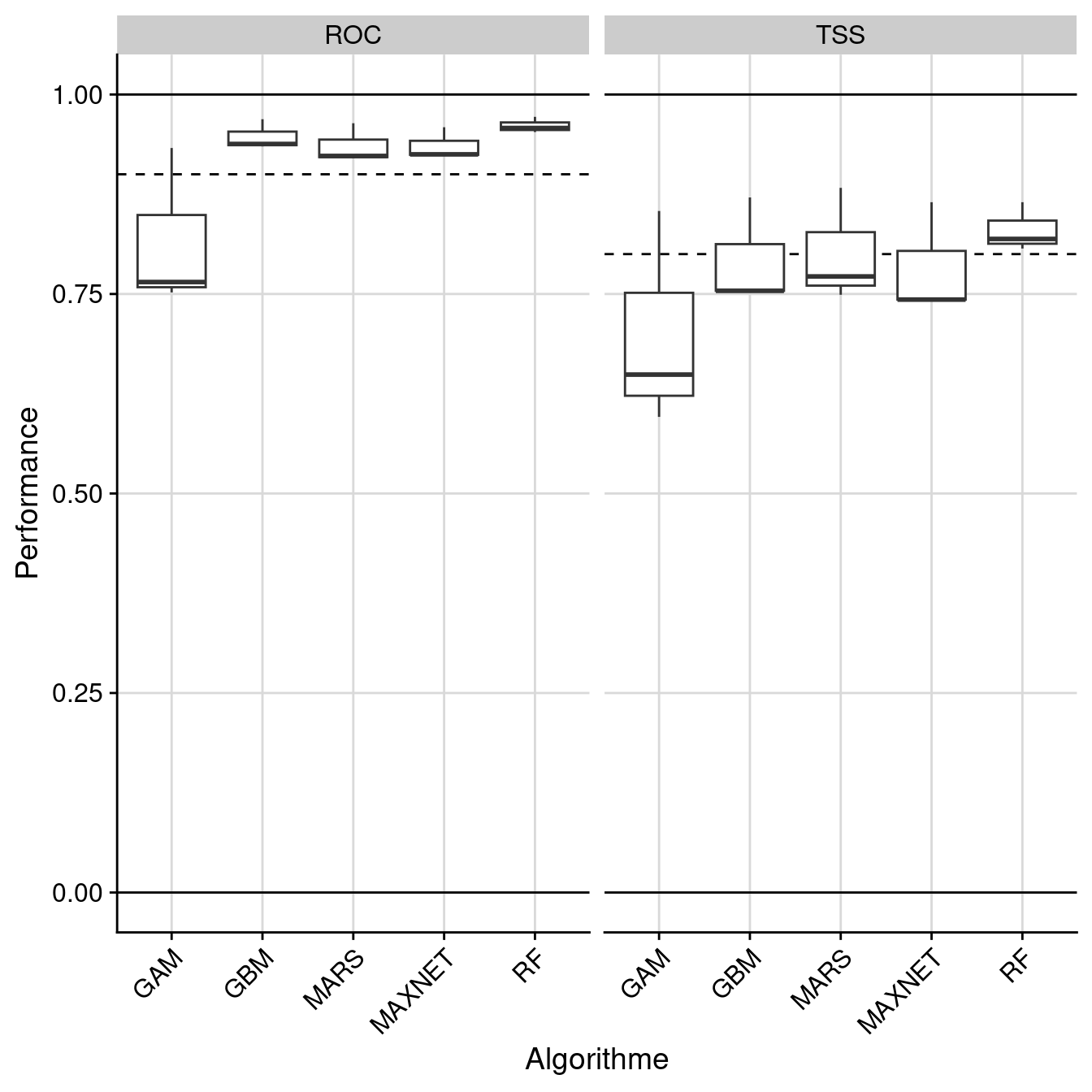
Figure 240.7: Évaluation de la performance des modèles individuels : True Skill Statistic & aire sous la courbe de ROC.
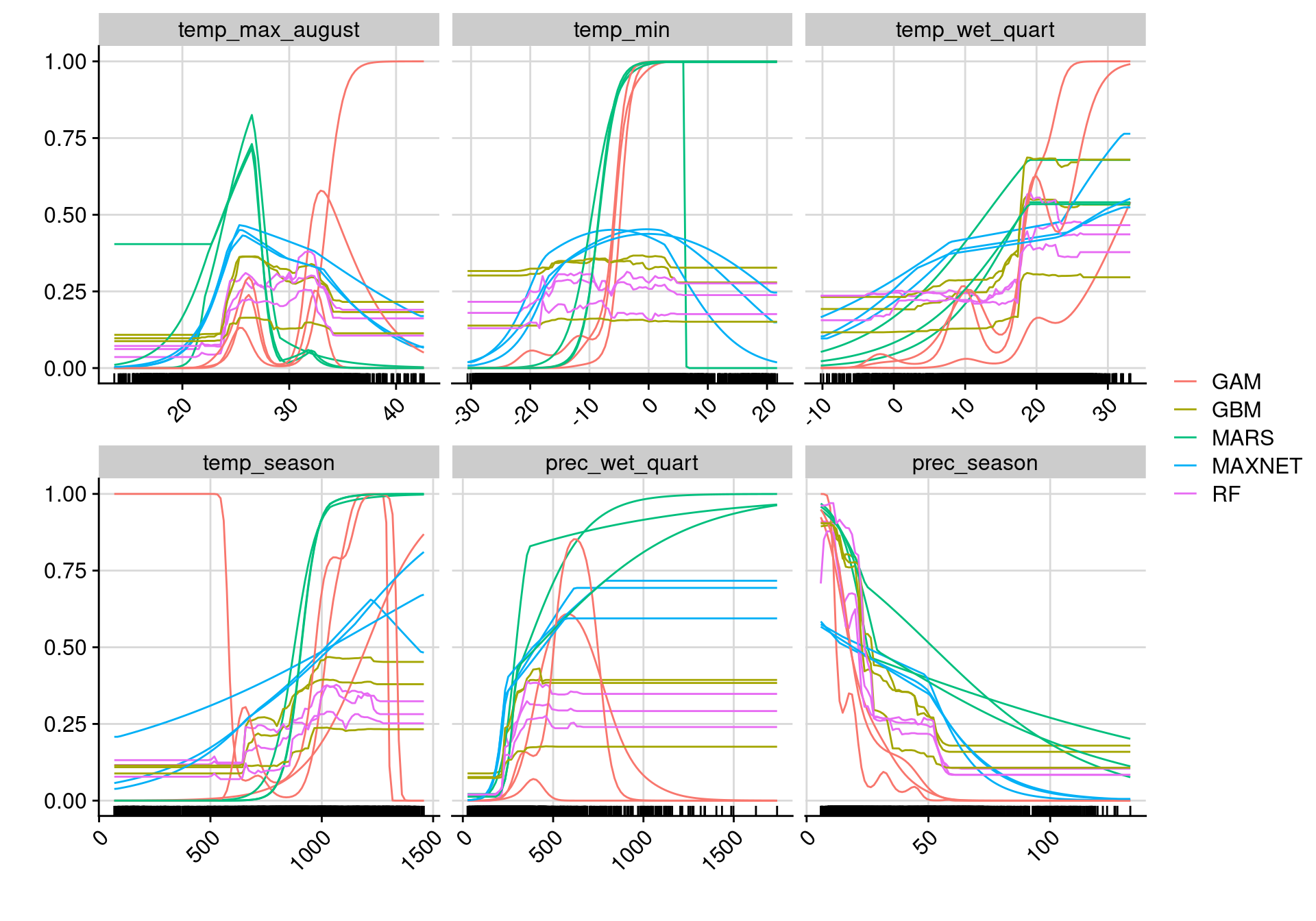
240.2.2.2 Importance des variables environnementales
full.name PA run algo expl.var rand var.imp
1 caco_PA1_RUN1_GAM PA1 RUN1 GAM temp_max_august 1 0.173457
2 caco_PA1_RUN1_GAM PA1 RUN1 GAM temp_min 1 0.384377
3 caco_PA1_RUN1_GAM PA1 RUN1 GAM temp_wet_quart 1 0.091599
4 caco_PA1_RUN1_GAM PA1 RUN1 GAM temp_season 1 0.265423
5 caco_PA1_RUN1_GAM PA1 RUN1 GAM prec_wet_quart 1 0.149037
6 caco_PA1_RUN1_GAM PA1 RUN1 GAM prec_season 1 0.516534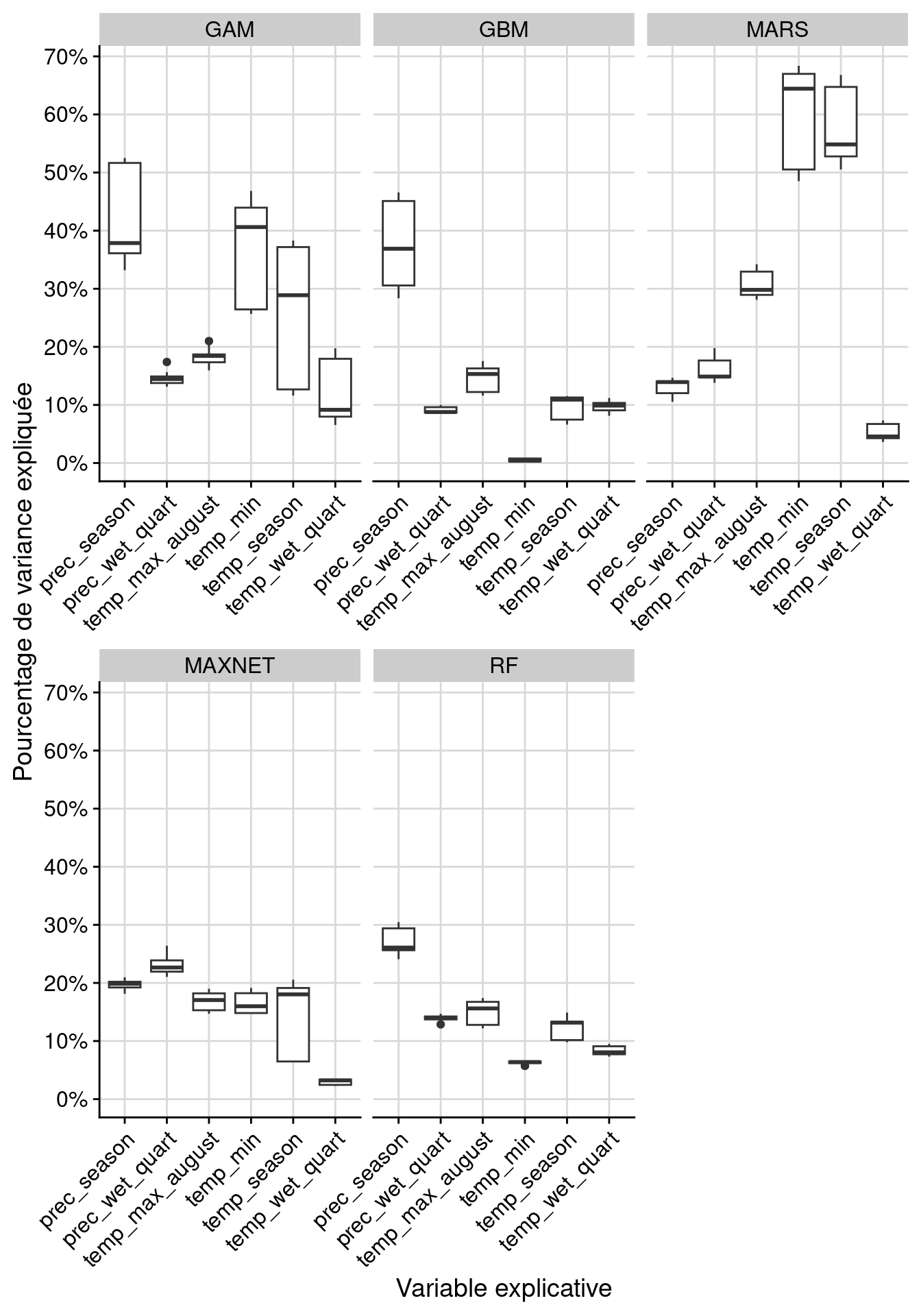
Figure 240.8: Pourcentage de variance expliquée pour chaque variable, décomposé par algorithme.
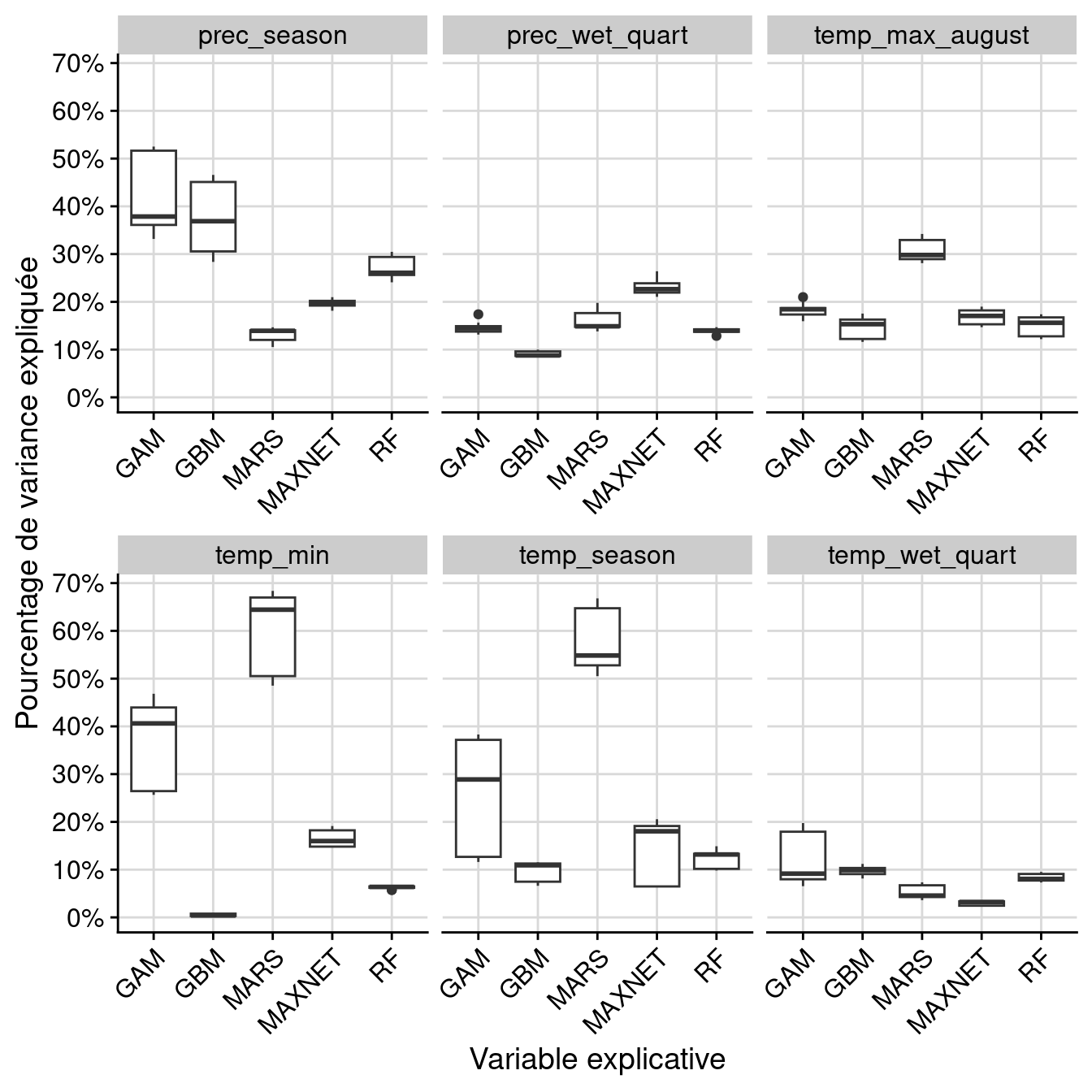
Figure 240.9: Pourcentage de variance expliquée pour chaque variable, décomposé par variable.
240.2.3 Modèles d’ensemble de niche
Seulement modèles avec TSS > 0.8
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.ensemble.models.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
sp.name : caco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
models computed:
caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, caco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
models failed: none
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=240.2.3.1 Évaluation de la qualité
full.name merged.by.PA merged.by.run merged.by.algo
1 caco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
2 caco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo
filtered.by algo metric.eval cutoff sensitivity specificity calibration validation evaluation
1 TSS EMwmean TSS 632.0 95.082 90.498 0.856 NA NA
2 TSS EMwmean ROC 634.5 95.082 90.732 0.981 NA NA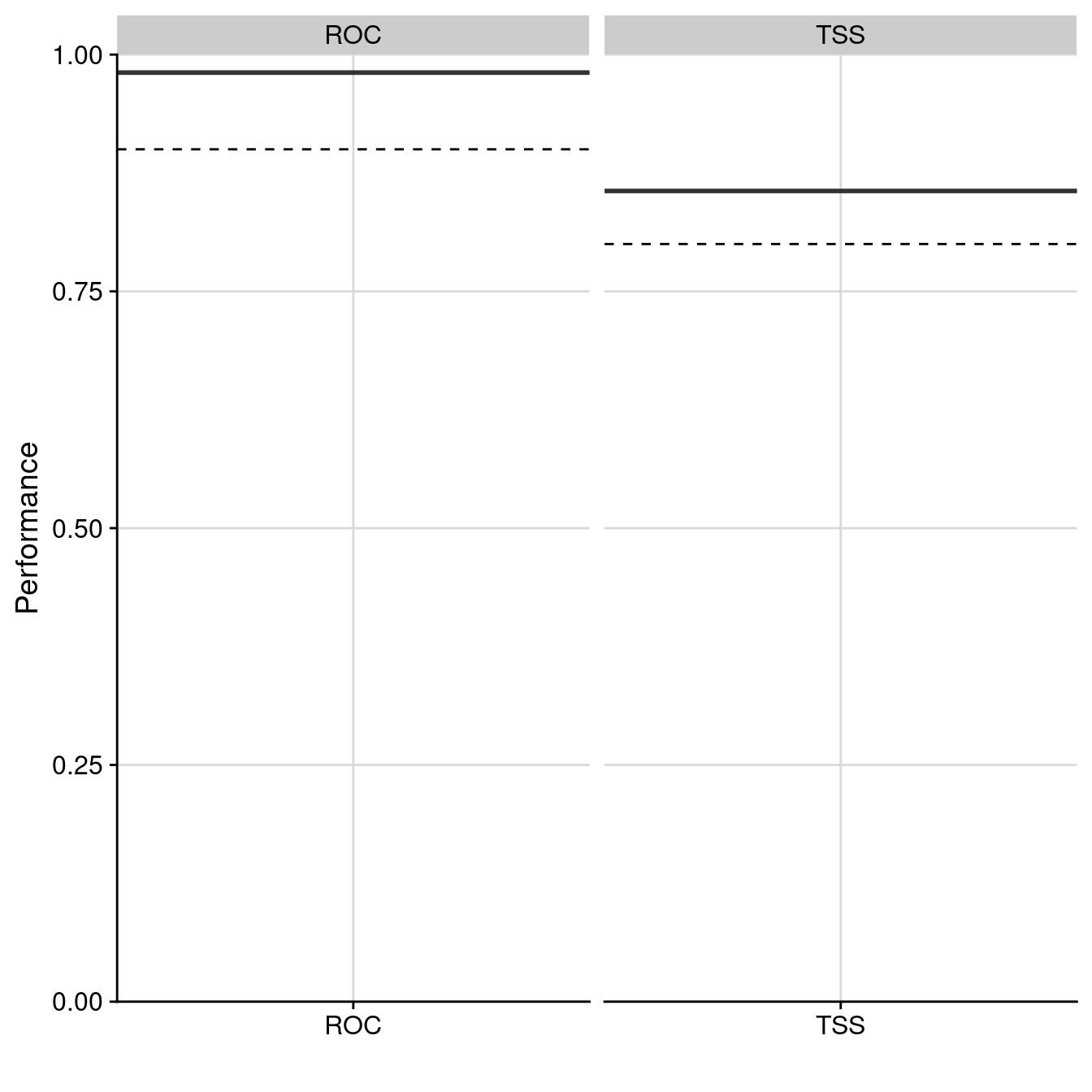
Figure 240.11: Évaluation de la qualité du modèle d’ensemble : Aire sous la courbe de ROC & True Skill Statistic.
full.name merged.by.PA merged.by.run merged.by.algo filtered.by
1 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
2 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
3 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
4 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
5 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
6 caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun mergedAlgo TSS
algo expl.var rand var.imp
1 EMcv temp_max_august 1 0.252239
2 EMcv temp_min 1 0.196424
3 EMcv temp_wet_quart 1 0.106642
4 EMcv temp_season 1 0.176006
5 EMcv prec_wet_quart 1 0.223041
6 EMcv prec_season 1 0.239735Par variable :
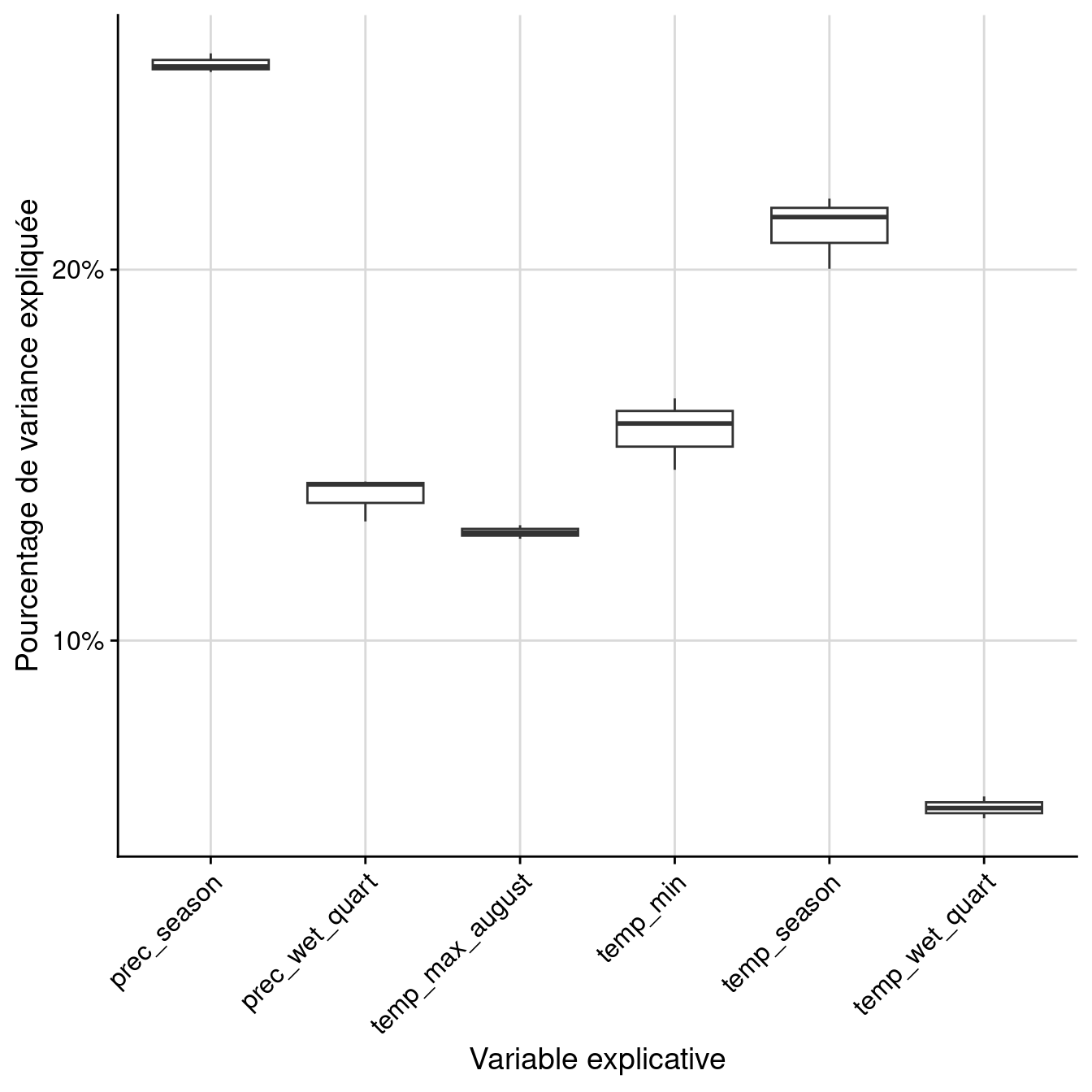
Figure 240.12: Pourcentage de variance expliquée par variable dans le modèle d’ensemble.
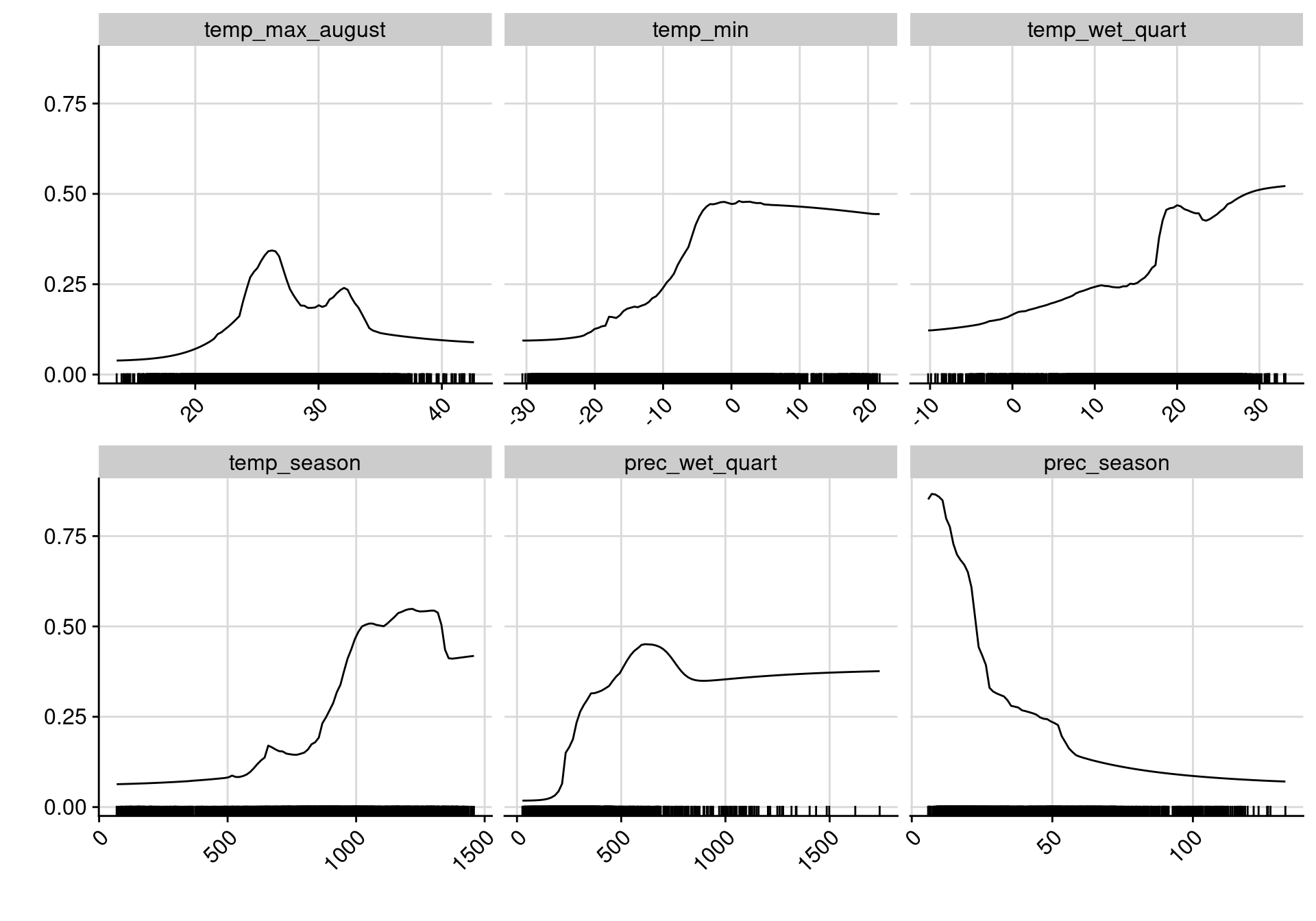
Figure 240.13: Courbes de réponse de chaque variable climatique dans le modèle d’ensemble.
240.3 Projections
240.3.1 Distribution potentielle contemporaine
Contemporaine : Cartographie des observations sur couches climatiques Extraction des conditions climatiques favorables
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/caco/current
sp.name : caco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/caco/caco.AllModels.models.out )
models.projected :
caco_PA1_RUN1_GAM, caco_PA1_RUN1_MARS, caco_PA1_RUN1_MAXNET, caco_PA1_RUN1_GBM, caco_PA1_RUN1_RF, caco_PA2_RUN1_GAM, caco_PA2_RUN1_MARS, caco_PA2_RUN1_MAXNET, caco_PA2_RUN1_GBM, caco_PA2_RUN1_RF, caco_PA3_RUN1_GAM, caco_PA3_RUN1_MARS, caco_PA3_RUN1_MAXNET, caco_PA3_RUN1_GBM, caco_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=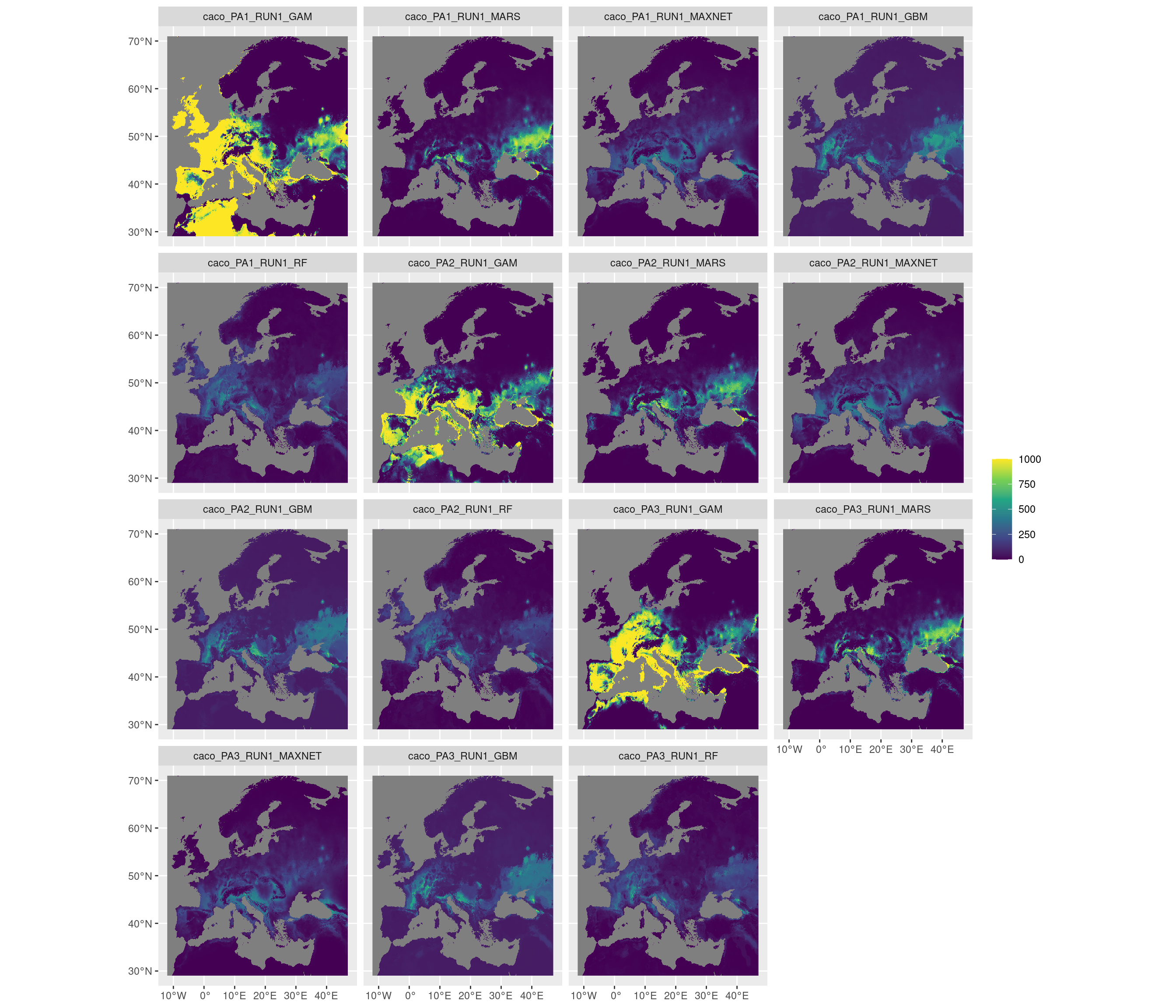
Figure 240.14: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
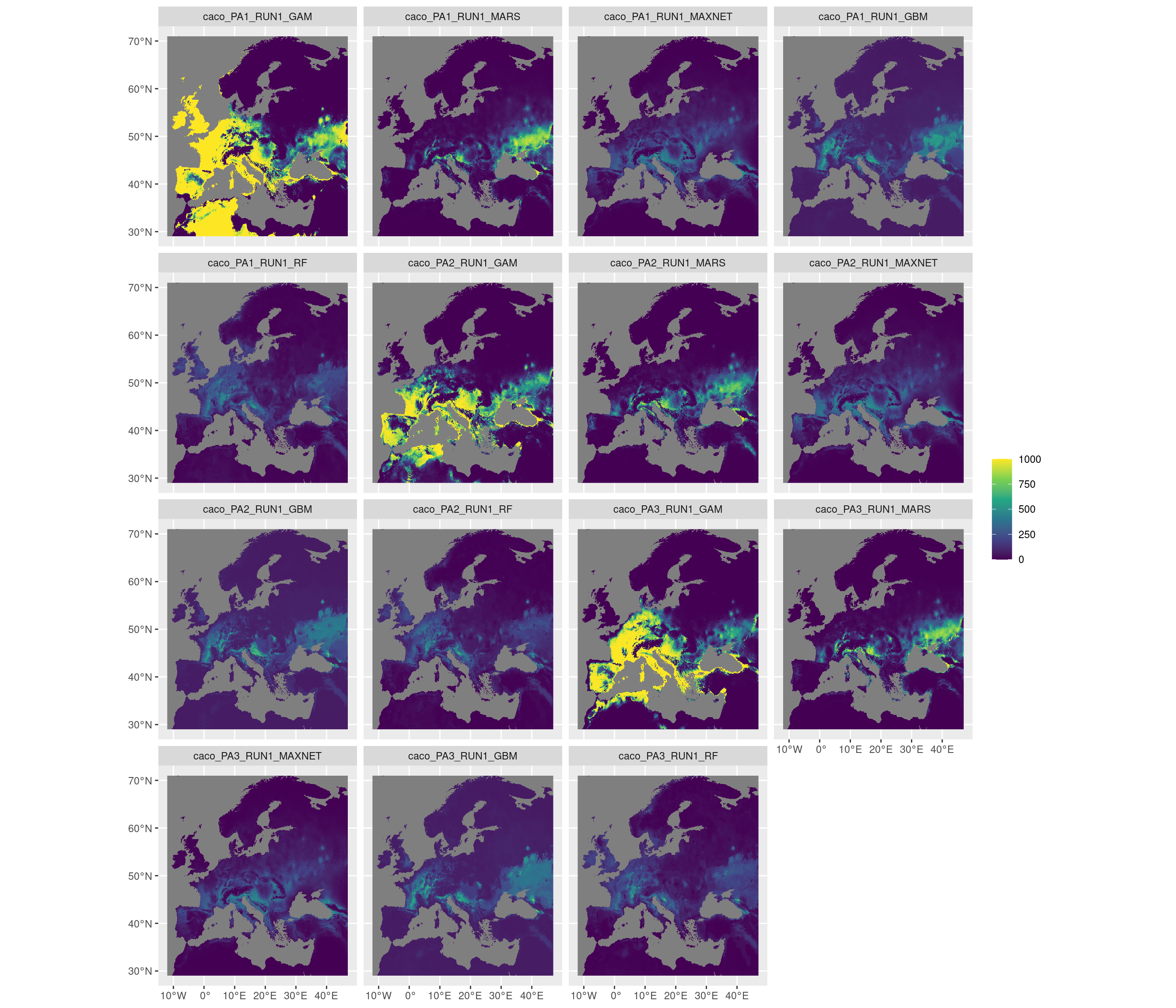
Figure 240.15: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/caco/current
sp.name : caco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/caco/caco.AllModels.ensemble.models.out )
models.projected :
caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, caco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=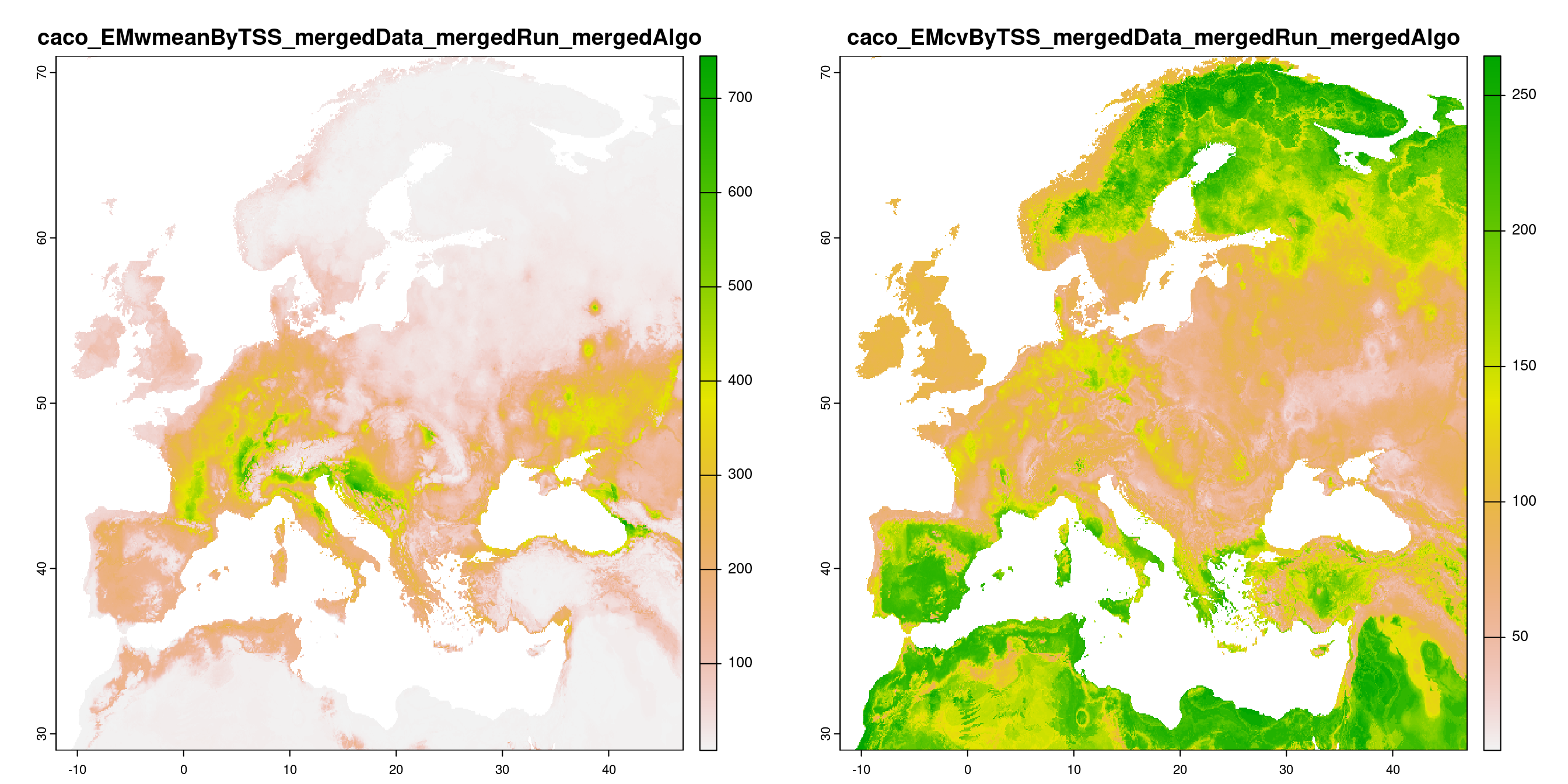
Figure 240.16: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) ; B) Incertitude associée à la projection.
240.3.2 Interpolation à l’échelle de la métropole
- → conditions climatiques favorables à fine échelle
On projette la niche climatique sur ce raster à fine échelle.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/caco/cont_gre
sp.name : caco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/caco/caco.AllModels.models.out )
models.projected :
caco_PA1_RUN1_GAM, caco_PA1_RUN1_MARS, caco_PA1_RUN1_MAXNET, caco_PA1_RUN1_GBM, caco_PA1_RUN1_RF, caco_PA2_RUN1_GAM, caco_PA2_RUN1_MARS, caco_PA2_RUN1_MAXNET, caco_PA2_RUN1_GBM, caco_PA2_RUN1_RF, caco_PA3_RUN1_GAM, caco_PA3_RUN1_MARS, caco_PA3_RUN1_MAXNET, caco_PA3_RUN1_GBM, caco_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=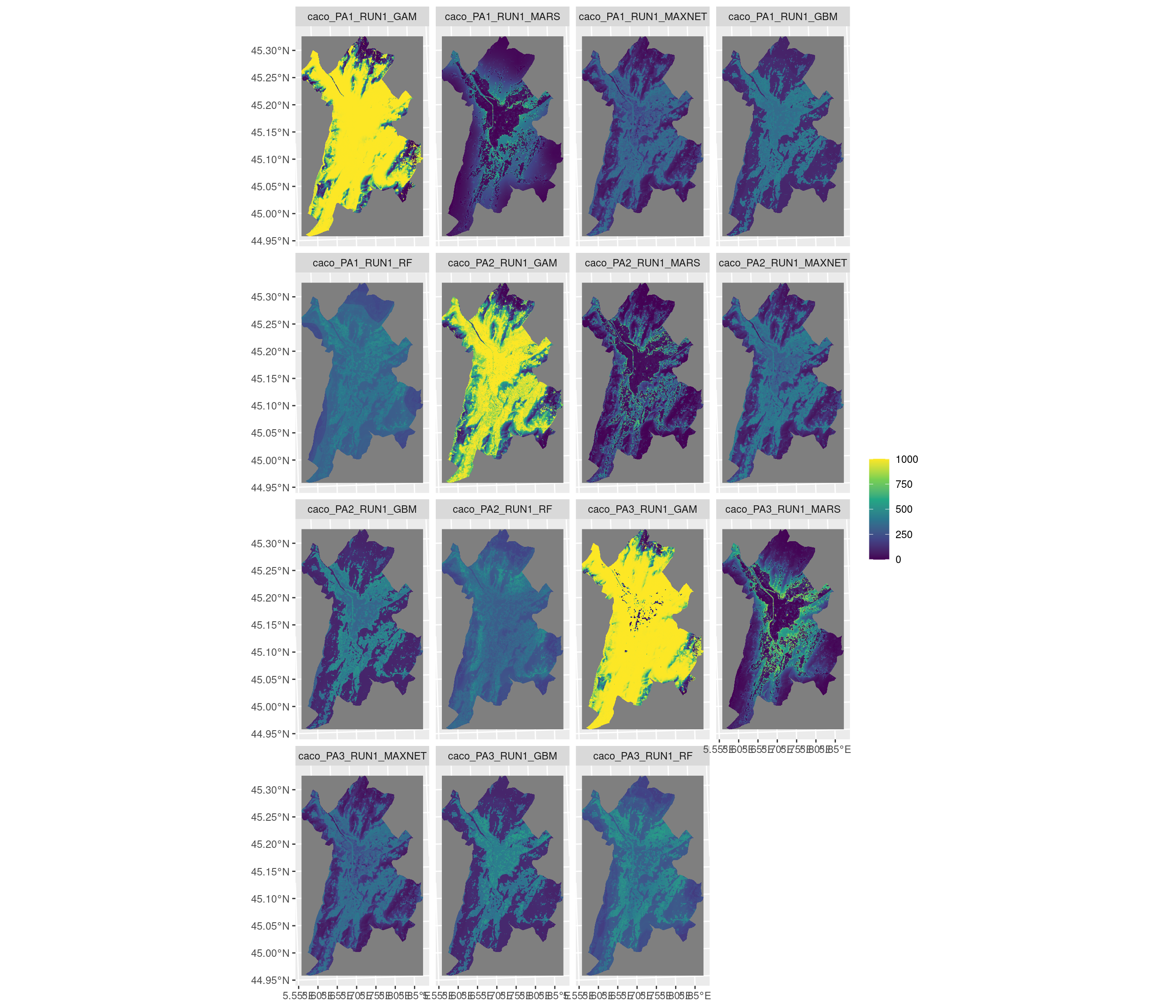
Figure 240.17: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
Figure 240.18: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/caco/cont_gre
sp.name : caco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season
modeling.id : AllModels ( Output/caco/caco.AllModels.ensemble.models.out )
models.projected :
caco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, caco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=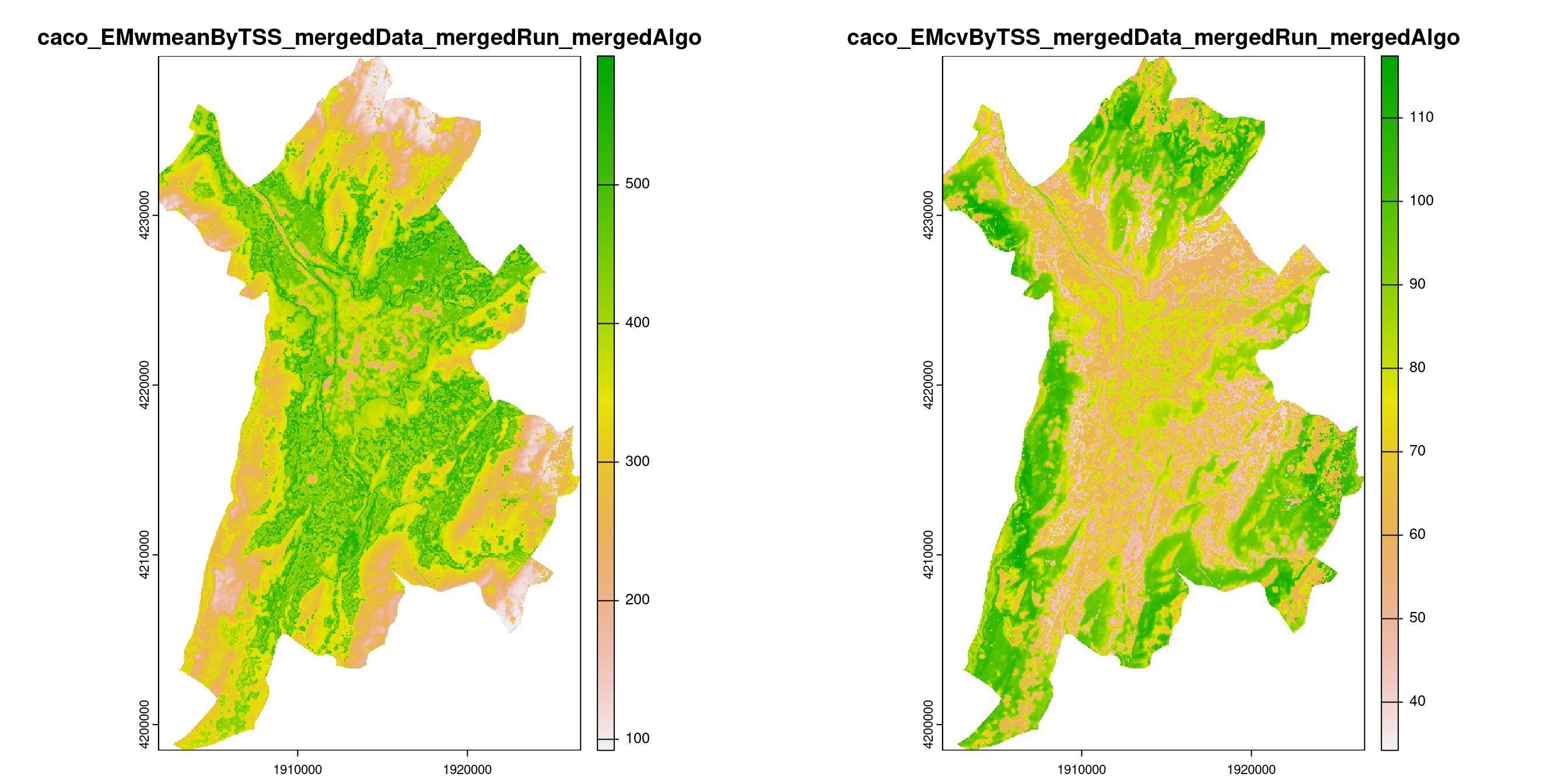
Figure 240.19: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) sur la métropole de Grenoble ; B) Incertitude associée à la projection.
240.3.3 Distributions potentielles futures
Futur : Projection à l’échelle de l’Europe jusqu’à l’horizon 2100
Toutes les fenêtres temporelles × scénarios pour le GCM « IPSL-CM6A-LR »
- SSP1-2.6
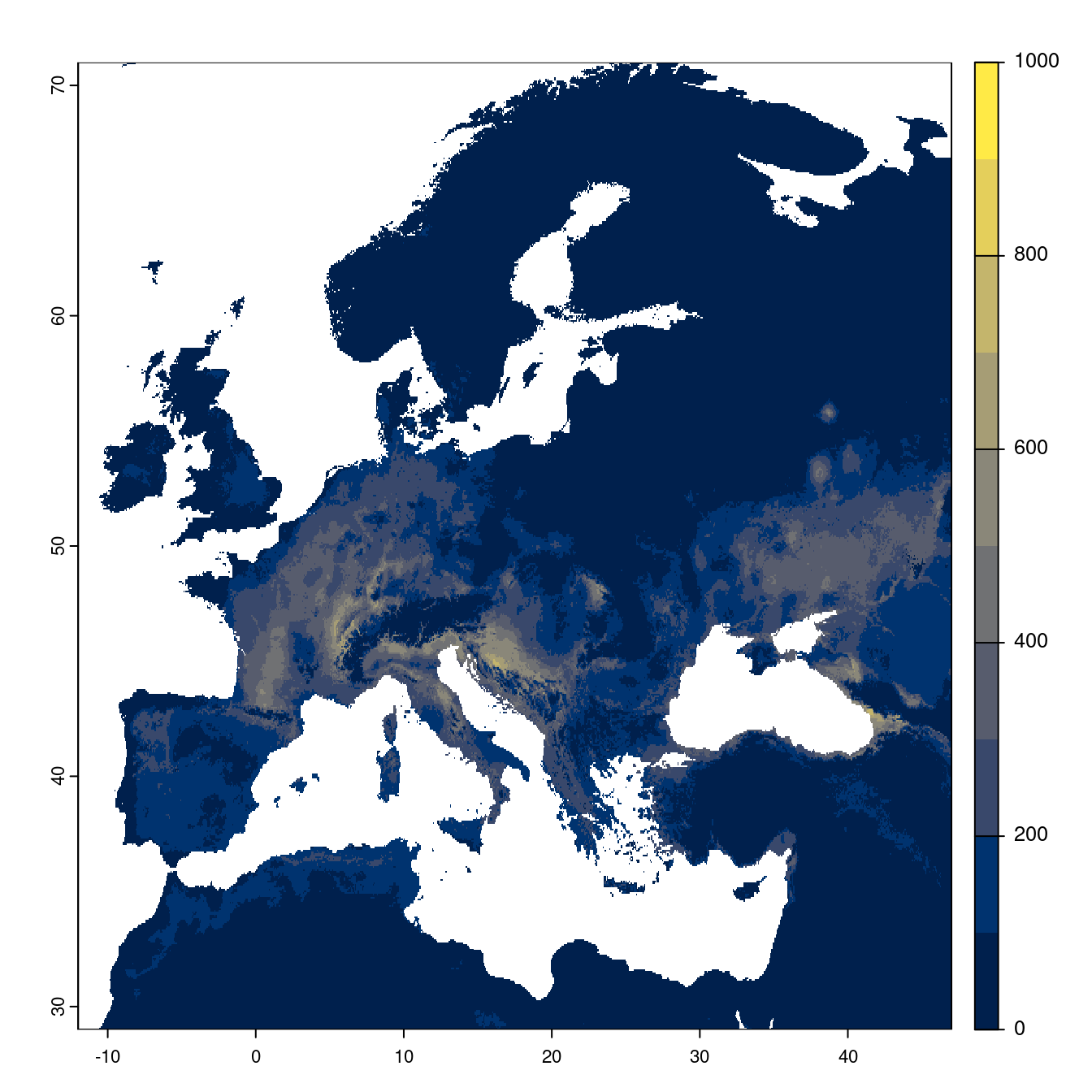
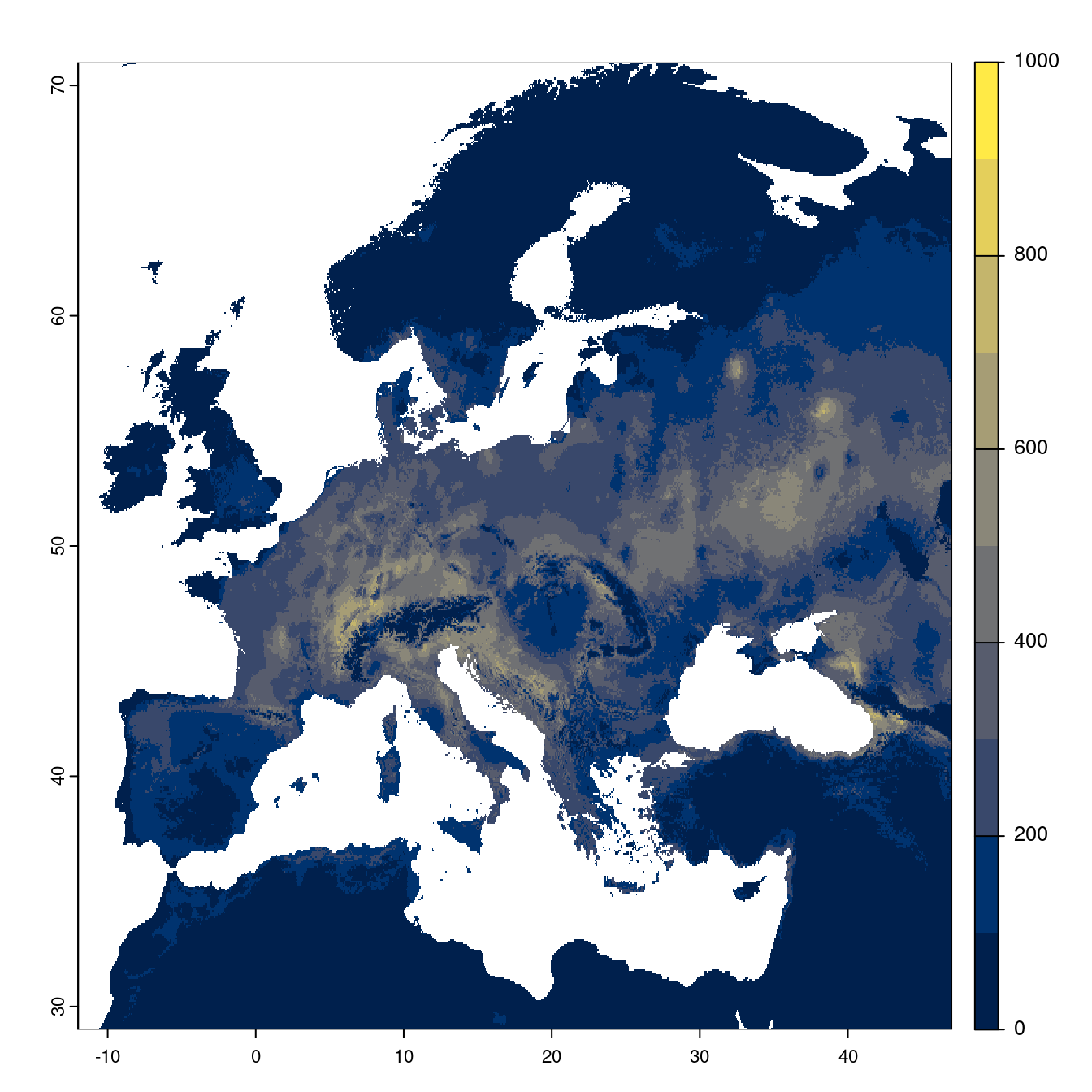
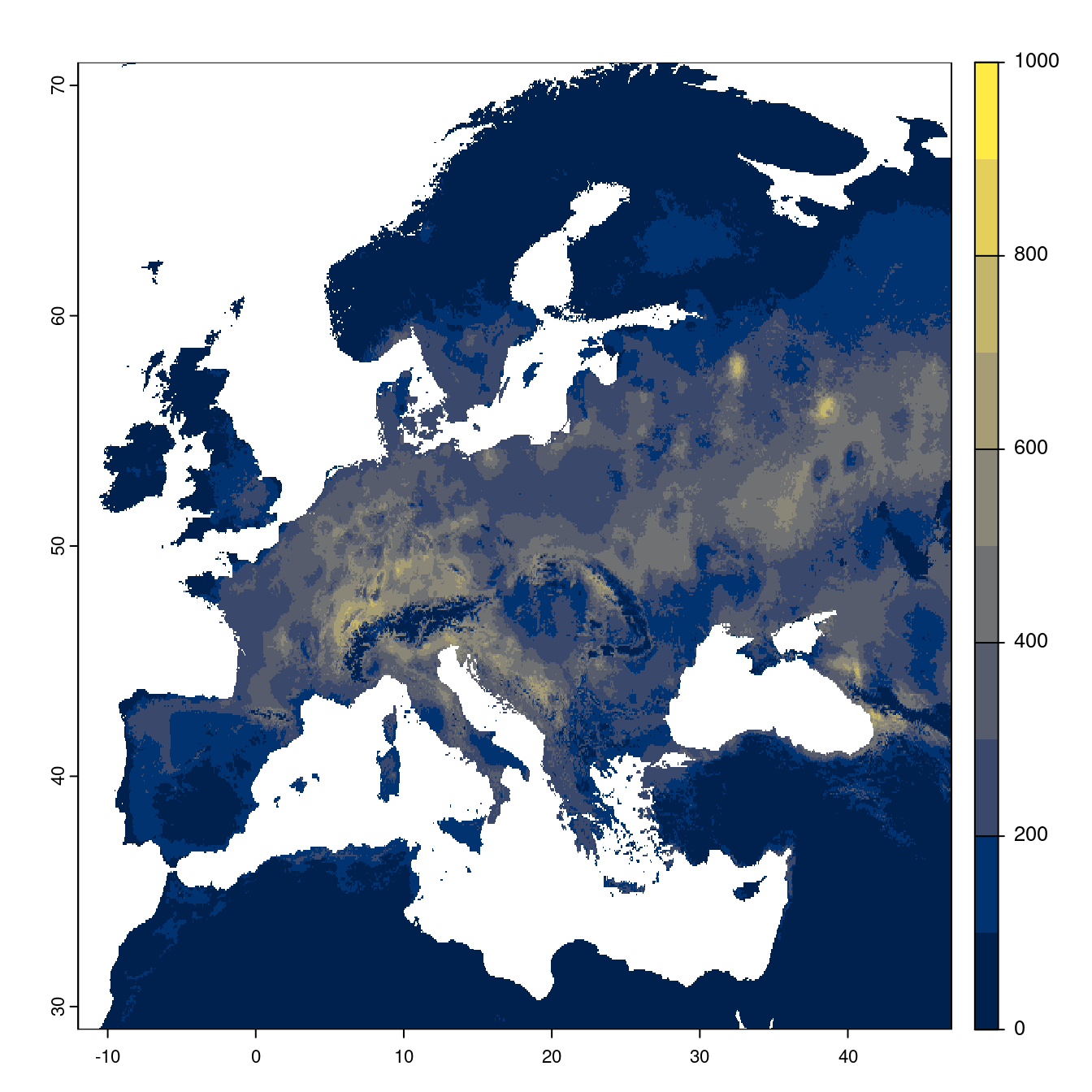
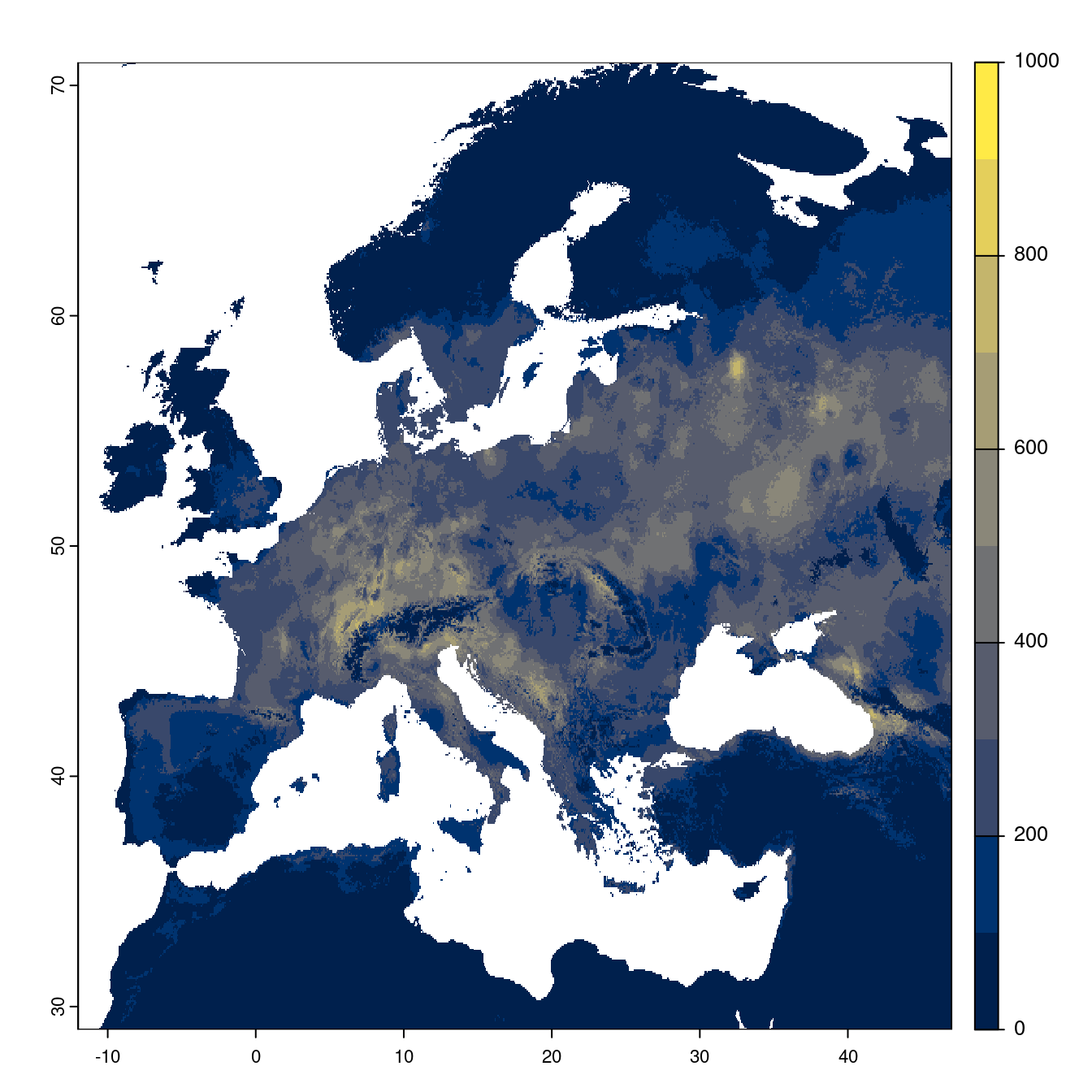
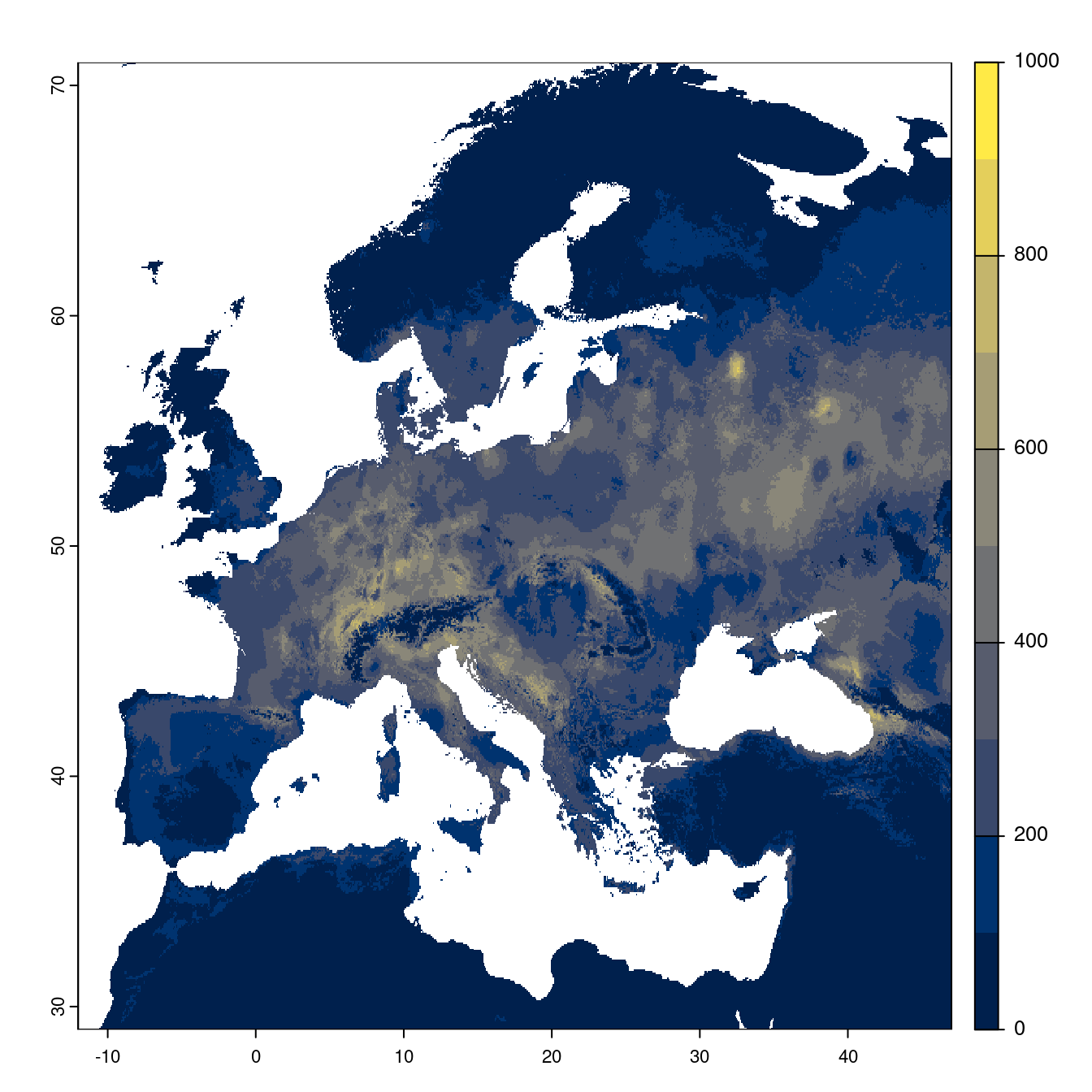
Figure 240.20: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP1-2.6 (modèle d’ensemble).
On obtient pour la métrique TSS une carte unique de moyenne pondérée sur les algorithmes et sur les jeux de pseudo-absences.
- SSP2-4.5
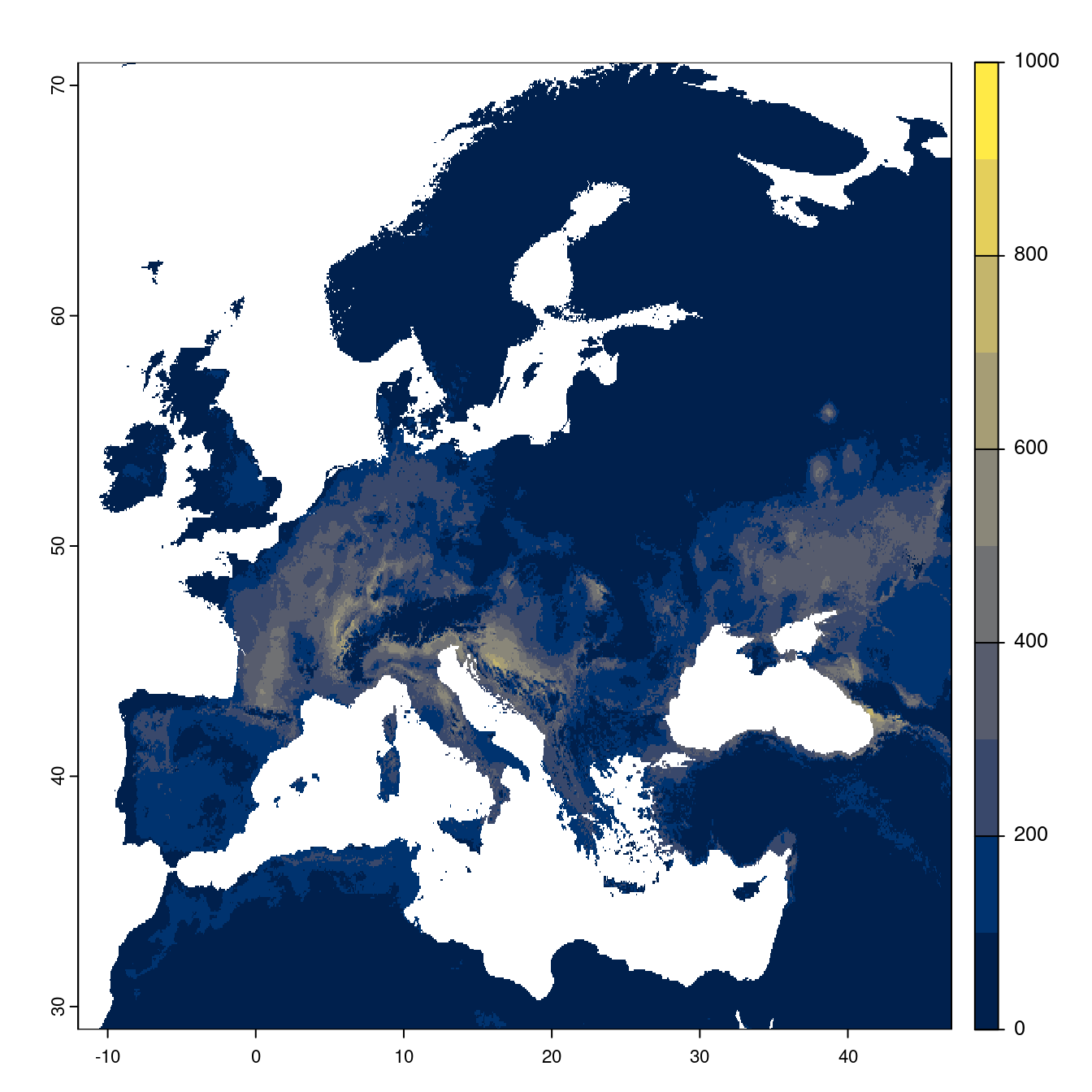
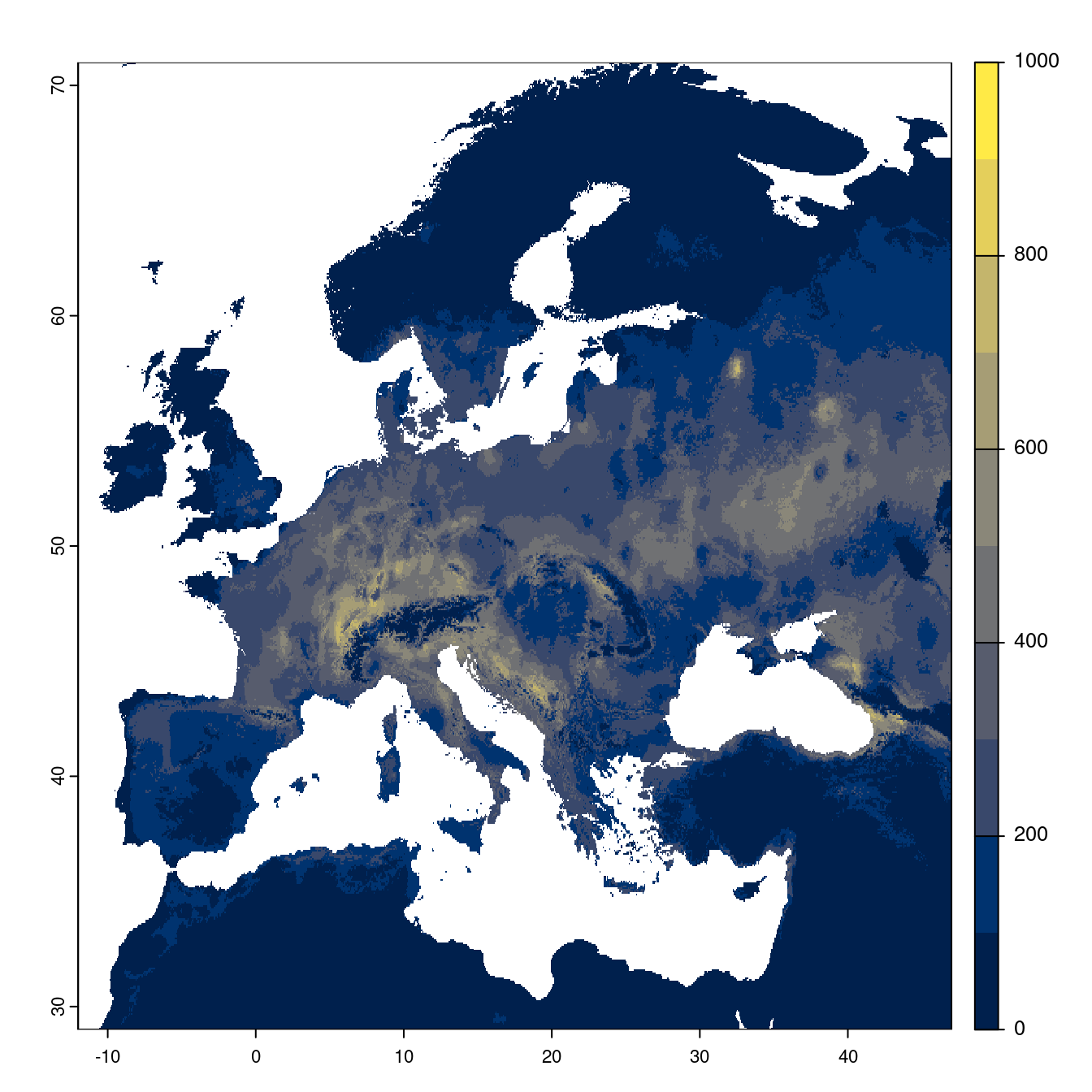
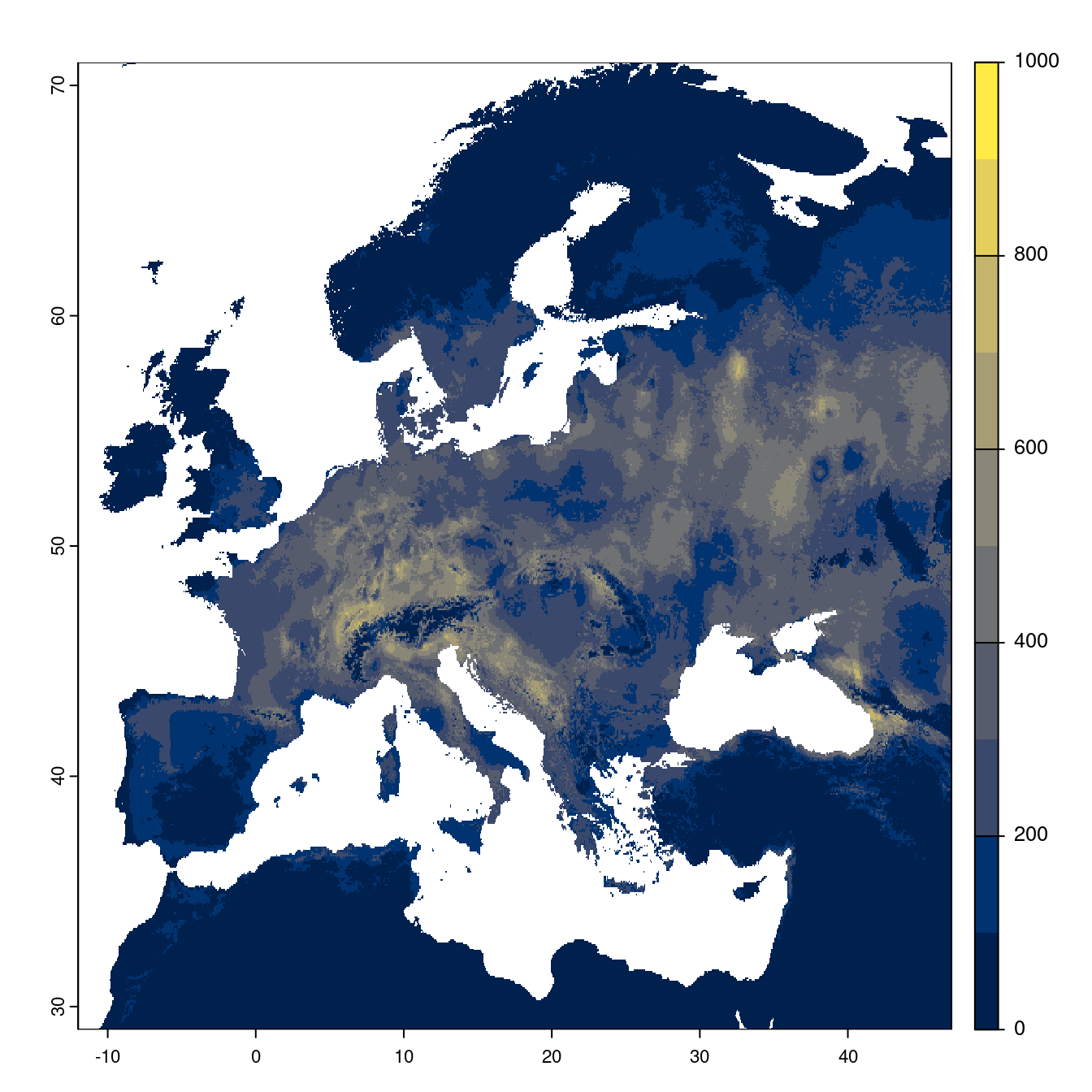
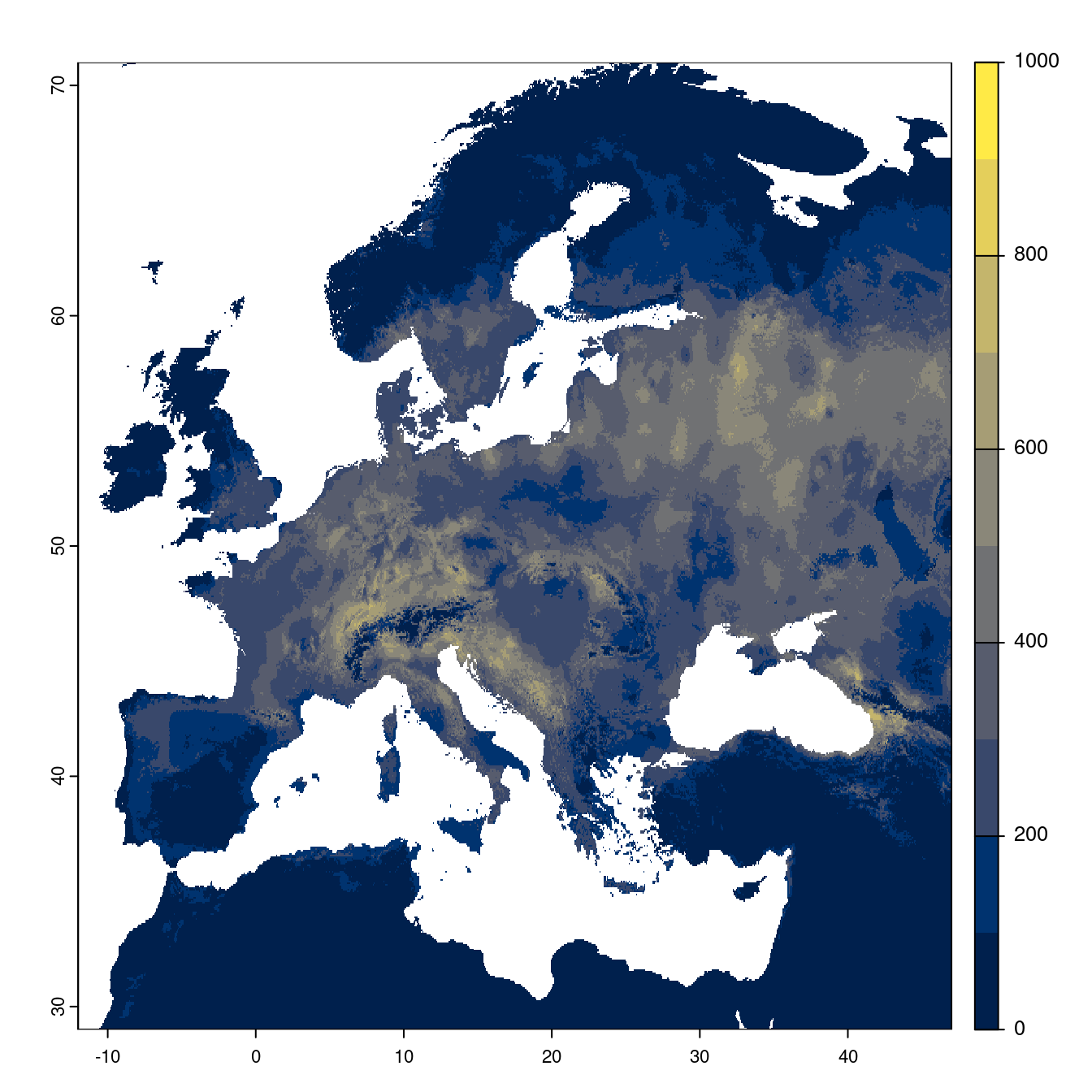
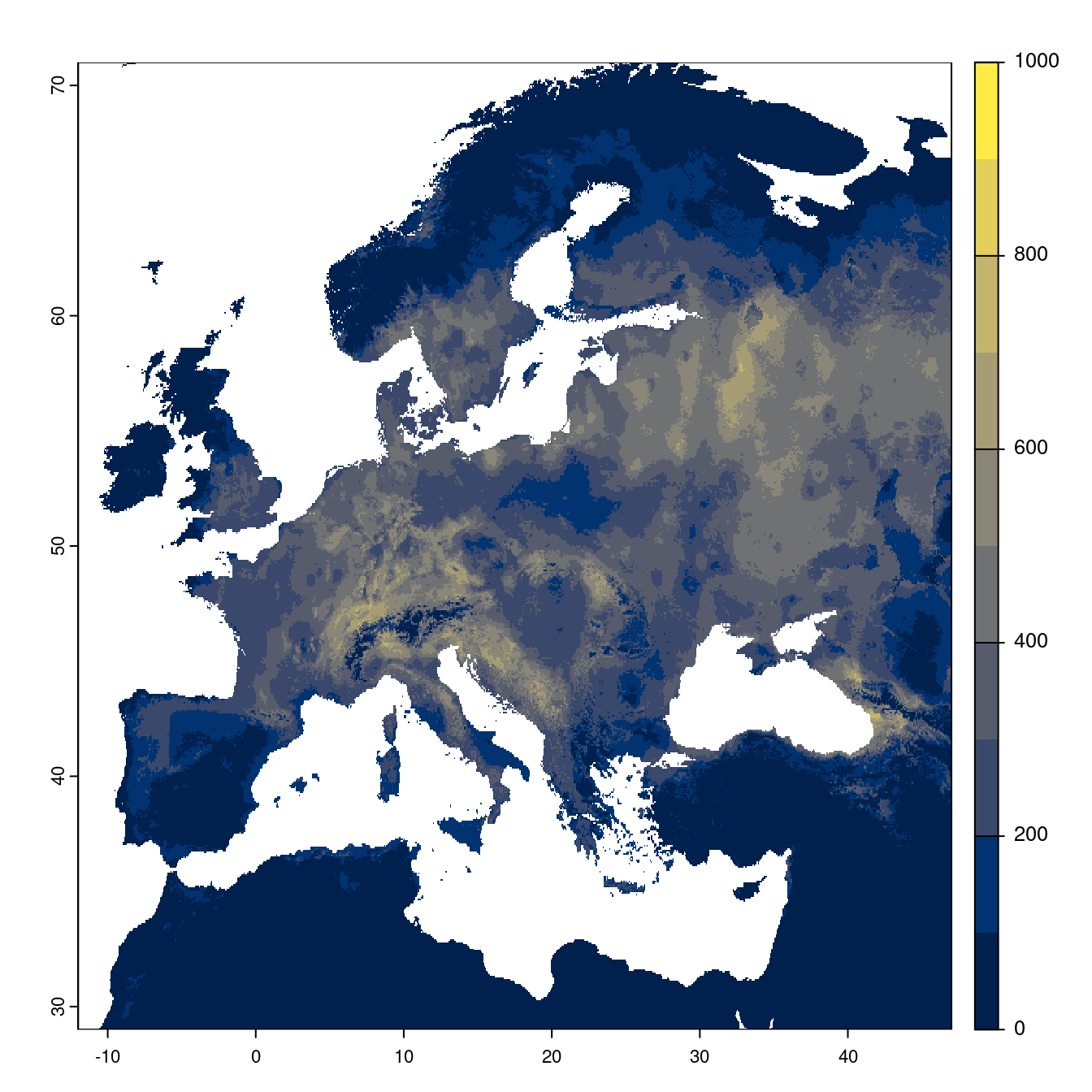
Figure 240.21: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP2-4.5 (modèle d’ensemble).
- SSP3-7.0
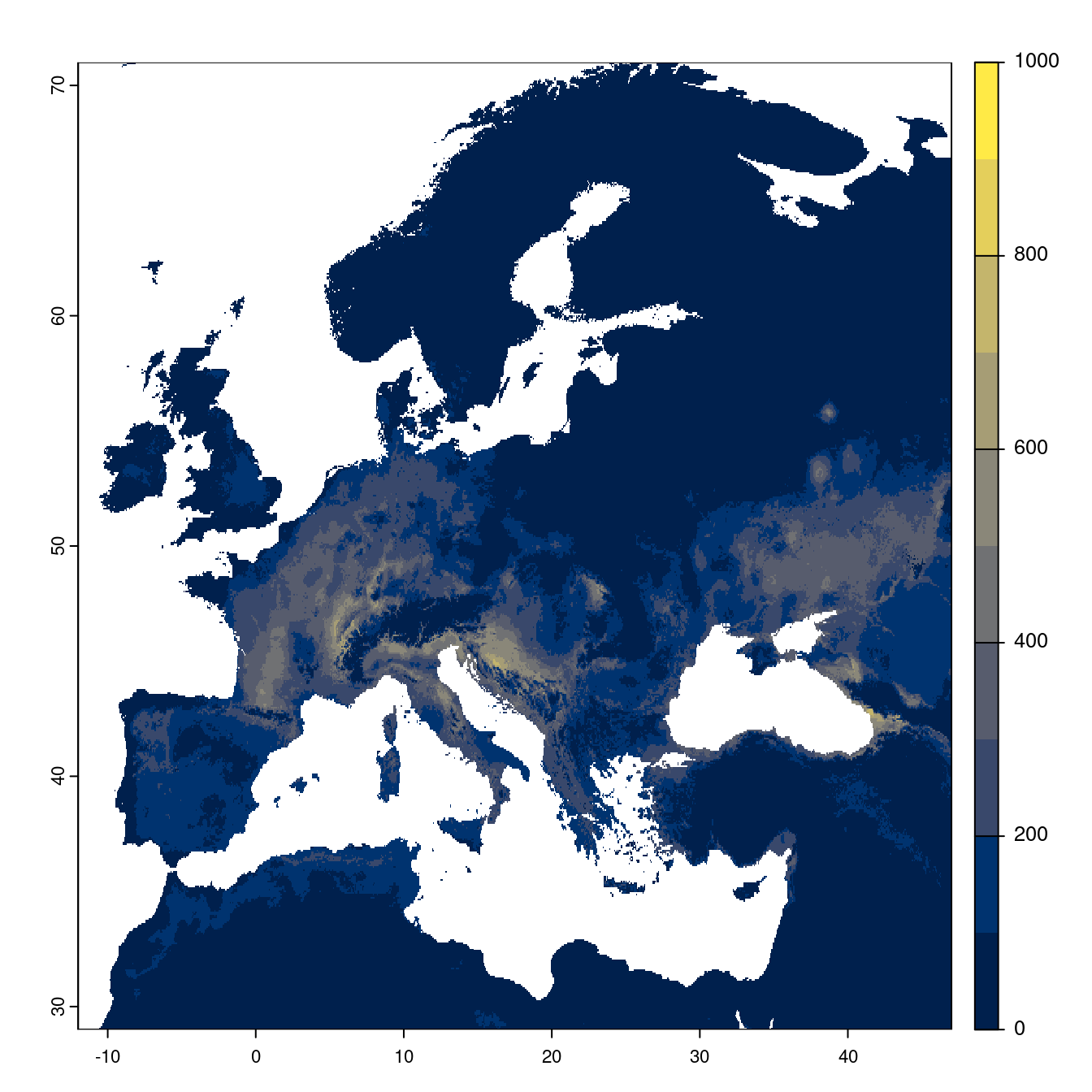
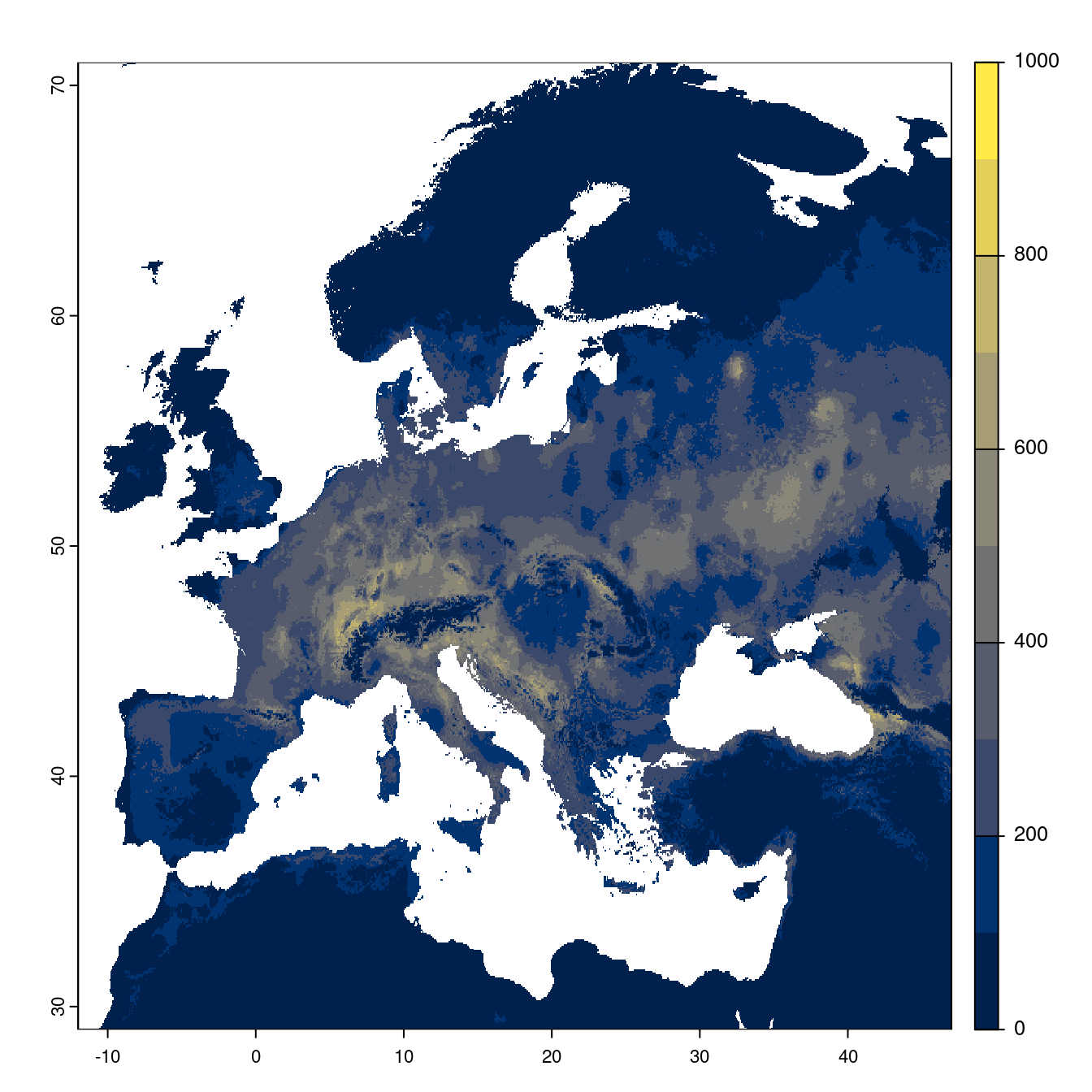
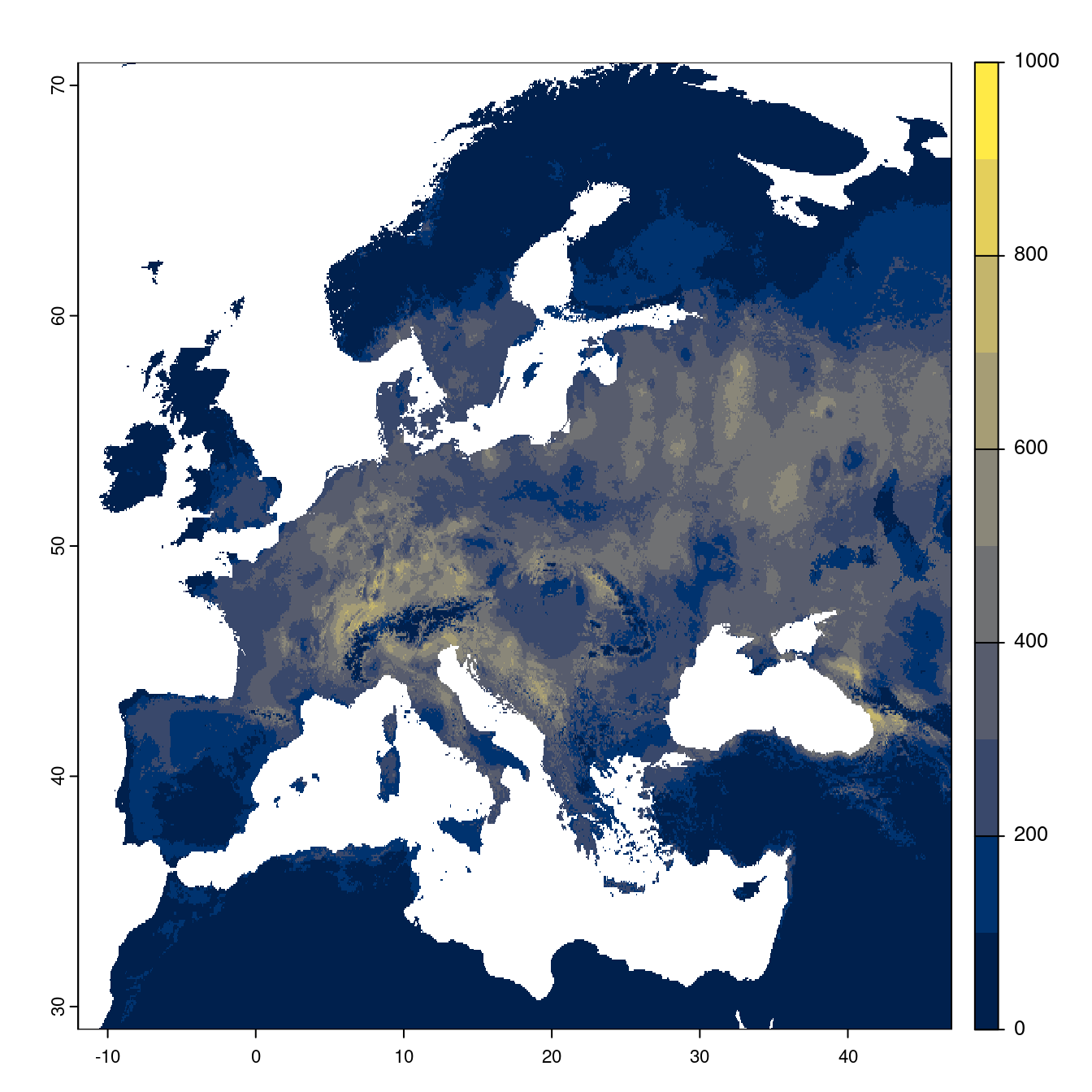
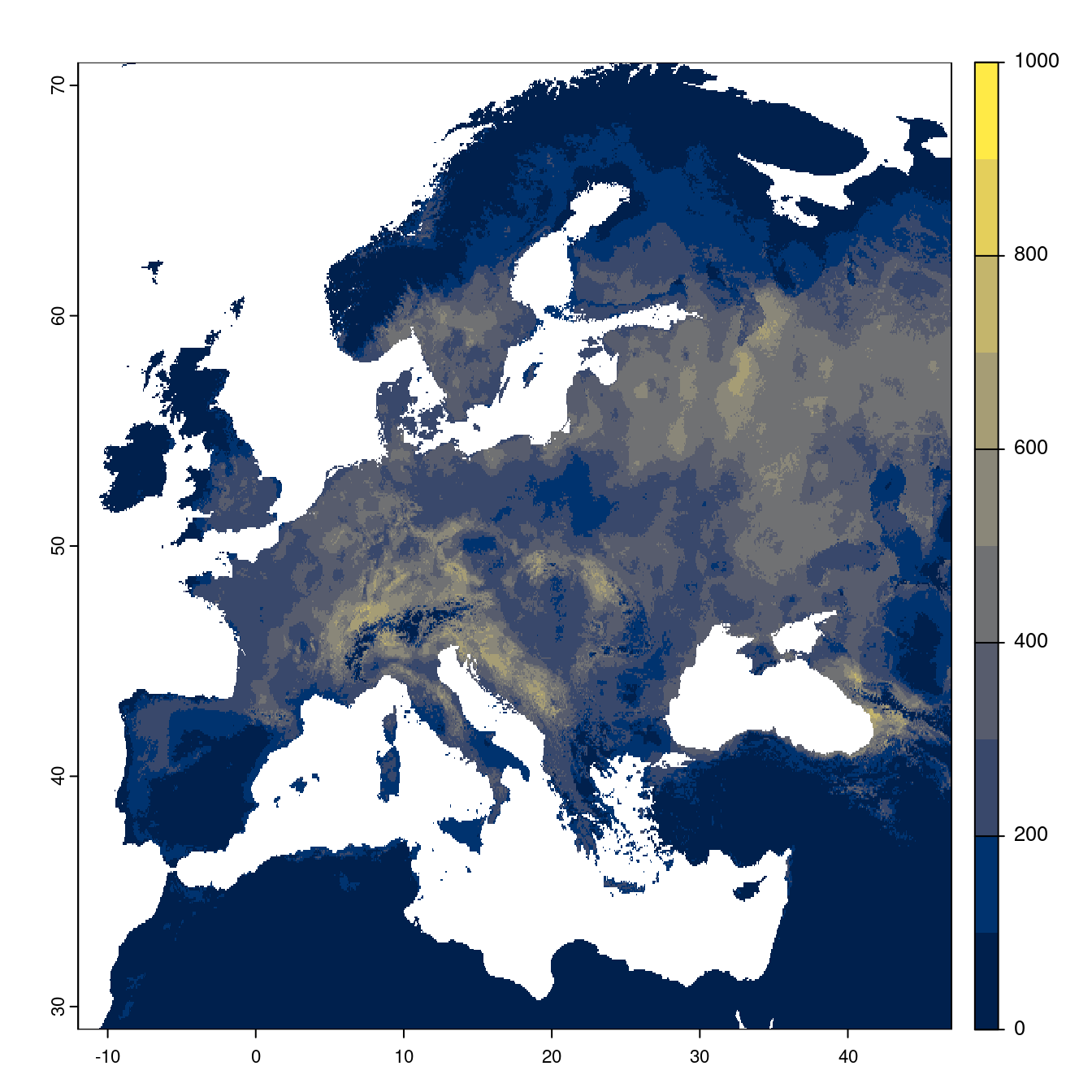
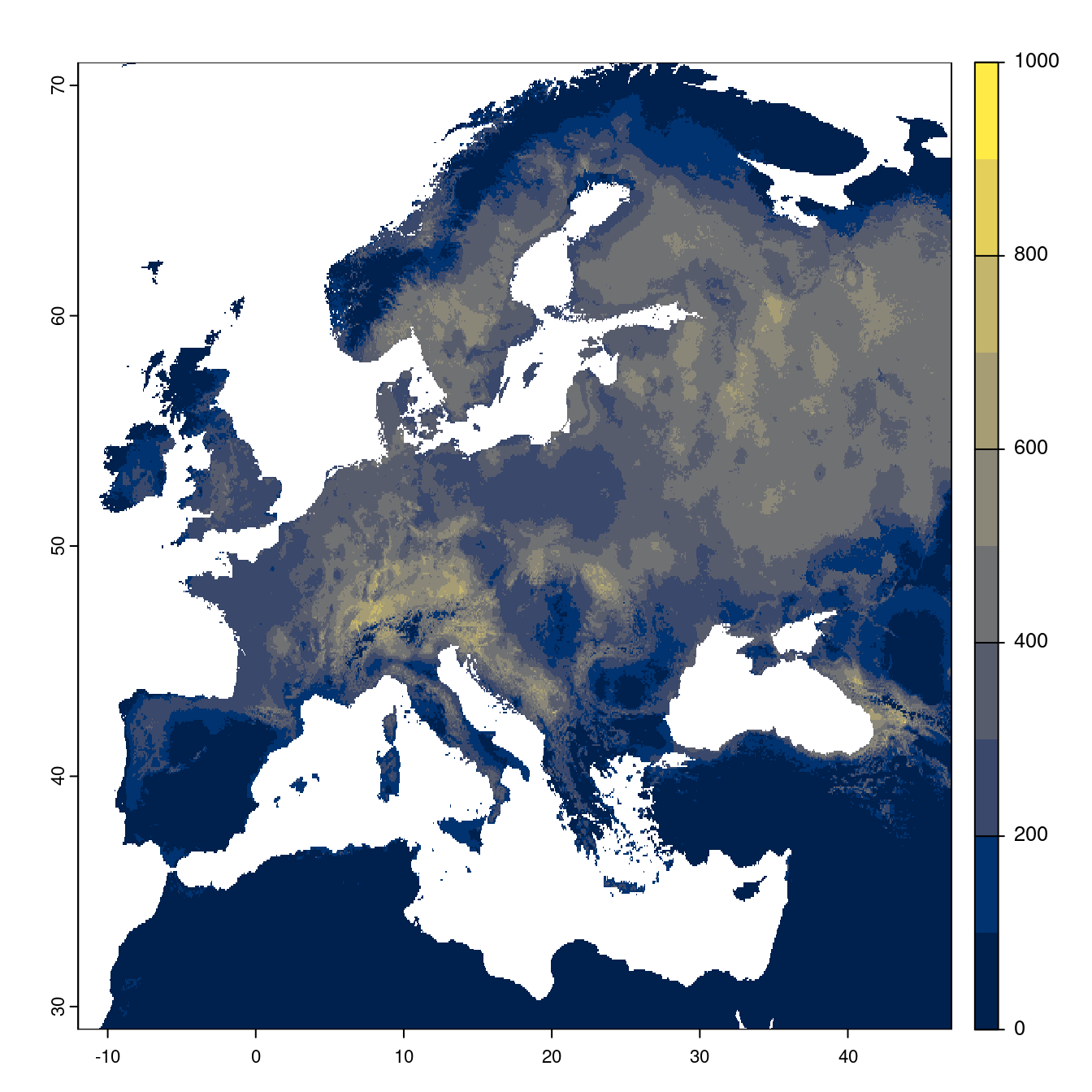
Figure 240.22: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP3-7.0 (modèle d’ensemble).
- SSP5-8.5
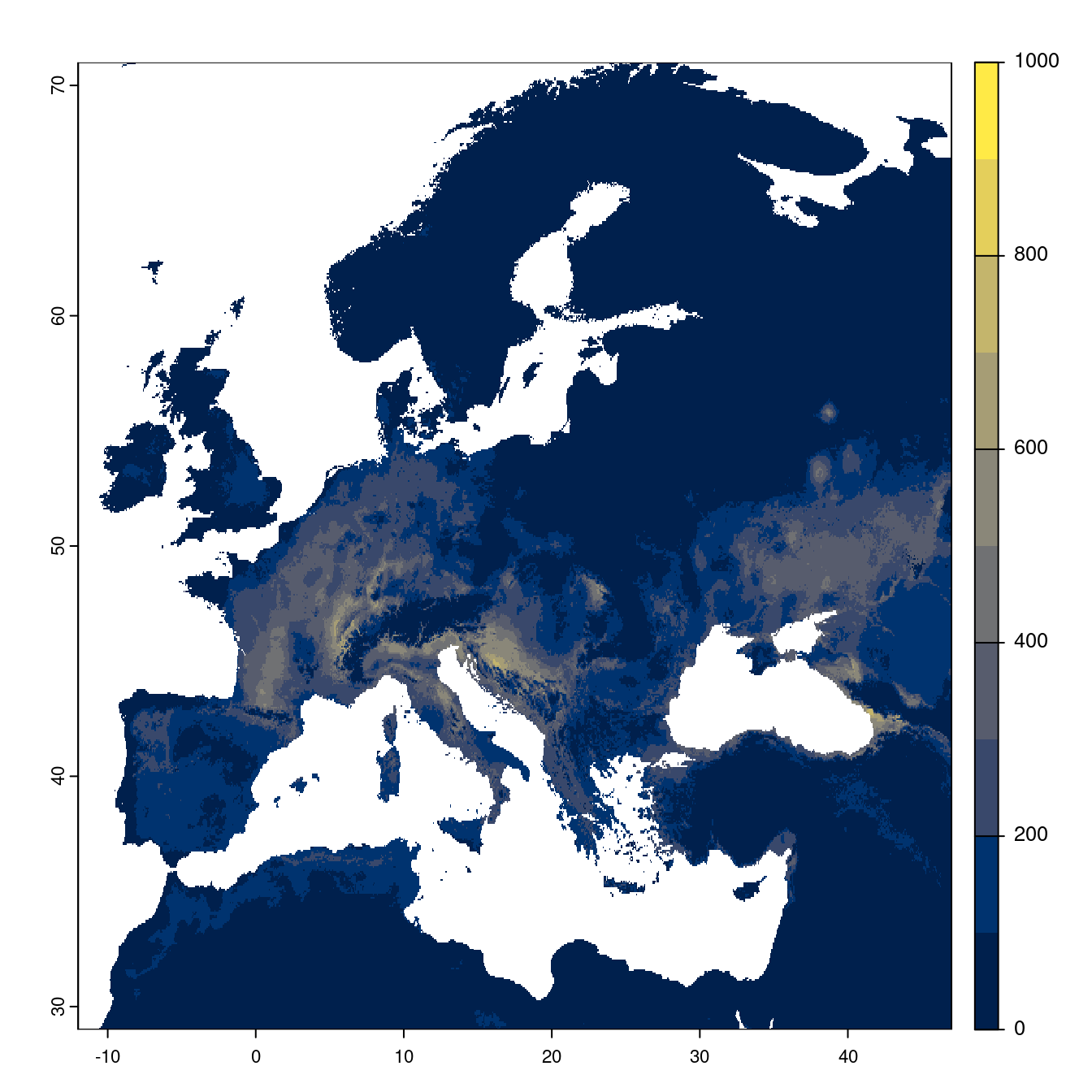
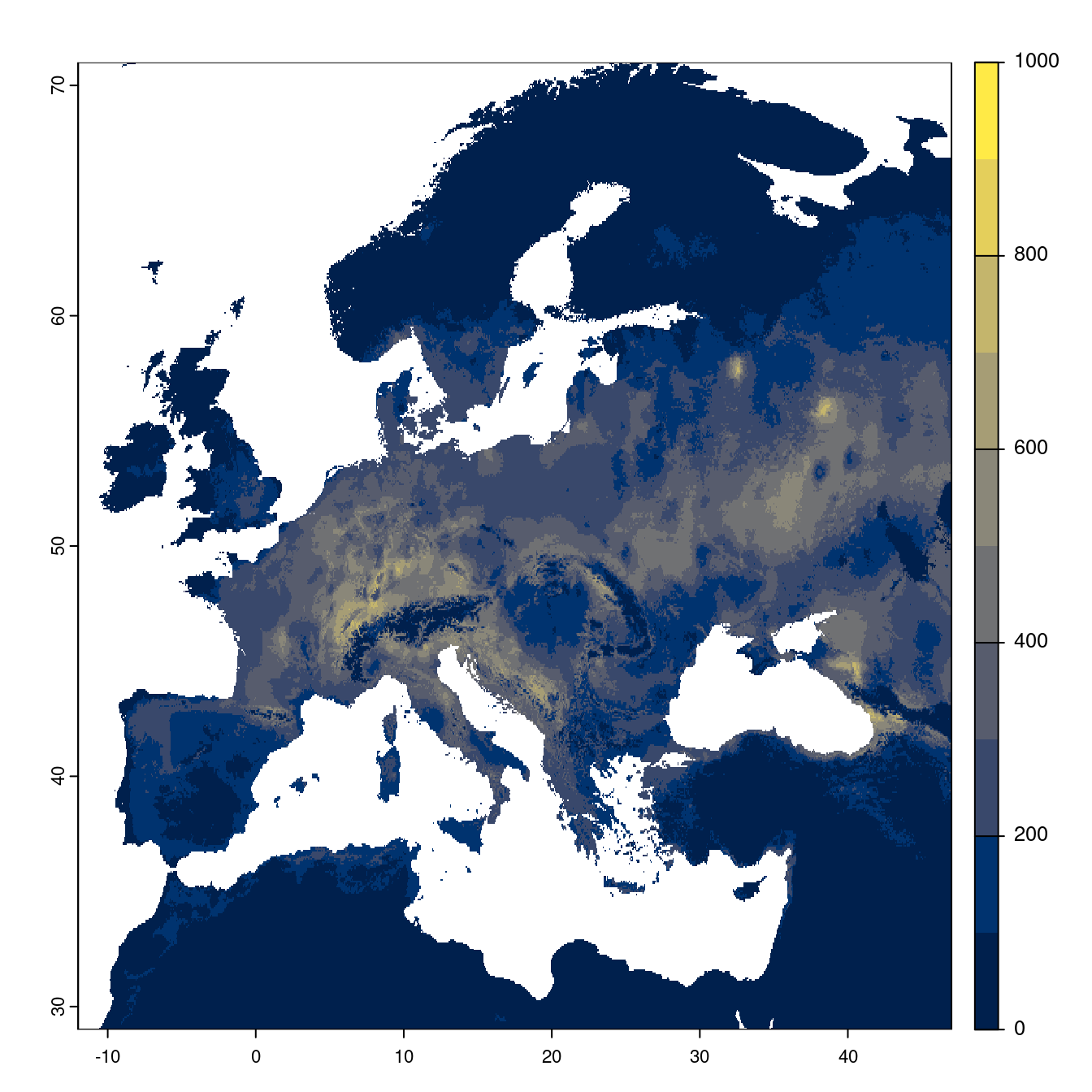
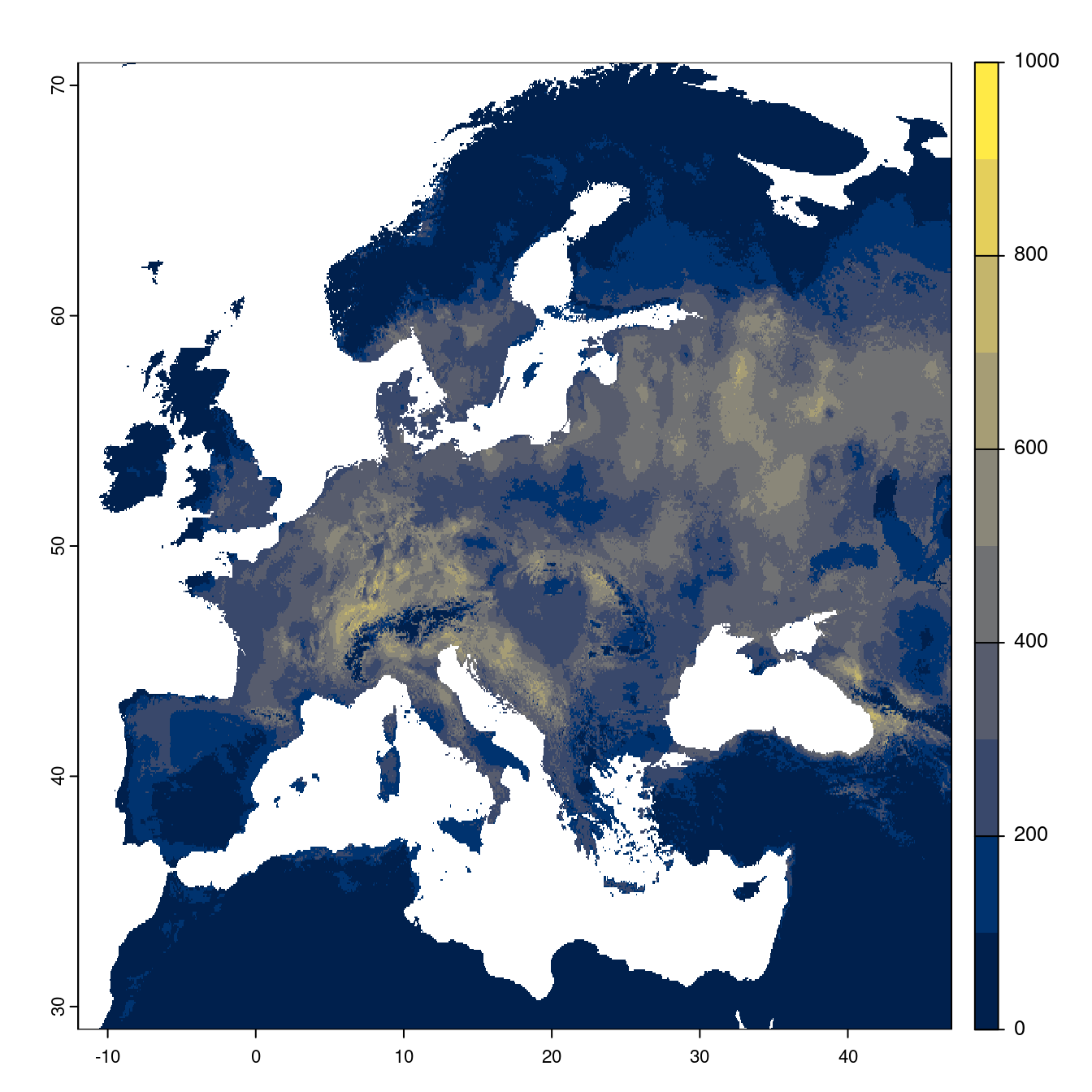
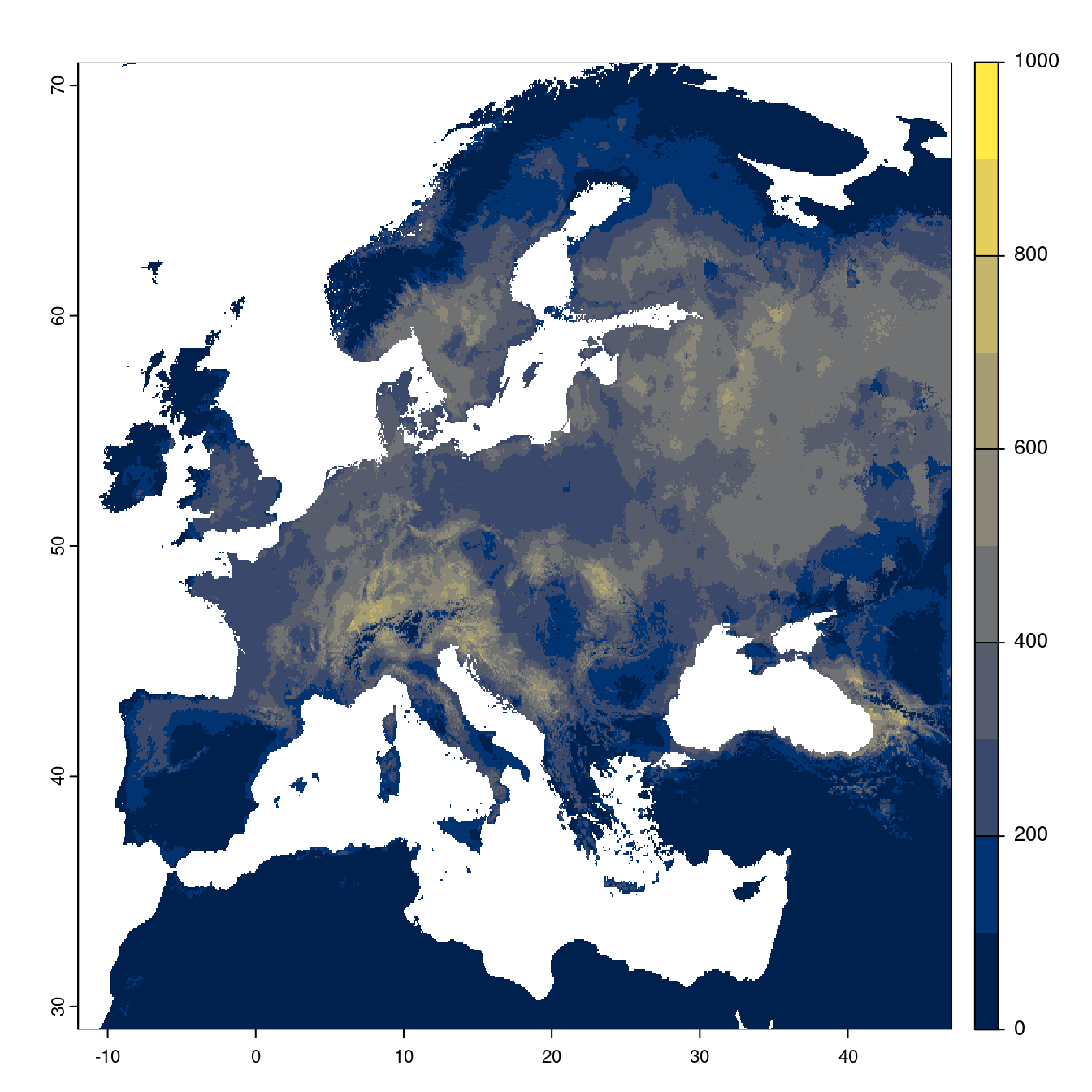
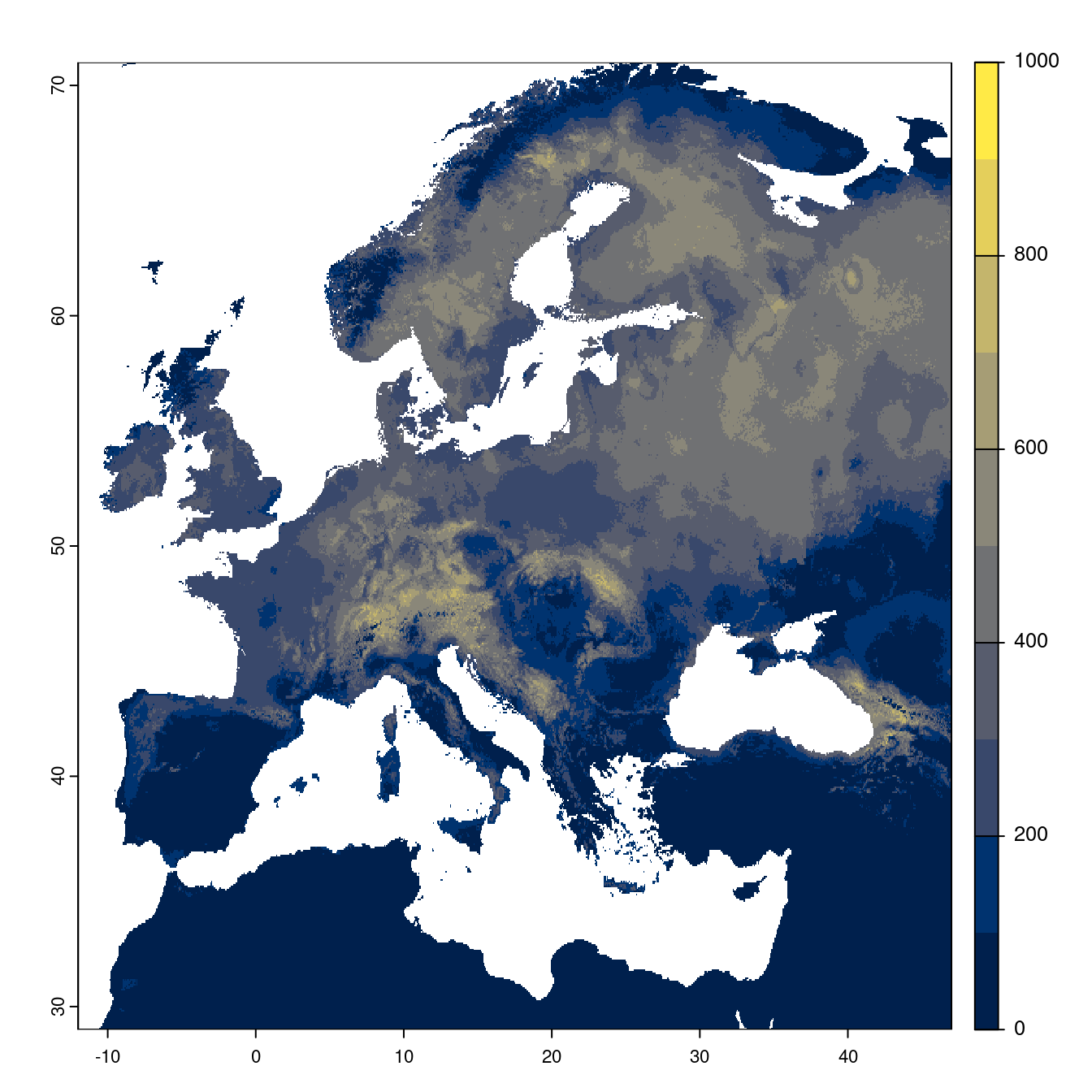
Figure 240.23: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble).
240.4 Inférence à l’échelle de la métropole
- Croisement avec températures de surface 2019 (fournie par la métropole)
- → conditions climatiques favorables à fine échelle
- à peu près 7 pixels × 9 (WorldClim)
240.4.1 Distributions potentielles futures
Même chose que précédemment mais sur les rasters de Grenoble
- SSP1-2.6
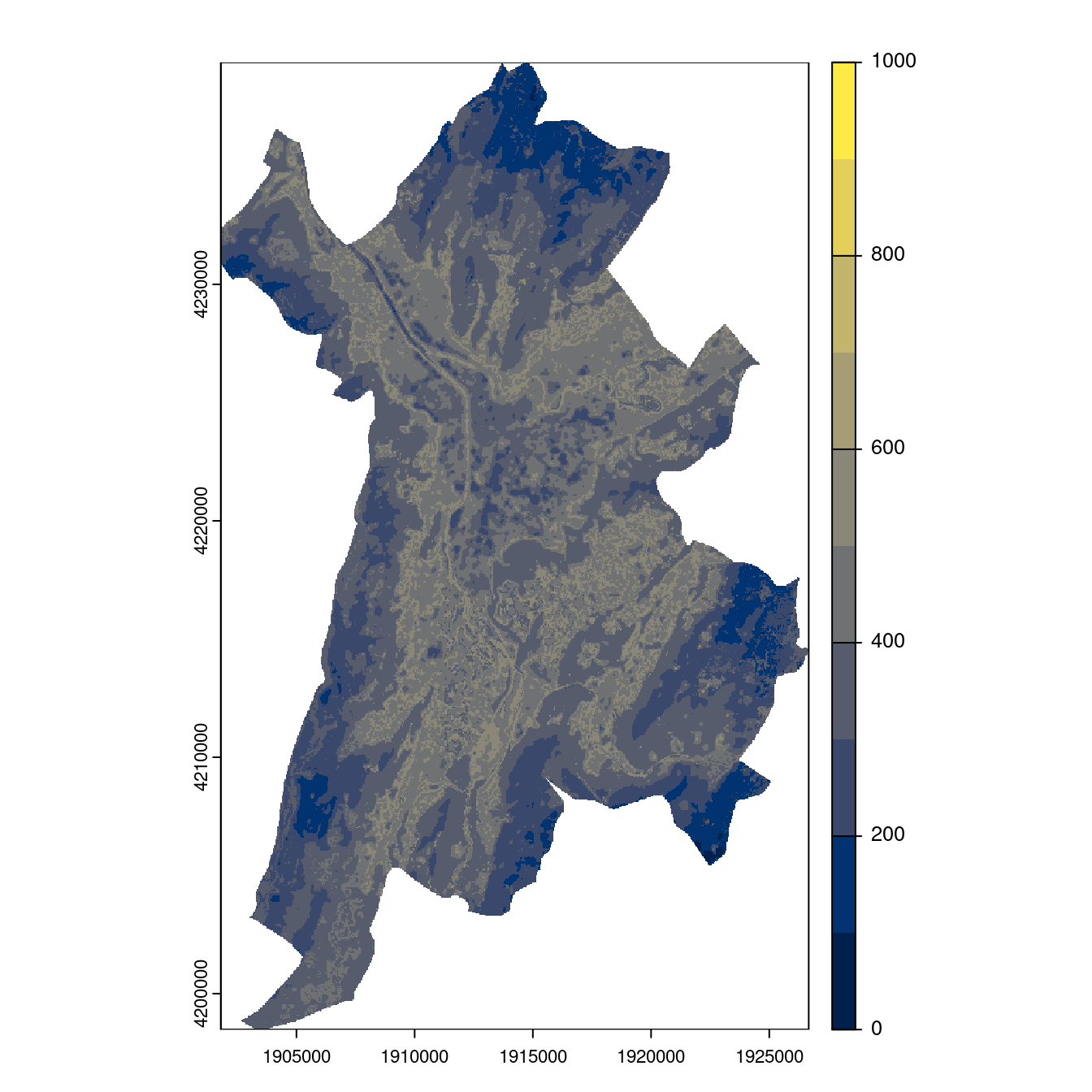
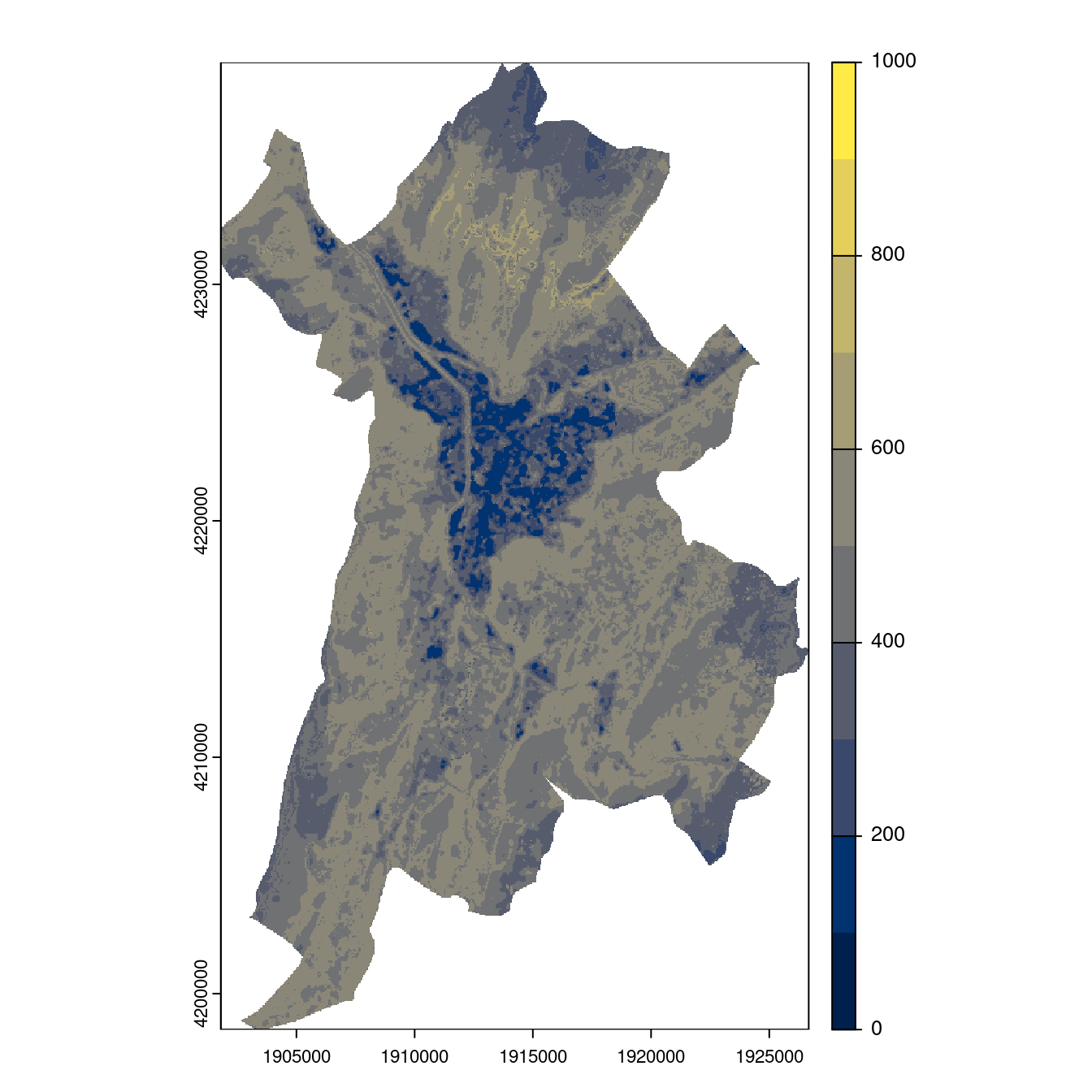
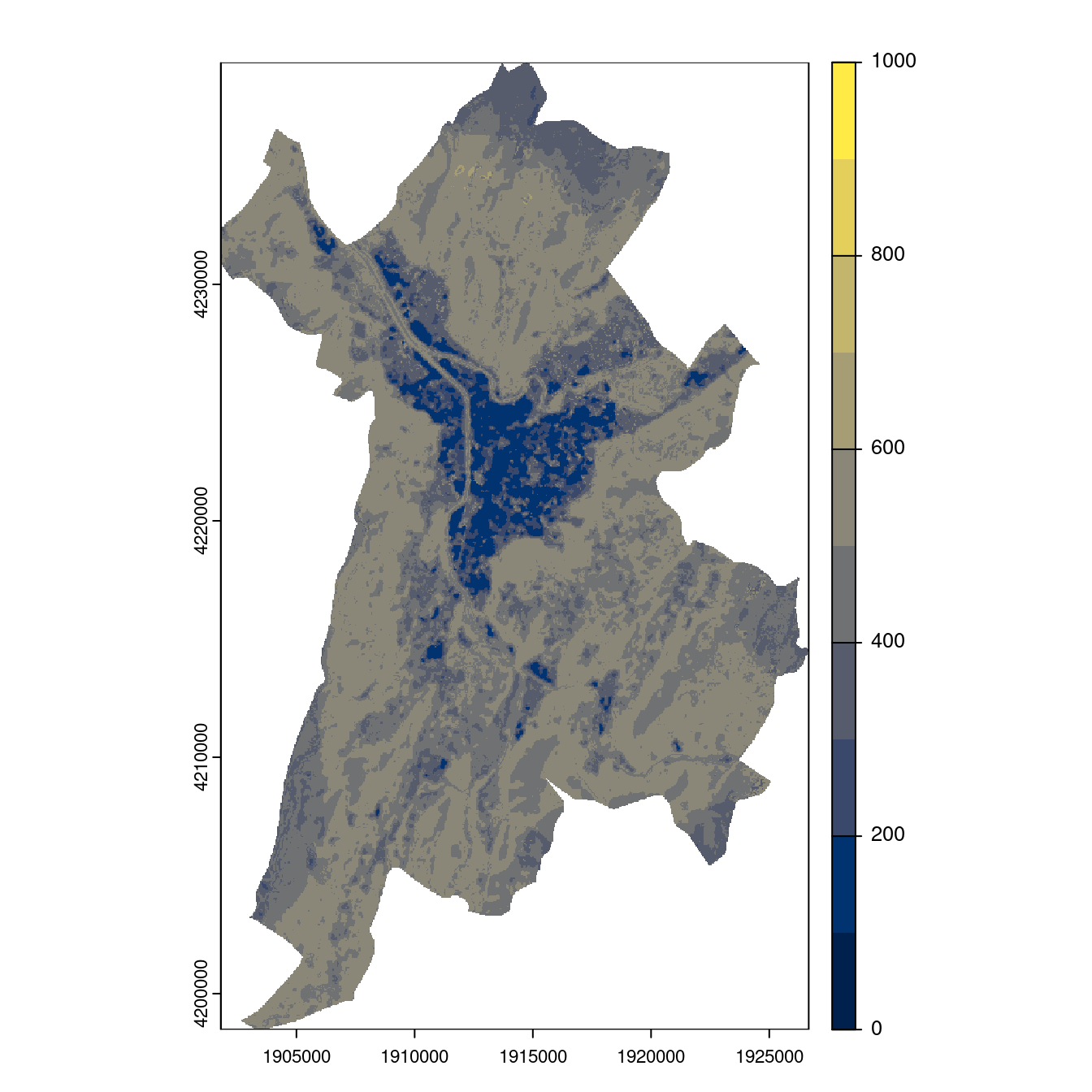
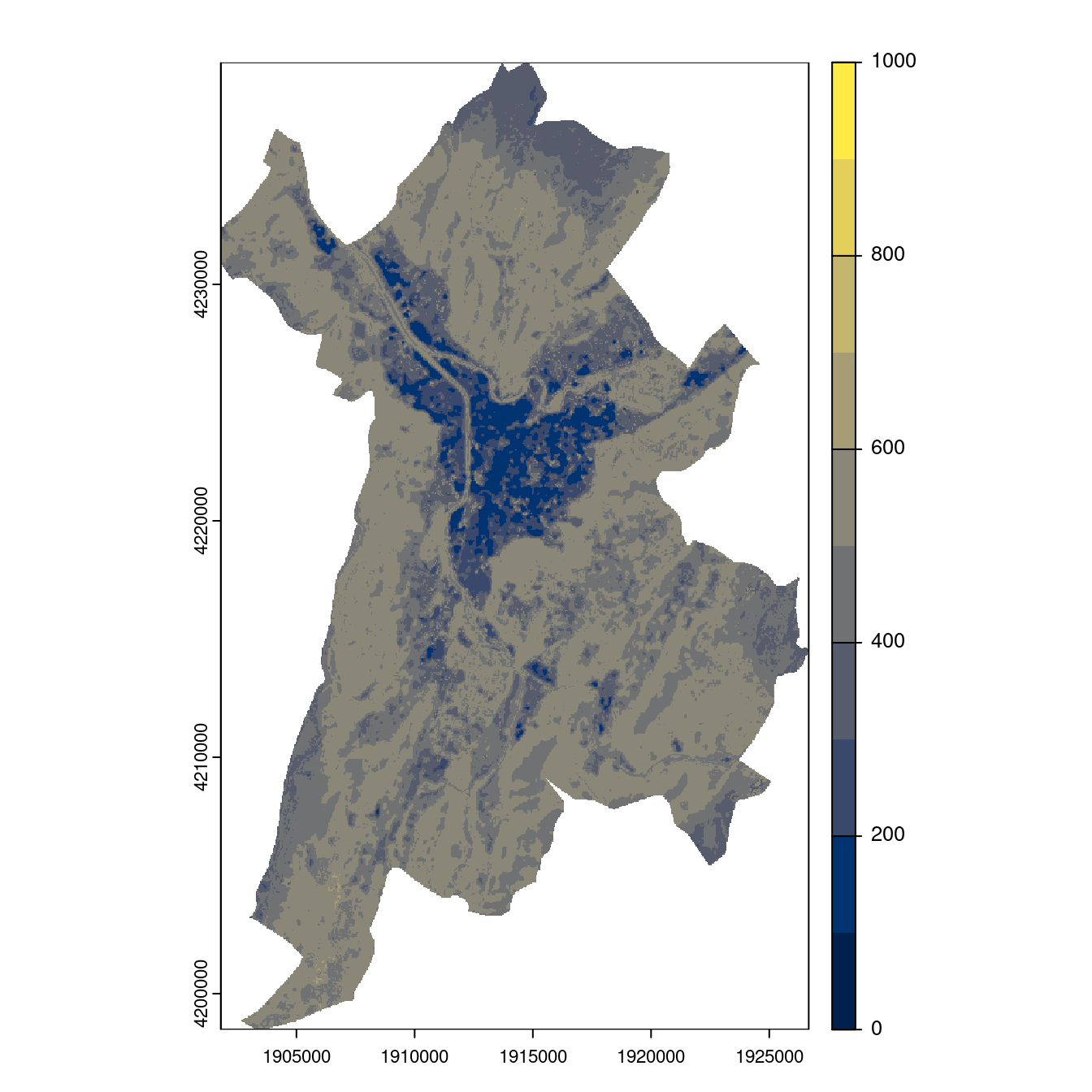
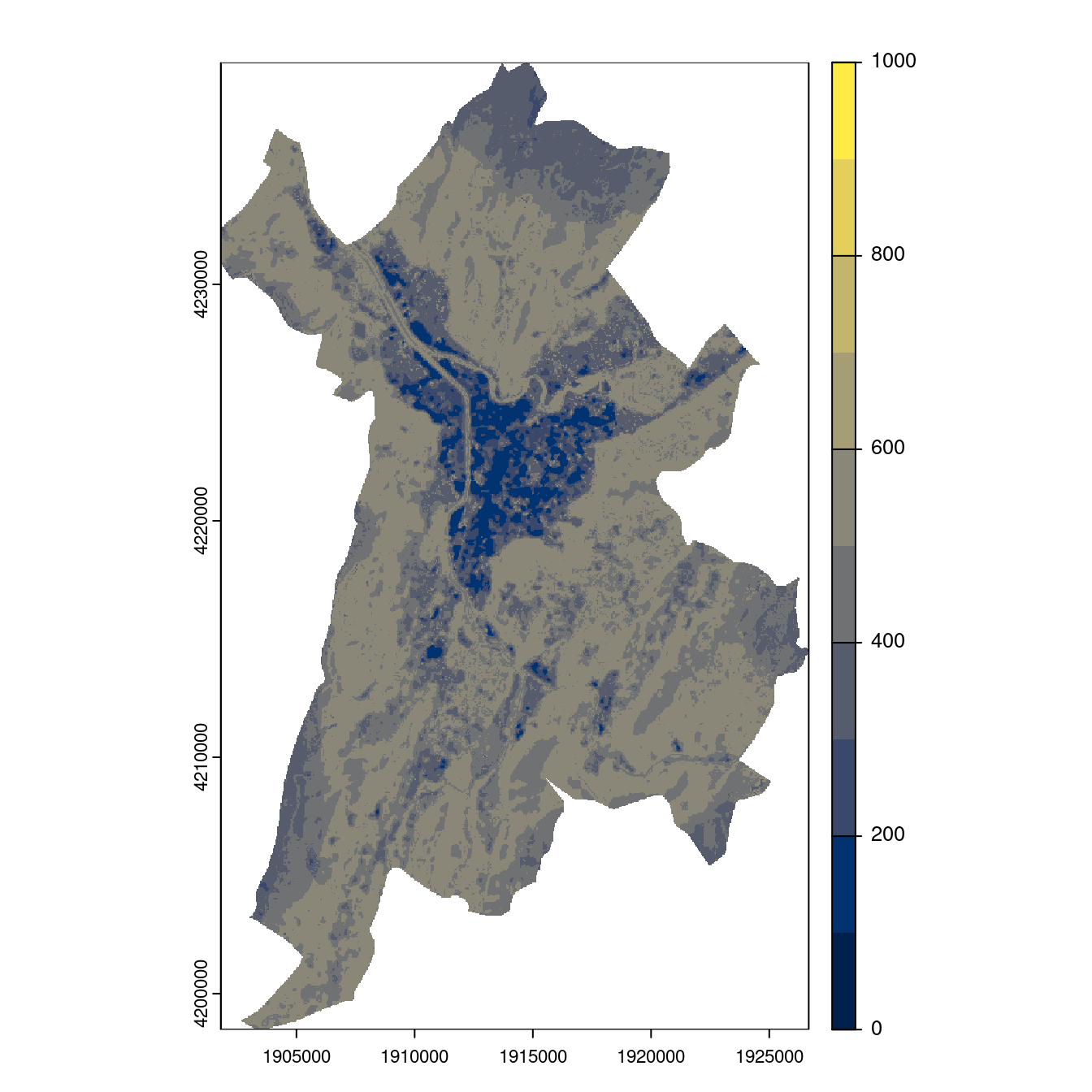
Figure 240.24: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP2-4.5
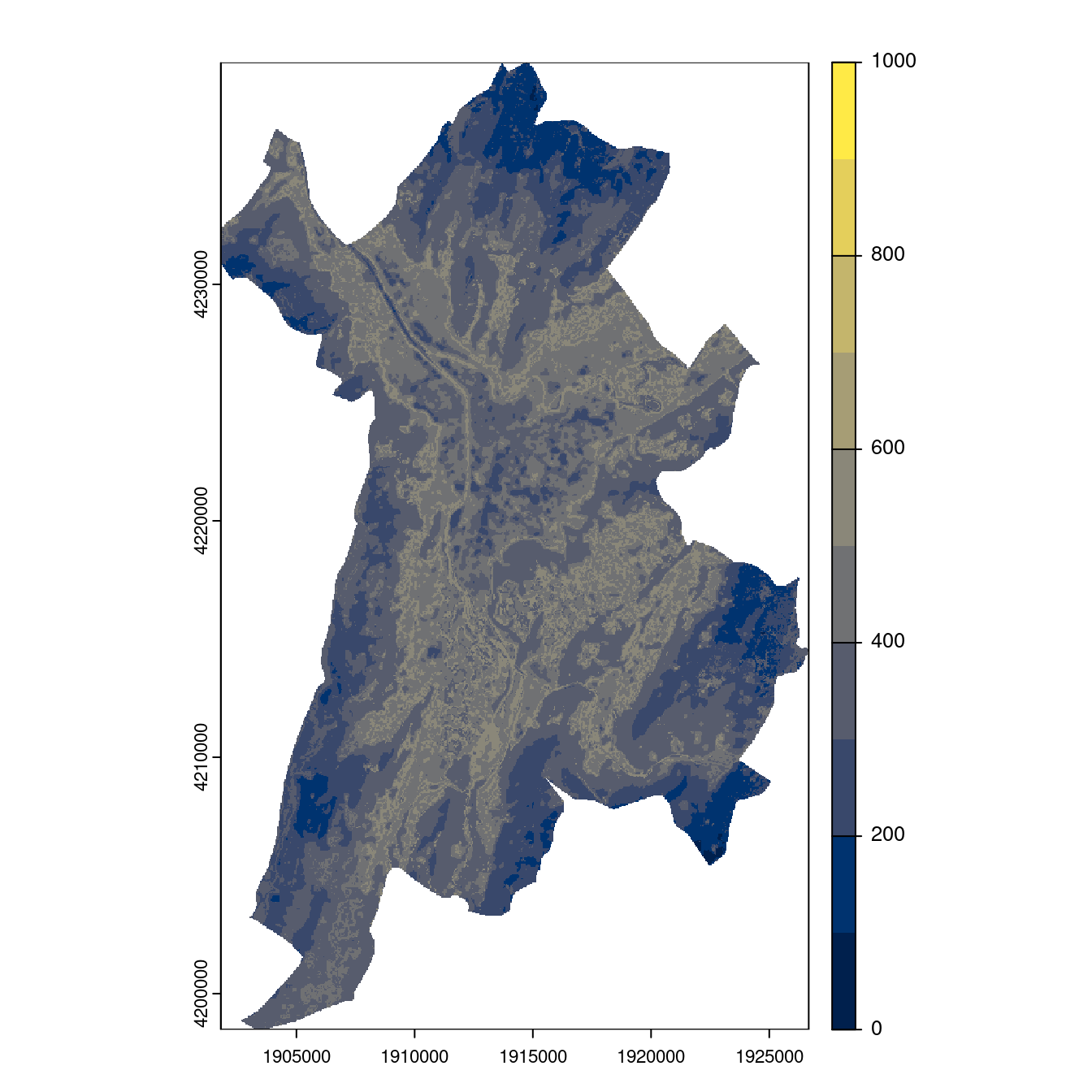
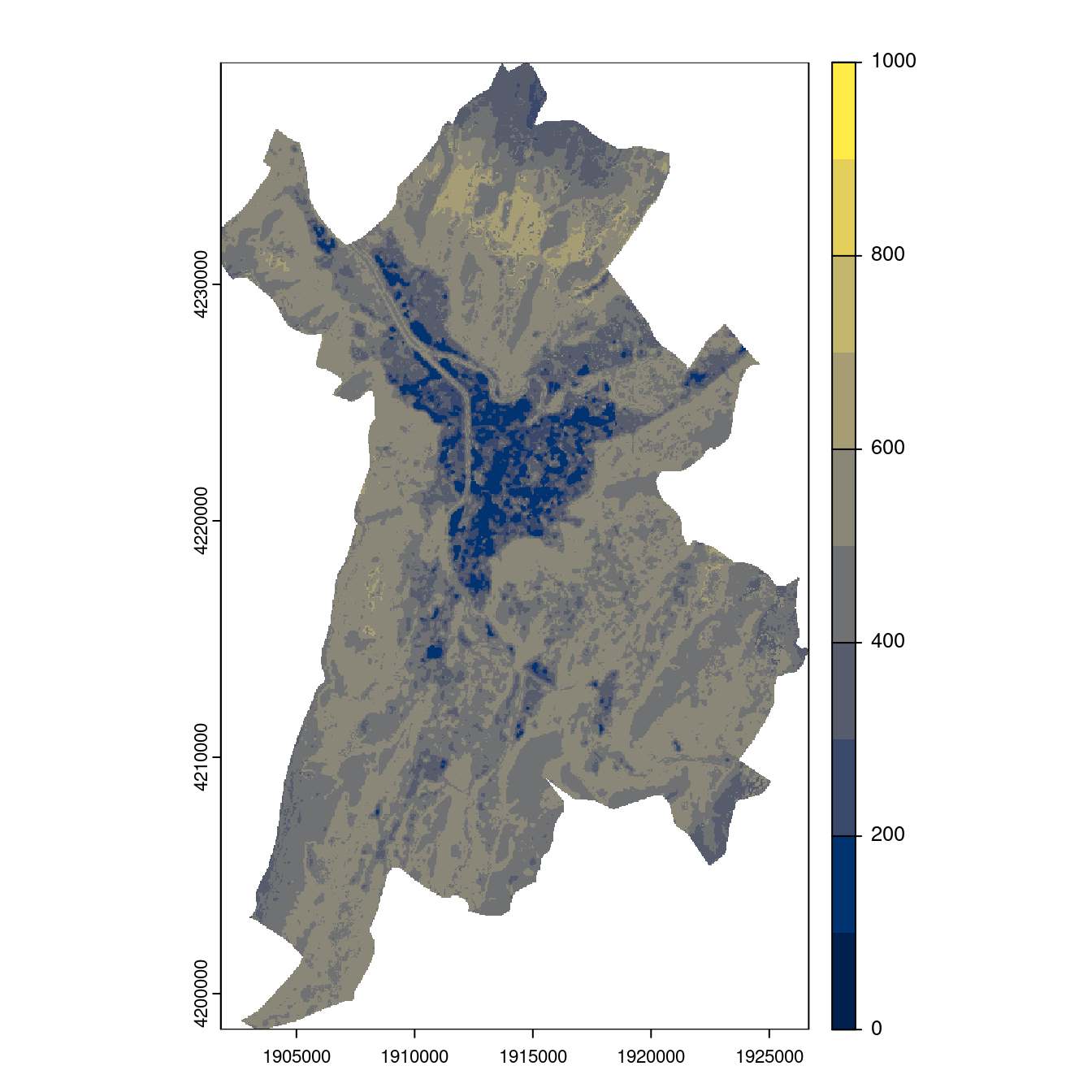
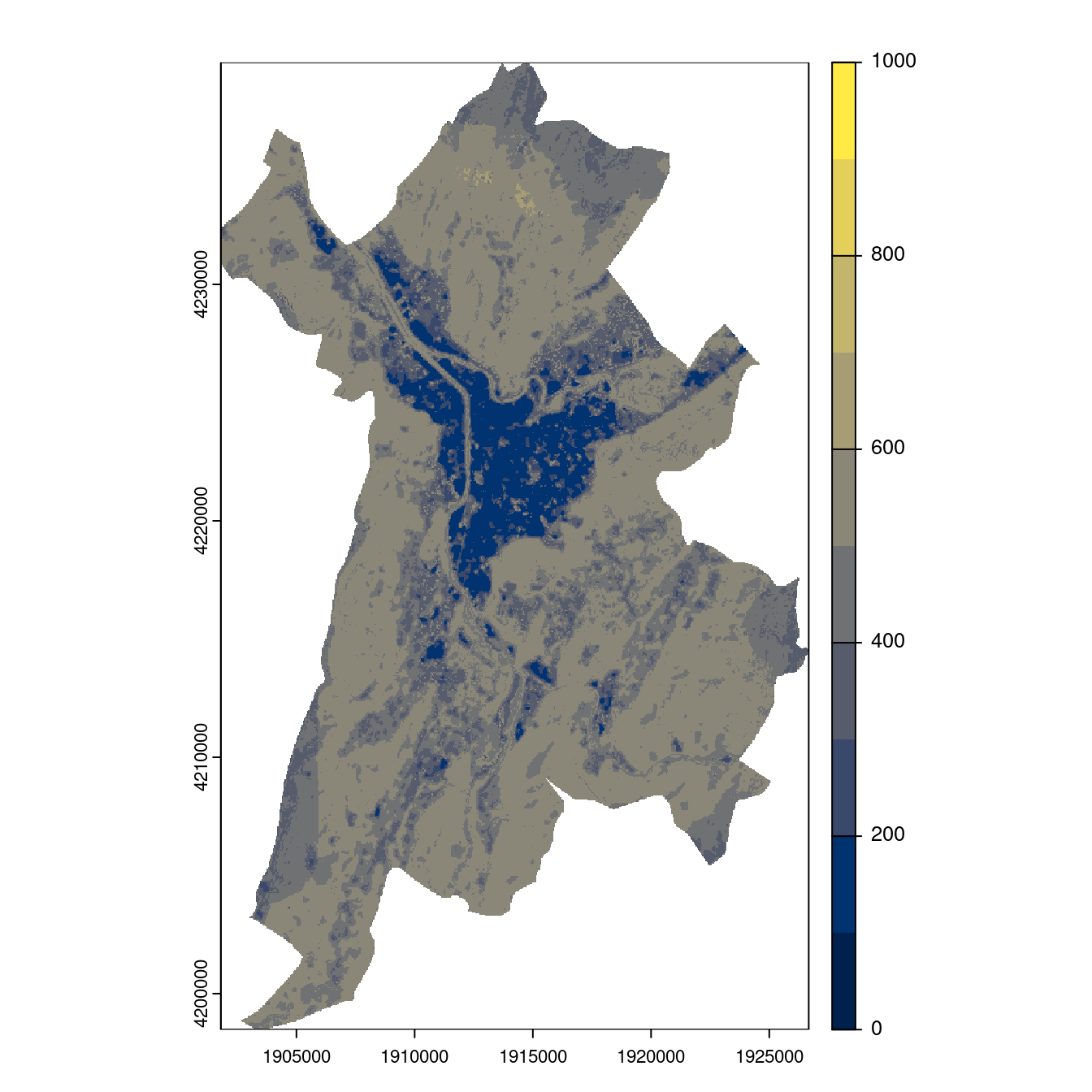
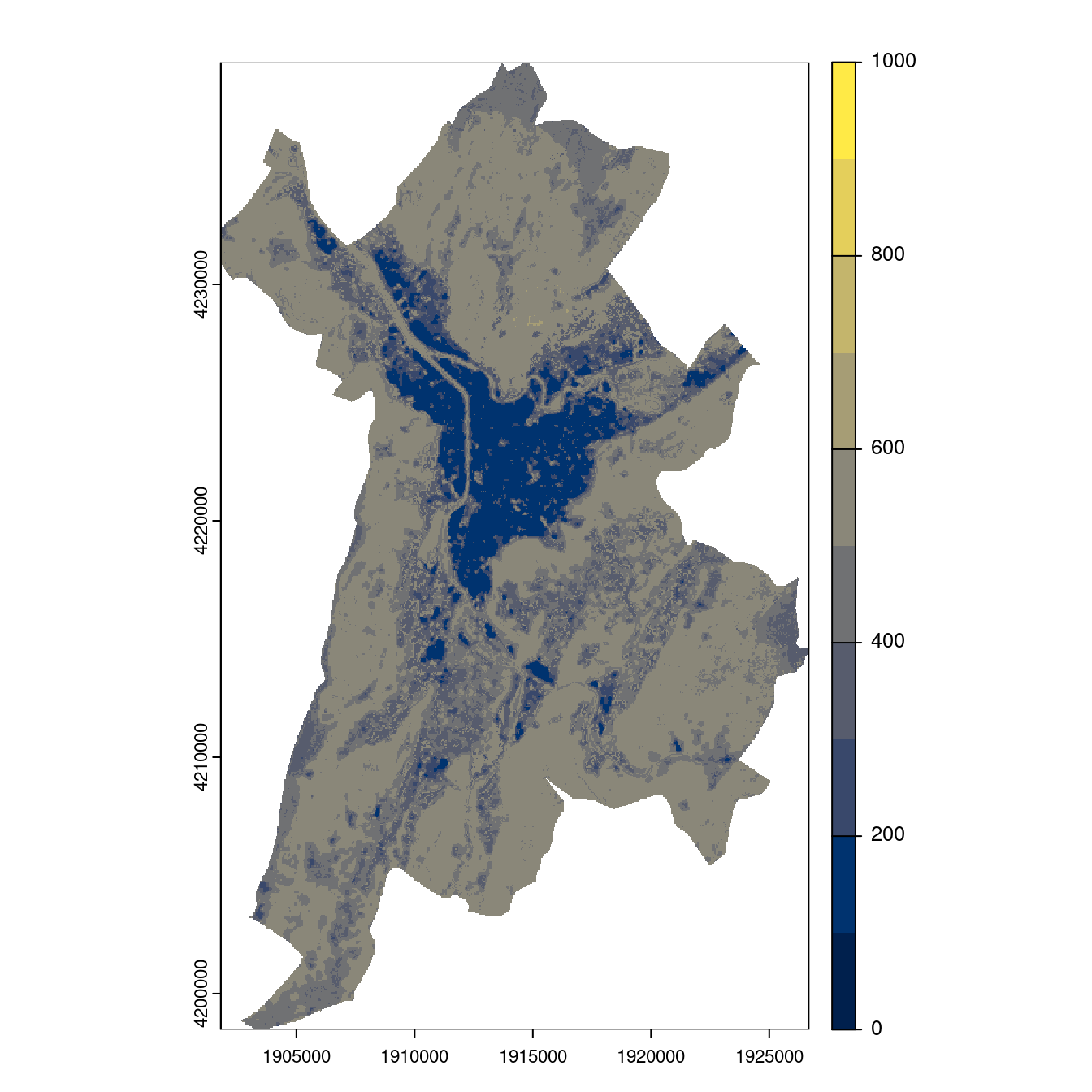
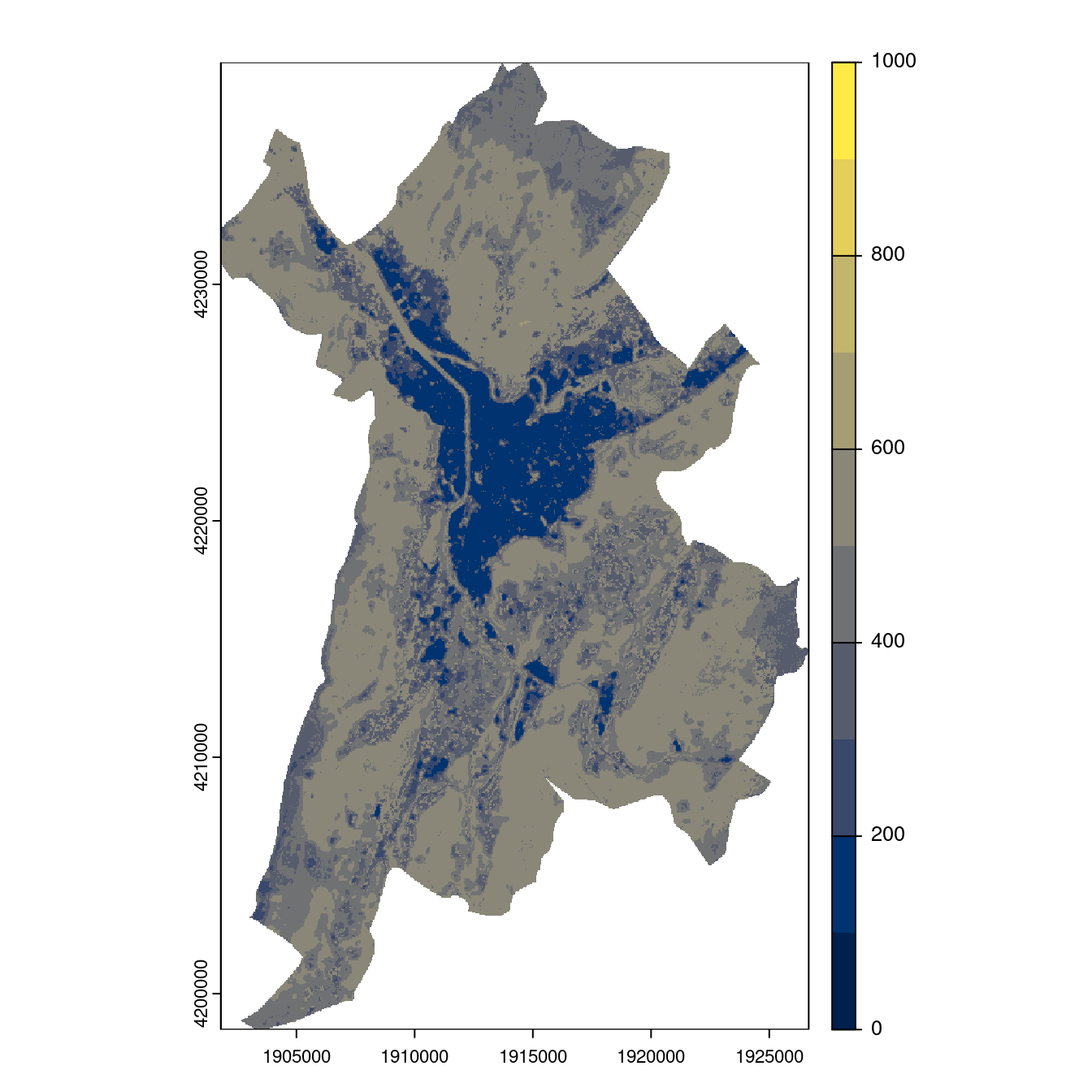
Figure 240.25: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP3-7.0
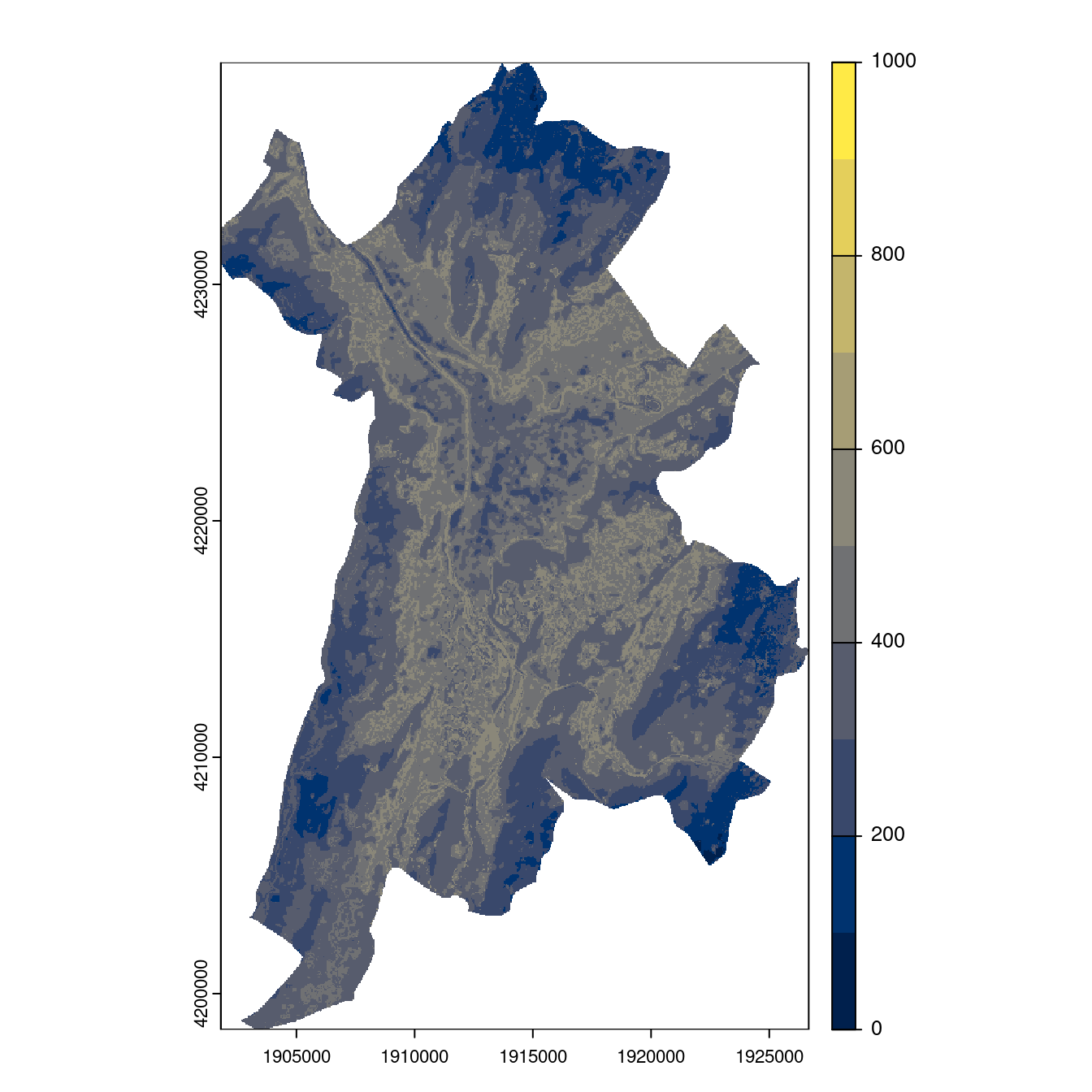
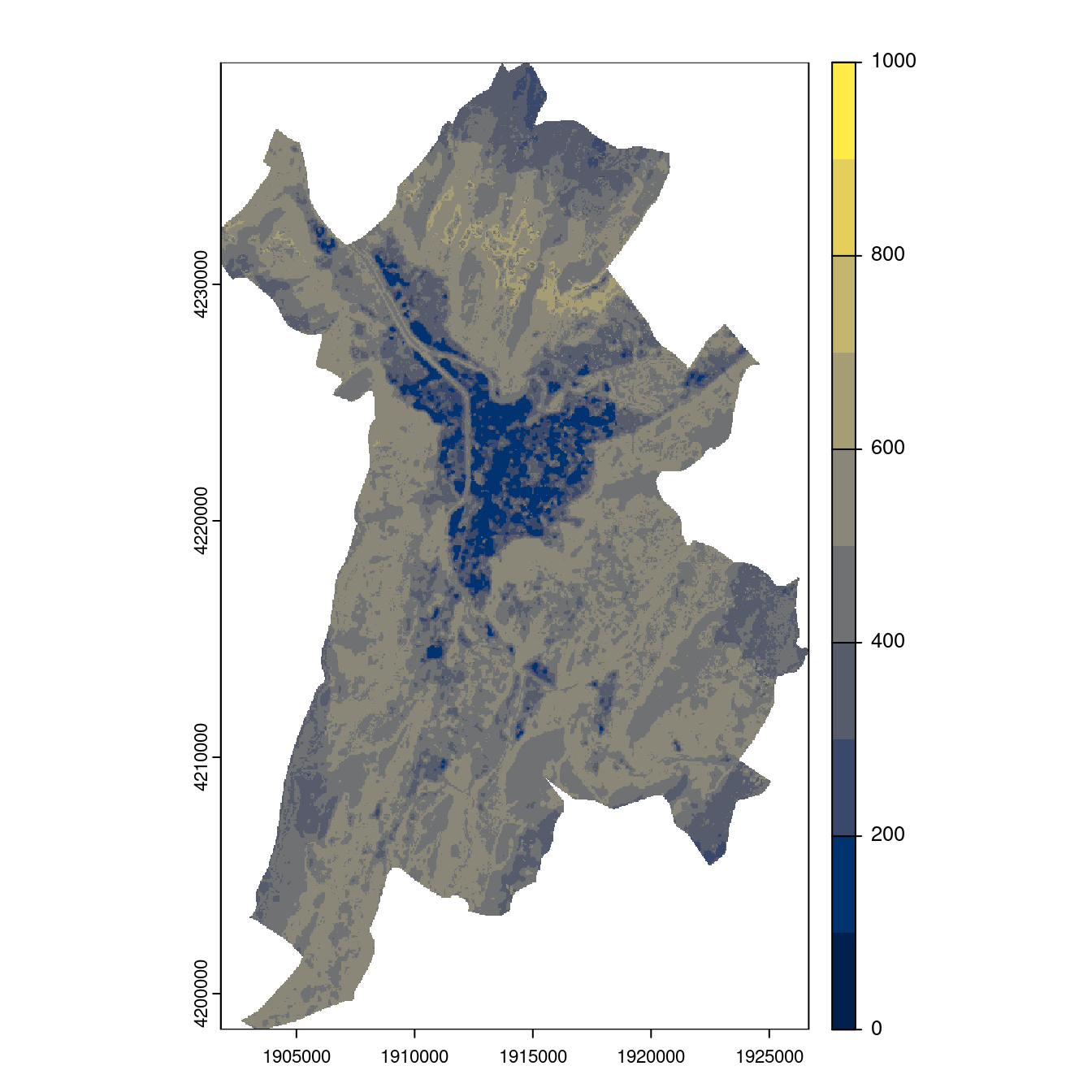
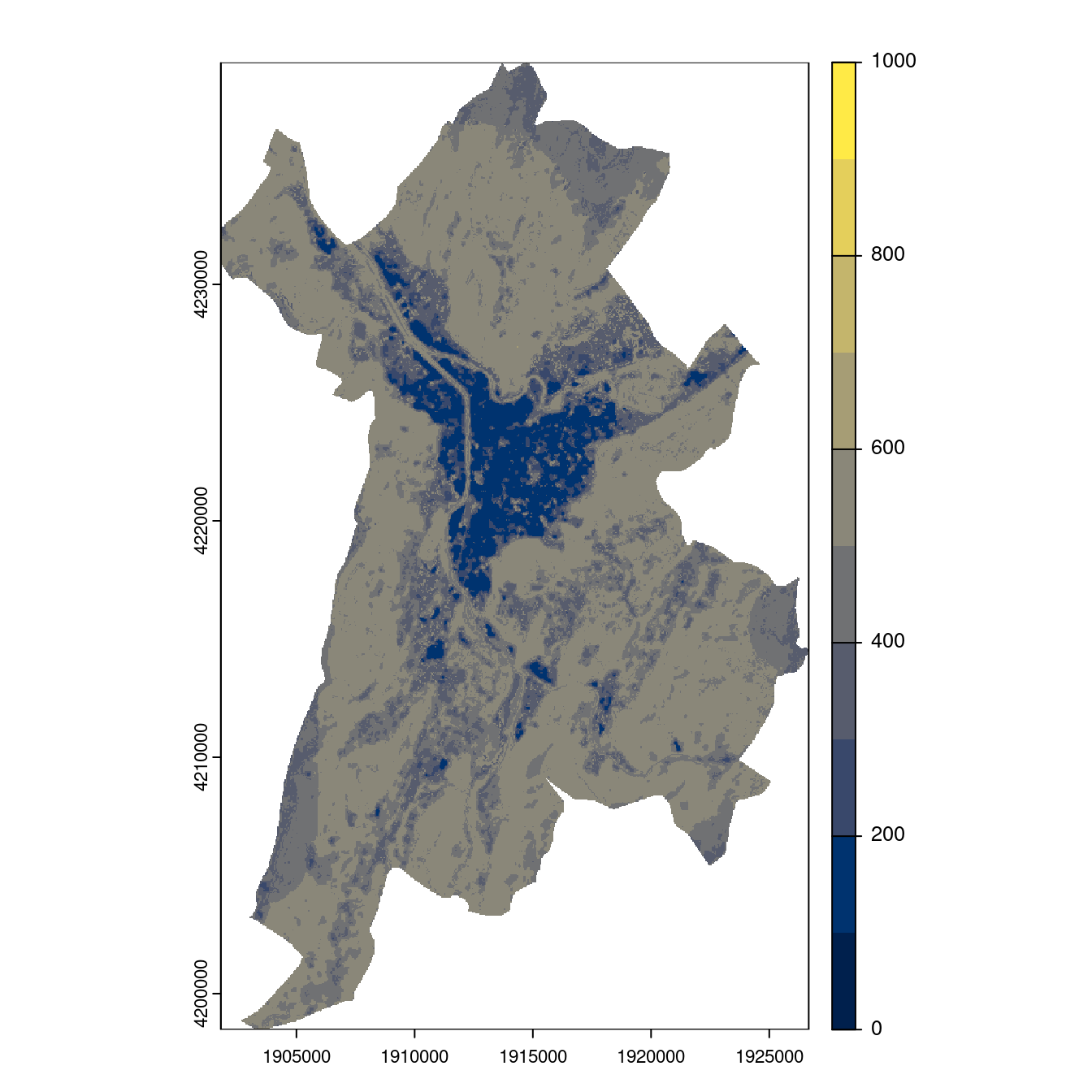
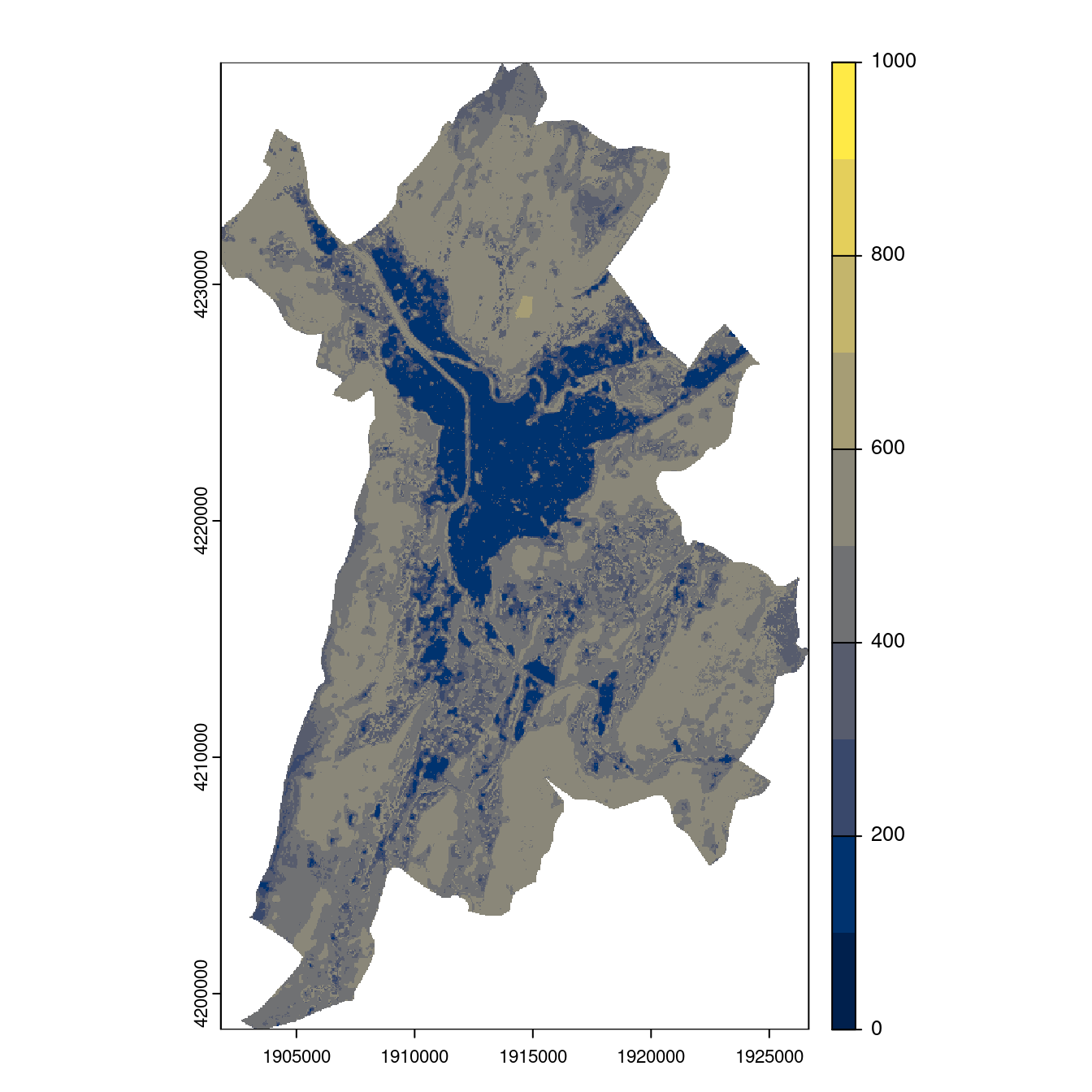
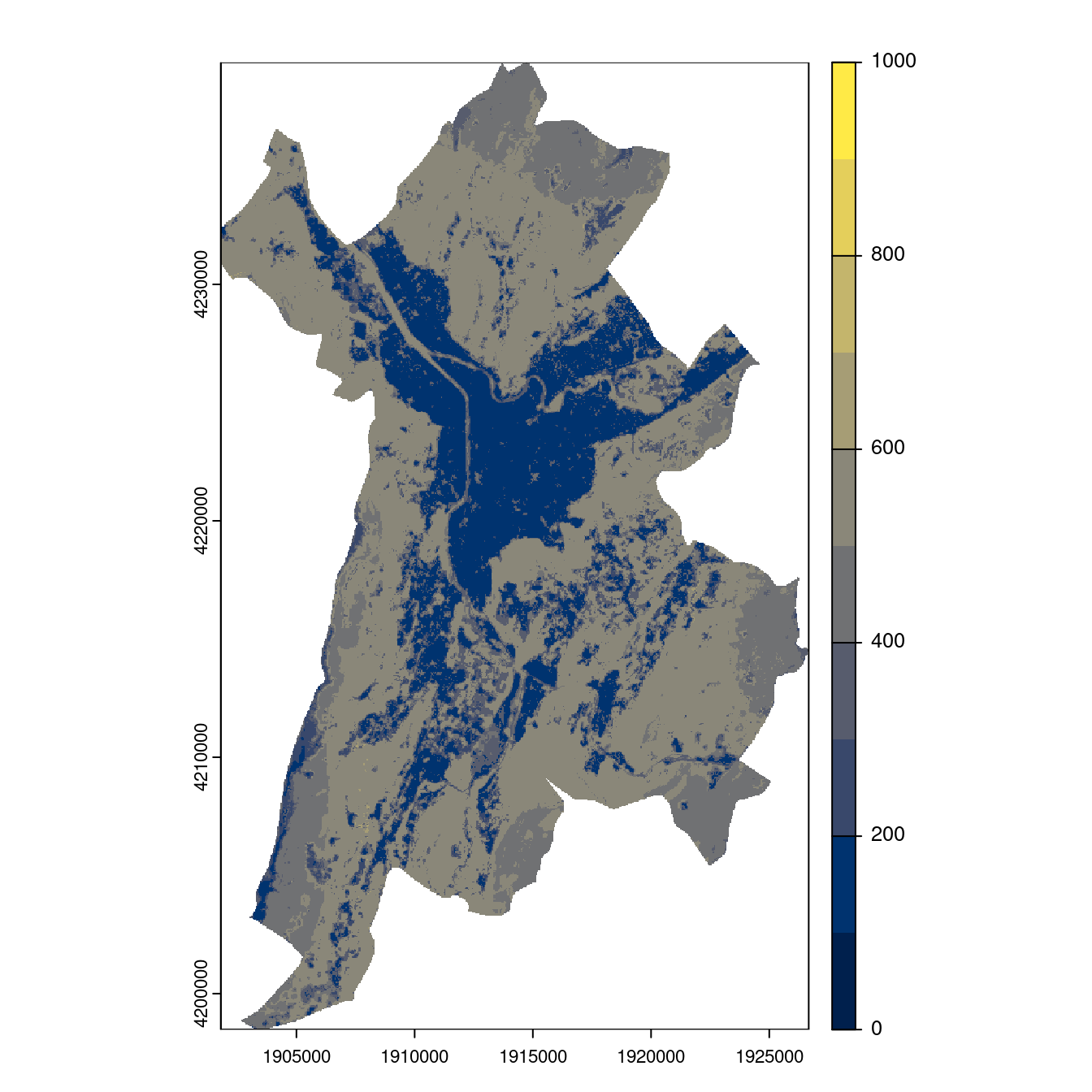
Figure 240.26: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP5-8.5
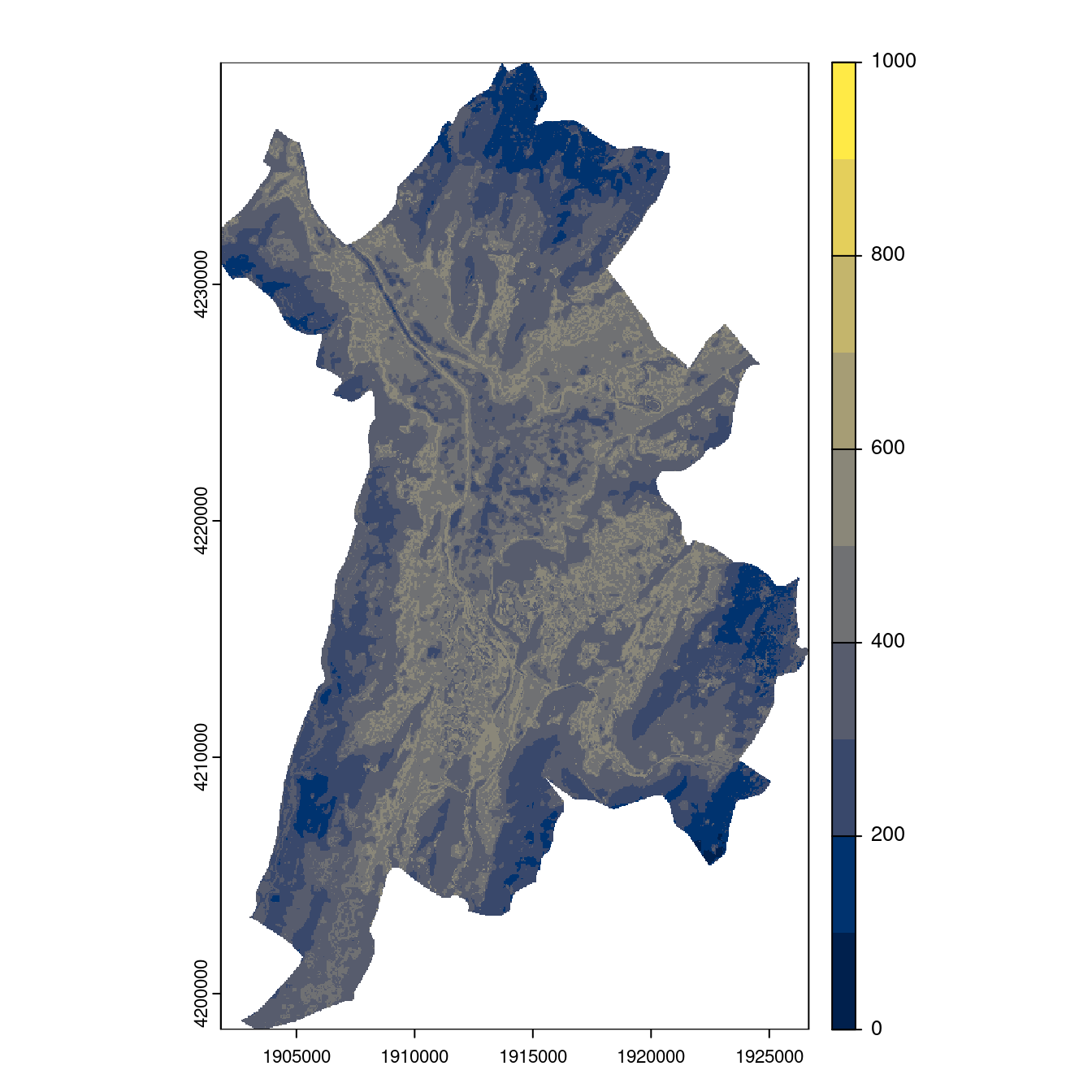
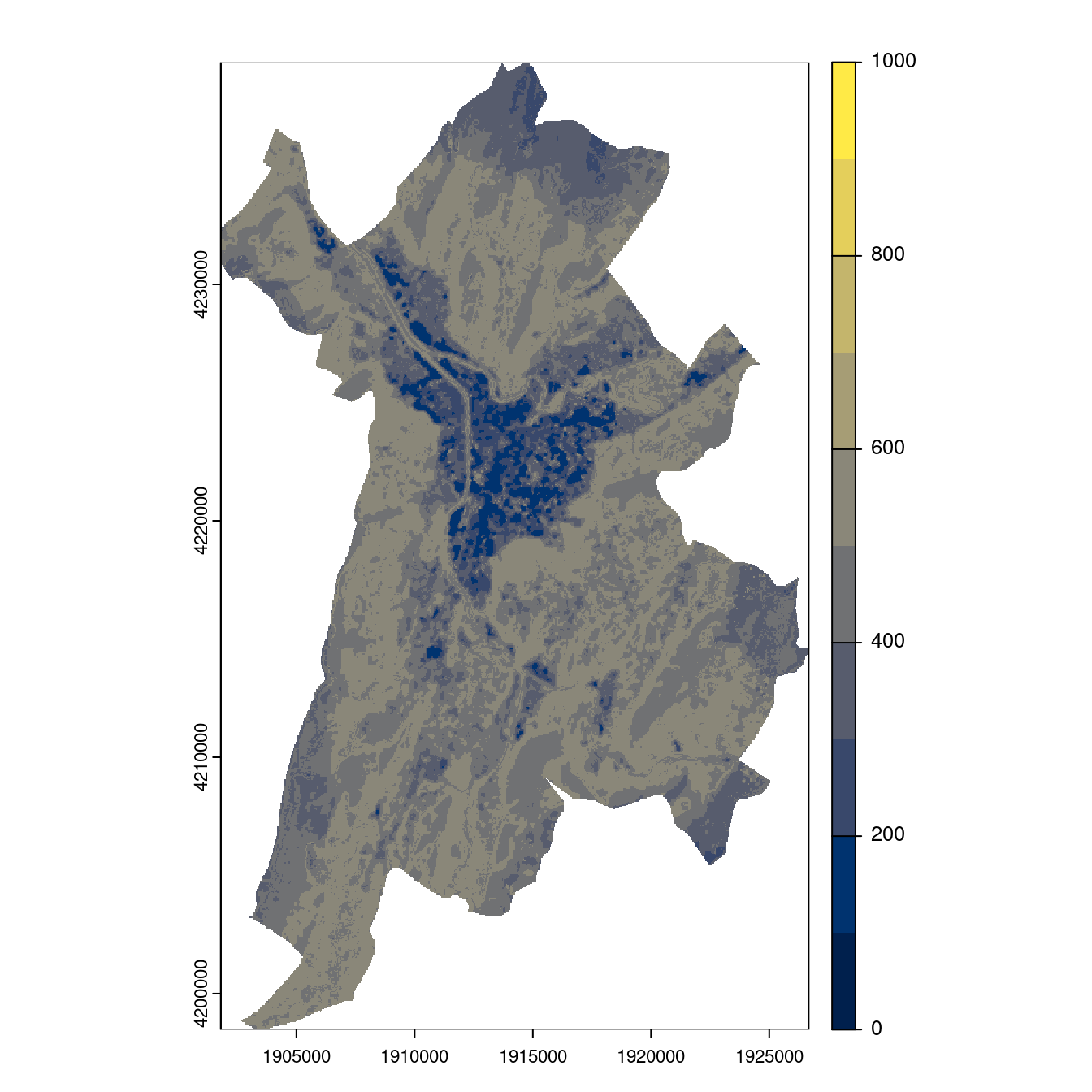
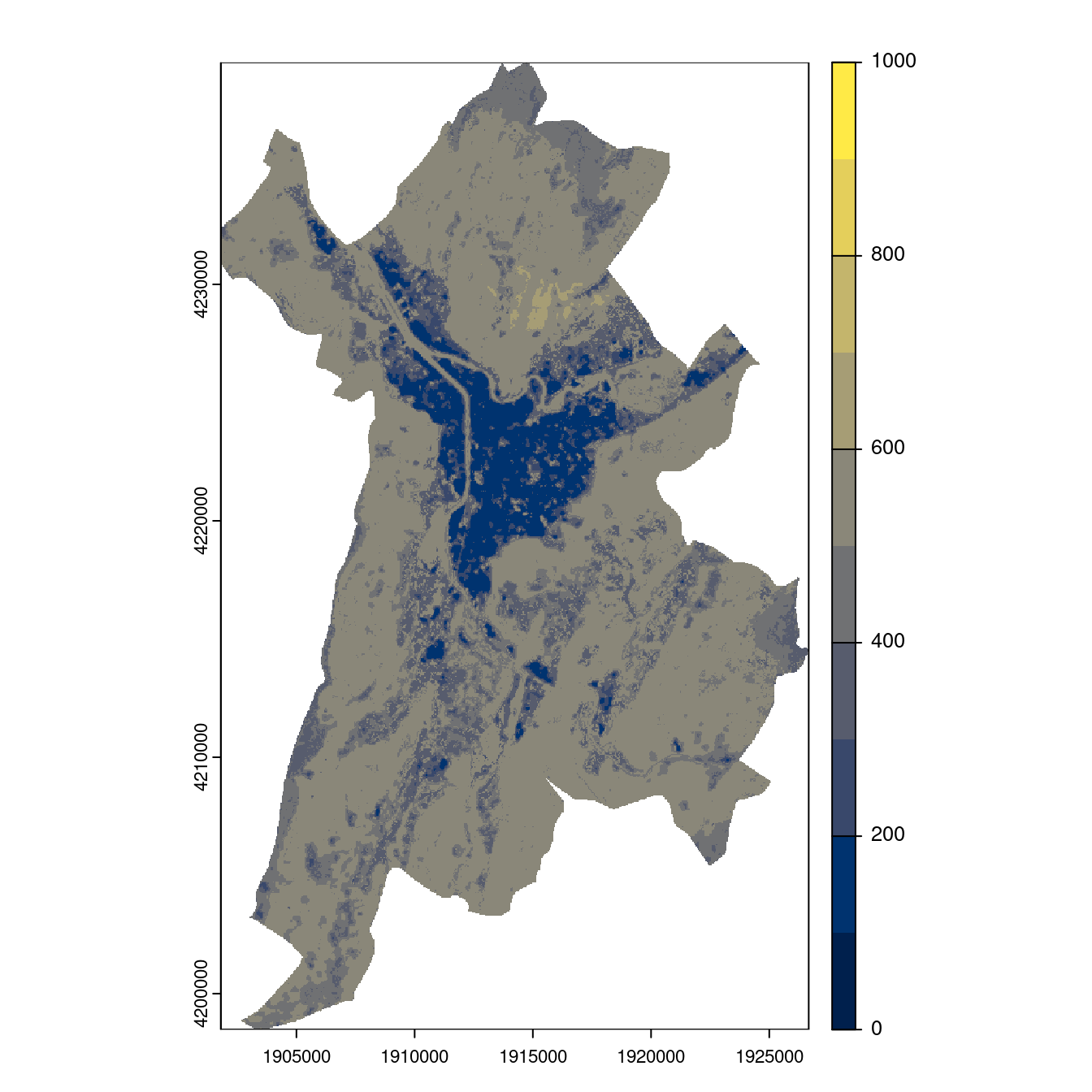
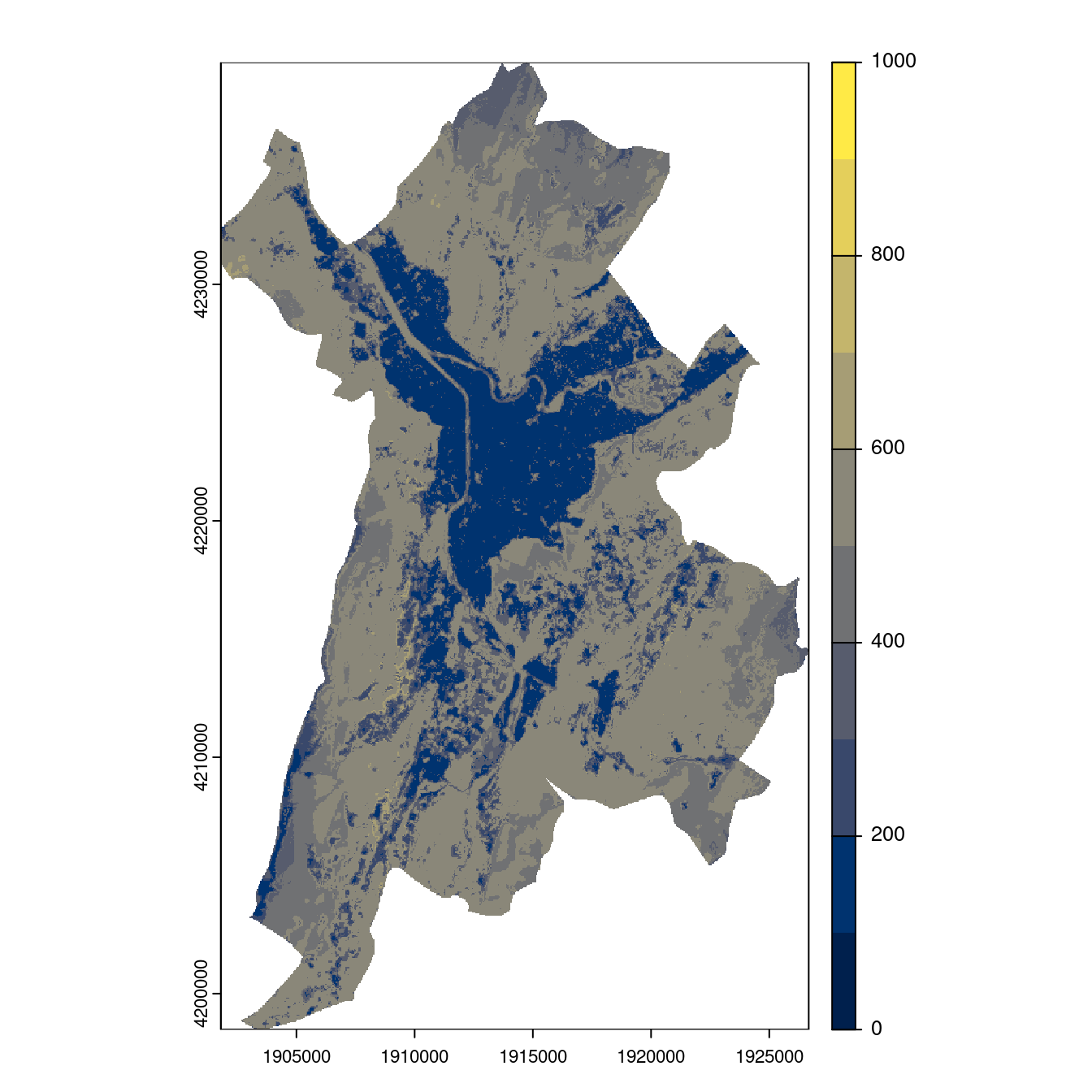
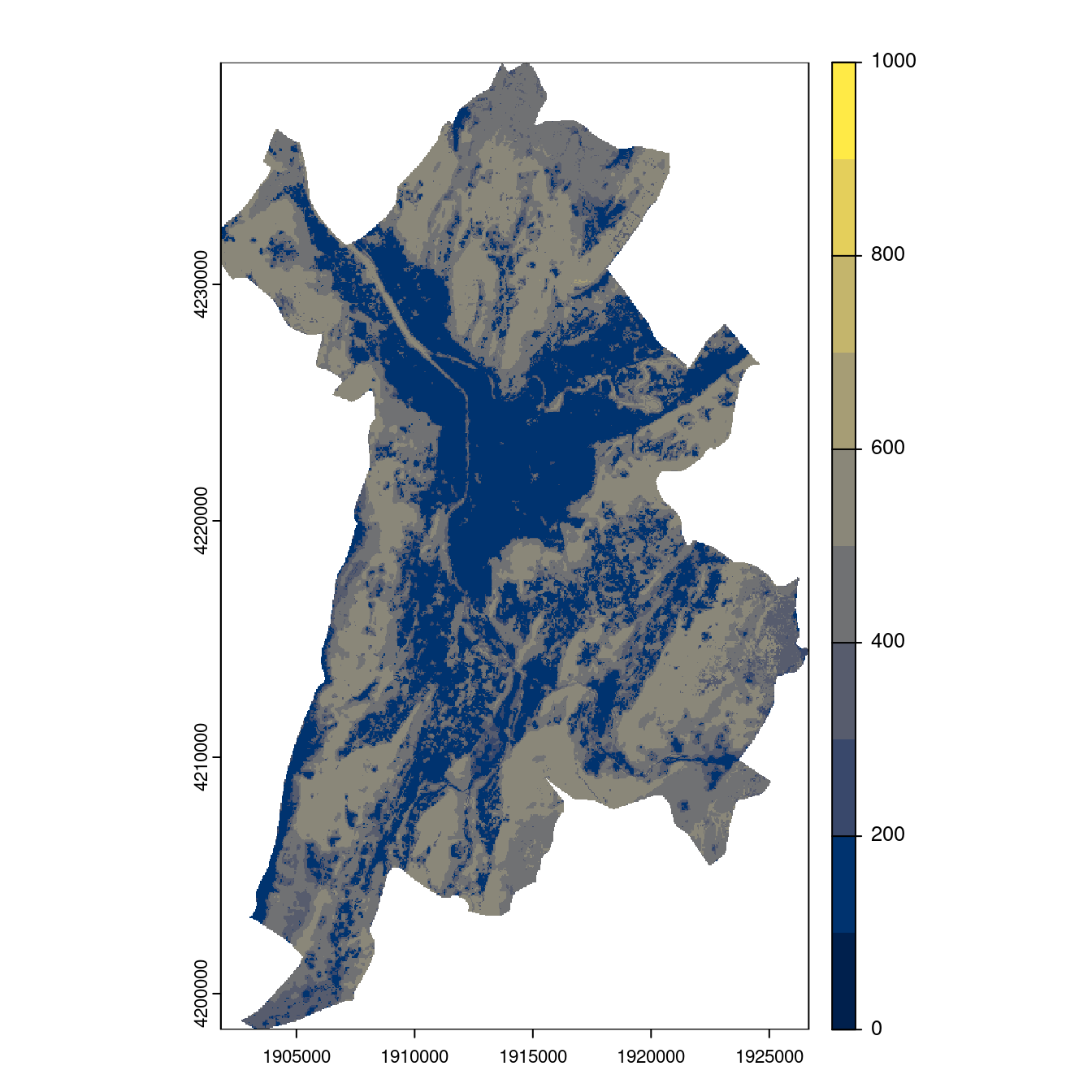
Figure 240.27: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
240.4.2 Évolution temporelle
# A tibble: 17 × 8
min q1 median mean q3 max ssp year
<table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <fct> <dbl>
1 92 302 371 366.2165409 435 593 0 2000
2 156 408 463 457.0430755 543 627 126 2040
3 153 415 489 463.3838256 548 607 126 2060
4 166 421 499 470.4470309 556 603 126 2080
5 171 411 495 468.2104793 554 595 126 2100
6 160 422 485 470.4345484 553 658 245 2040
7 154 426 512 472.3048884 561 608 245 2060
8 153 411 512 469.4913131 563 603 245 2080
9 158 378 495 453.6381682 548 601 245 2100
10 141 411 470 460.2128248 548 634 370 2040
11 154 428 516 474.1901110 559 600 370 2060
12 154 364 487 440.6380027 534 608 370 2080
13 157 321 498 422.7054787 530 618 370 2100
14 169 411 471 458.8459245 547 598 585 2040
15 163 428 530 482.3280557 569 610 585 2060
16 147 346 503 431.5411802 535 617 585 2080
17 114 172 429 373.8073663 506 605 585 2100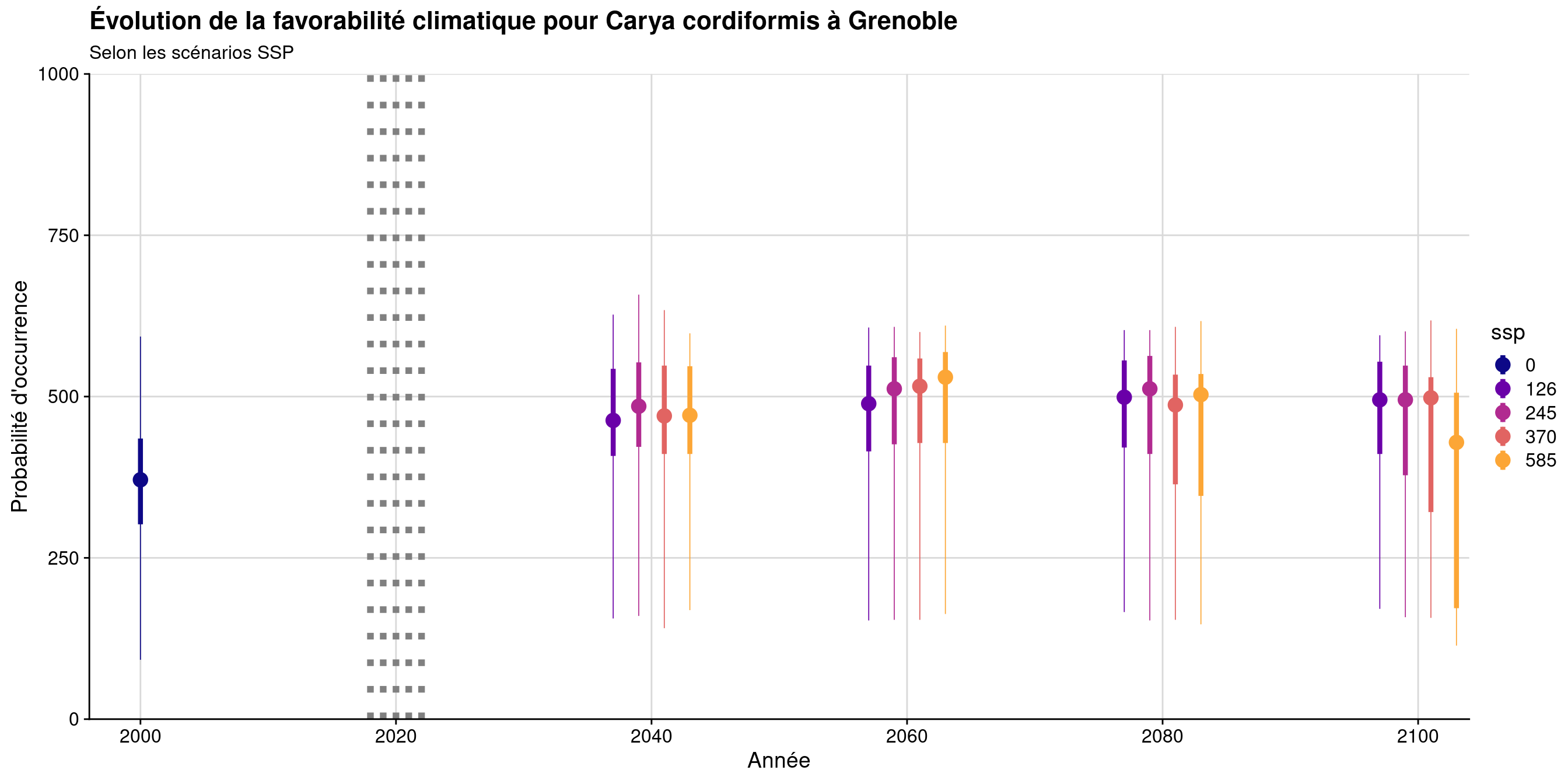
Figure 240.28: Évolution de la favorabilité climatique au cours du XXIème siècle. Le point représente la médiane sur l’étendue de la métropole (i.e. tous les pixels de la carte) ; le trait gras représente 50 % des données intermédiaires (espace inter-quartile) ; le trait fin représente le reste des données du minimum au maximum.