128 Cornus controversa
128.1 Données d’occurrences GBIF
Collecte des données du GBIF :
<<gbif download metadata>>
Status: SUCCEEDED
DOI: 10.15468/dl.g8psra
Format: SIMPLE_CSV
Download key: 0252518-230224095556074
Created: 2023-05-20T16:08:49.784+00:00
Modified: 2023-05-20T16:09:38.728+00:00
Download link: https://api.gbif.org/v1/occurrence/download/request/0252518-230224095556074.zip
Total records: 2345Conversion en objets géographiques (format Simple feature de sf) et
exploration rapide des données (classe du jeu de données et premières lignes du
tableau) :
Simple feature collection with 2345 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -123.233333 ymin: 15.066667 xmax: 145.667401 ymax: 54.1527
Geodetic CRS: WGS 84
# A tibble: 2,345 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 9 e8 1b412457-… 5423492 Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
2 9 e8 1b412457-… 5423491 Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
3 7.e8 aab0cf80-… LD:General:… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
4 7 e8 86185376-… 7b45ff66-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
5 7 e8 86185376-… 7b436c56-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
6 7 e8 86185376-… 7b2432aa-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
7 7 e8 86185376-… 7ac6fba8-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
8 7 e8 86185376-… 7aba71bc-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
9 7 e8 7adffacc-… beab7930-f1… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
10 7 e8 81031dbc-… urn:catalog… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
# ℹ 2,335 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <lgl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <lgl>, depthAccuracy <lgl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …Il y a 2 345 occurrences dans le jeu de données. On les affiche sur la carte du monde :
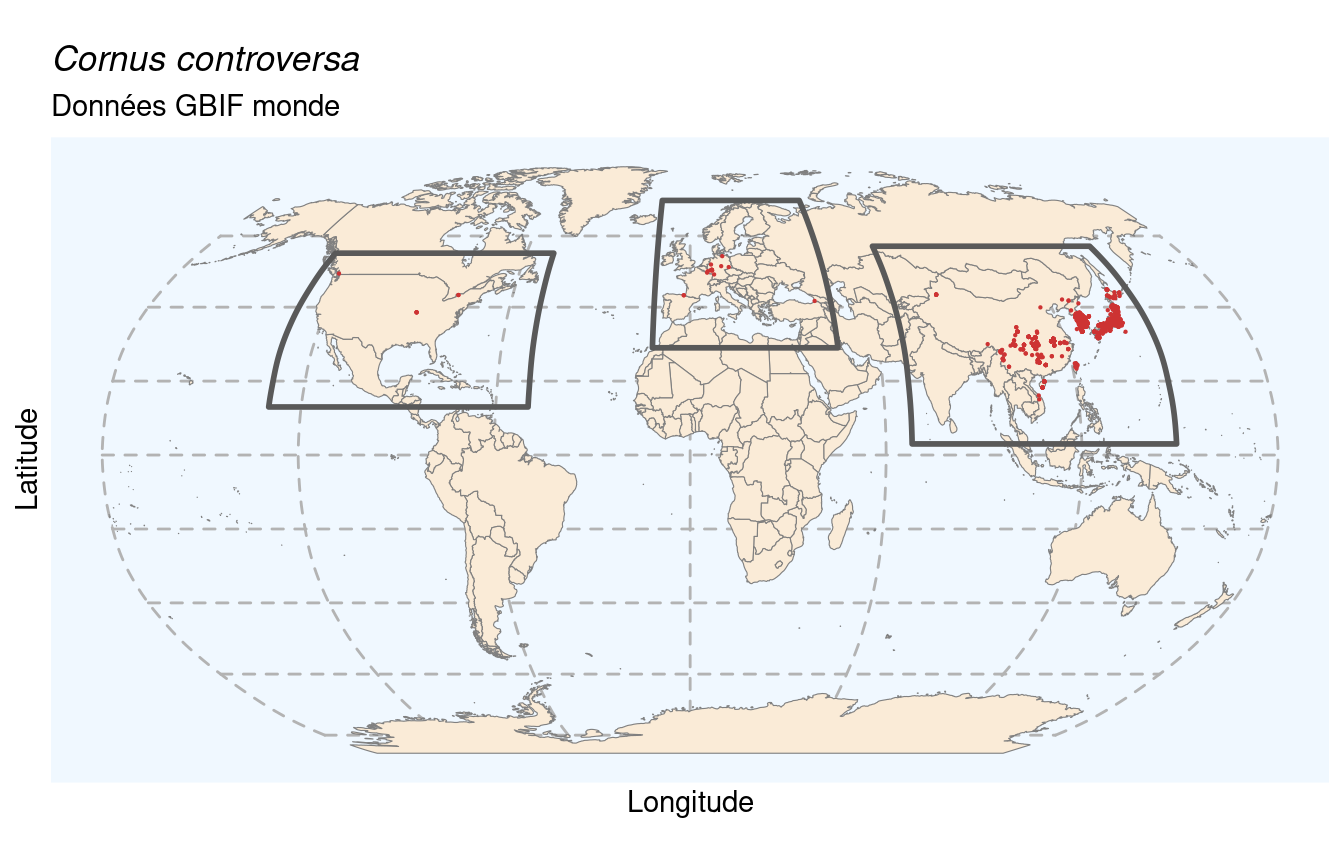
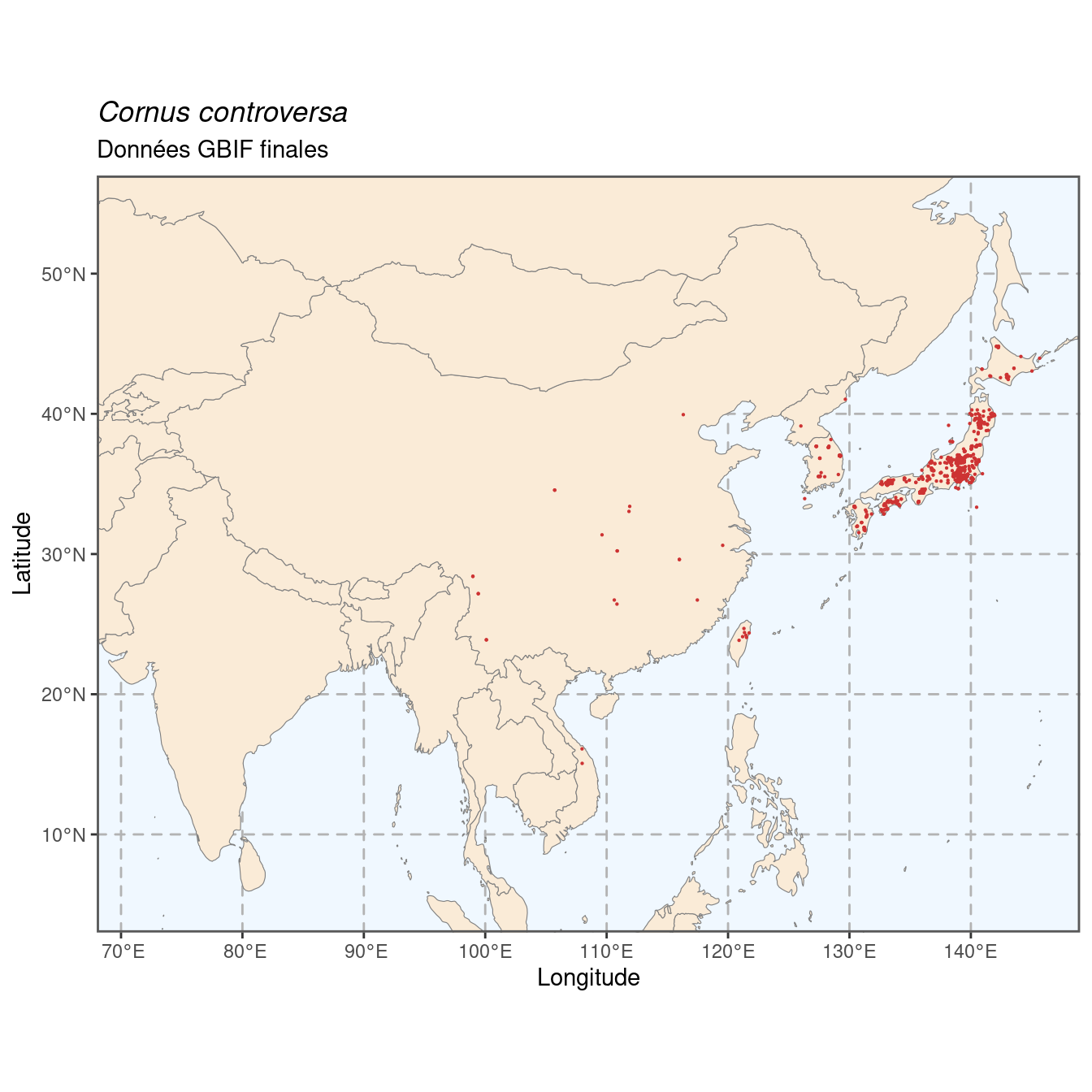
Figure 128.1: Occurrences de Cornus controversa dans le monde.
128.1.1 Données par région
On évalue la proportion d’occurrences dans chaque région, en utilisant les 3 masques créés précédemment. Si on a au moins 50 % des occurrences dans une région, on peut continuer en travaillant uniquement sur ces données. Sinon, on vérifie qu’au moins 67 % des données tombent dans l’ensemble des régions, et on prend la région la plus représentée. Au final, s’il s’agit d’une espèce endémique d’Europe, on n’a pas besoin de manipulation supplémentaire.
[1] 0.007249466951[1] 0.002985074627[1] 0.9897654584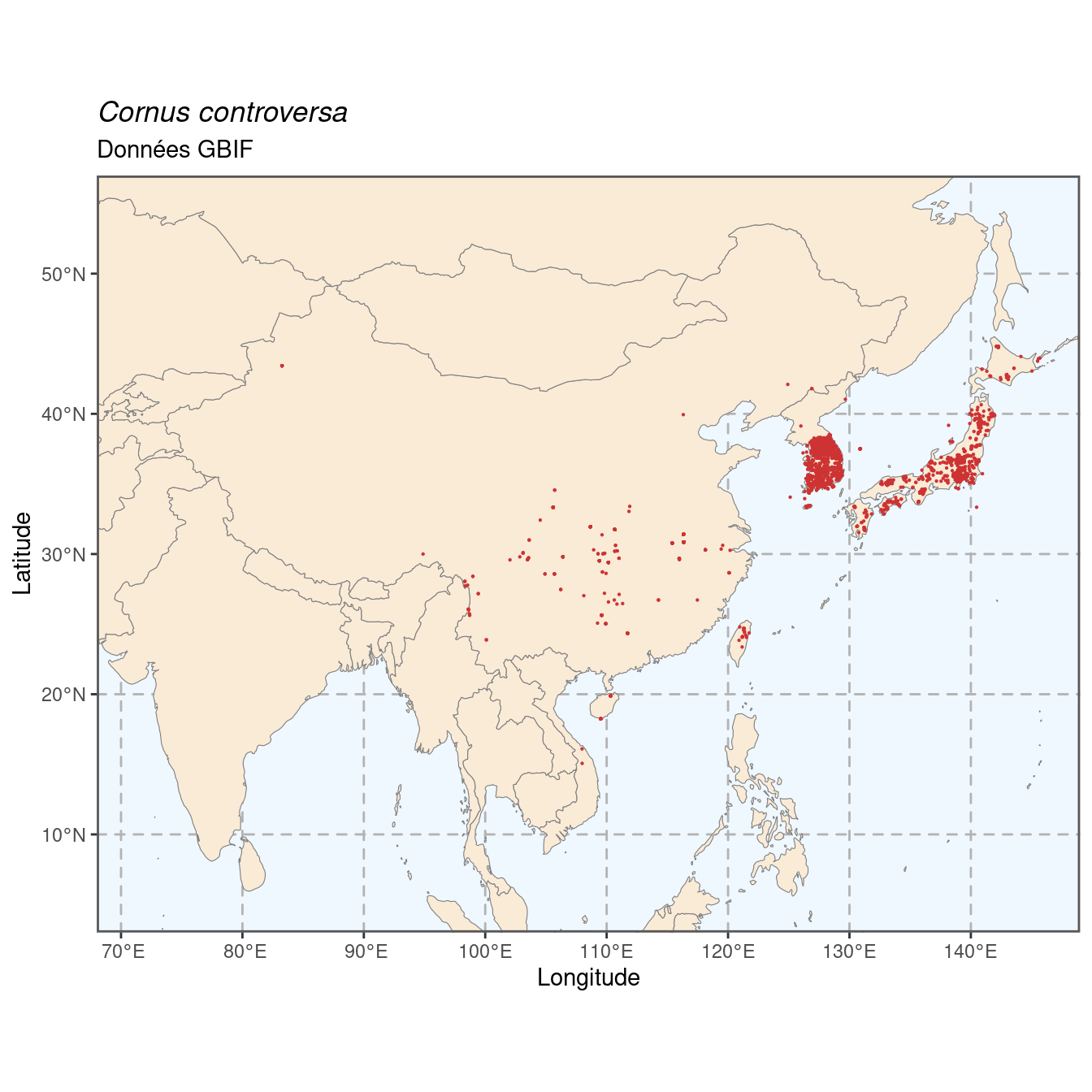
Figure 128.2: Occurrence de Cornus controversa dans la région d’endémisme.
128.1.2 Licences
On s’intéresse ici au problème particulier des licences :
CC_BY_4_0 CC_BY_NC_4_0 CC0_1_0
820 1429 72 [1] 38.43171047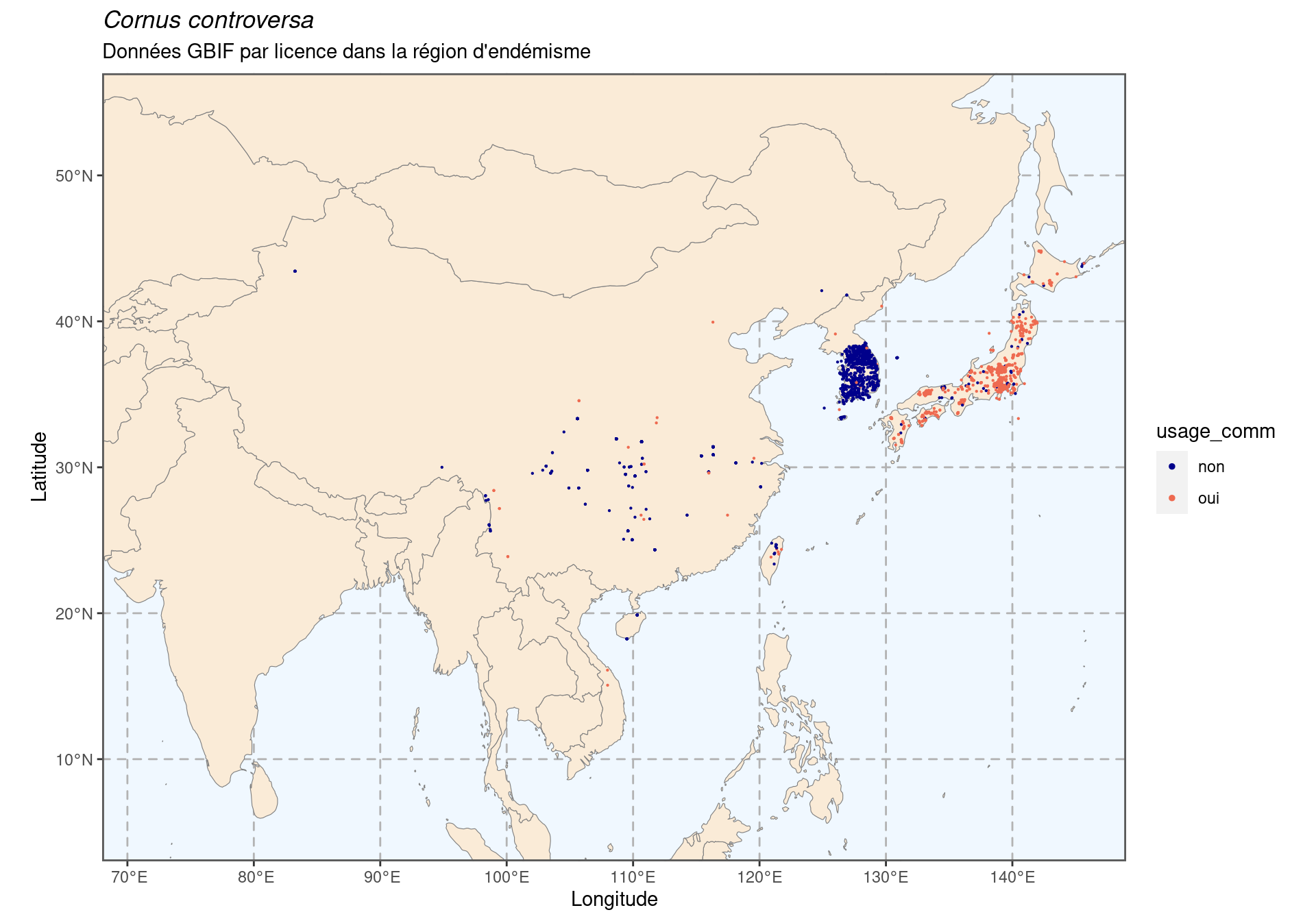
Figure 128.3: Occurrence de Cornus controversa dans la région d’endémisme, selon la licence : CC BY-NC en bleu ; CC0 et CC BY en orange.
Idéalement, on aimerait avoir au moins 50 % de données vraiment libres
d’utilisation (c’est-à-dire sans la clause -NC). On continue en supprimant les
données en CC BY-NC.
[1] 892128.1.3 Sous-échantillonnage
Si le jeu de données contient 25 000 occurrences ou plus, on procède au sous-échantillonnage aléatoire pour en conserver un maximum de 25 000.
Simple feature collection with 892 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: 98.9833 ymin: 15.066667 xmax: 145.665004 ymax: 44.8194
Geodetic CRS: WGS 84
# A tibble: 892 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
<int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 9e8 1b412457-… 5423492 Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
2 9e8 1b412457-… 5423491 Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
3 7e8 86185376-… 7b45ff66-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
4 7e8 86185376-… 7b436c56-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
5 7e8 86185376-… 7b2432aa-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
6 7e8 86185376-… 7ac6fba8-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
7 7e8 86185376-… 7aba71bc-f8… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
8 7e8 7adffacc-… beab7930-f1… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
9 6e8 80fc4302-… eb95ea24-f1… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
10 6e8 80fc4302-… ea49be20-f1… Plantae Trach… Magn… Corn… Corna… Corn… Cornus… ""
# ℹ 882 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <lgl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <lgl>, depthAccuracy <lgl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …128.2 Modélisation de la niche climatique
128.2.1 Préparer les données
Organisation des données d’entrées (occurrences et données environnementales) et des données produites de pseudo-absences :
class : SpatVector
geometry : points
dimensions : 892, 0 (geometries, attributes)
extent : 98.9833, 145.665, 15.06667, 44.8194 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= coco Data Formating -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
! Response variable is considered as only presences... Is it really what you want?
! No data has been set aside for modeling evaluation
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
Checking Pseudo-absence selection arguments...
> random pseudo absences selection
> Pseudo absences are selected in explanatory variables
! Some NAs have been automatically removed from your data
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.formated.data -=-=-=-=-=-=-=-=-=-=-=-=-=-=
dir.name = Output
sp.name = coco
888 presences, 0 true absences and 2676 undefined points in dataset
6 explanatory variables
temp_max_august temp_min temp_wet_quart temp_season
Min. : 1.904 Min. :-43.356 Min. :-12.36 Min. : 25.0
1st Qu.:22.449 1st Qu.:-21.501 1st Qu.: 15.78 1st Qu.: 748.9
Median :26.610 Median : -8.816 Median : 20.08 Median : 887.8
Mean :25.505 Mean : -9.508 Mean : 19.00 Mean : 941.0
3rd Qu.:29.786 3rd Qu.: 0.245 3rd Qu.: 23.41 3rd Qu.:1274.4
Max. :40.124 Max. : 24.567 Max. : 33.86 Max. :1908.3
prec_wet_quart prec_season
Min. : 11.0 Min. : 8.873
1st Qu.: 206.0 1st Qu.: 49.039
Median : 417.0 Median : 75.961
Mean : 478.4 Mean : 76.418
3rd Qu.: 661.0 3rd Qu.:100.589
Max. :3394.0 Max. :163.496
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=On visualise les pseudo-absences :
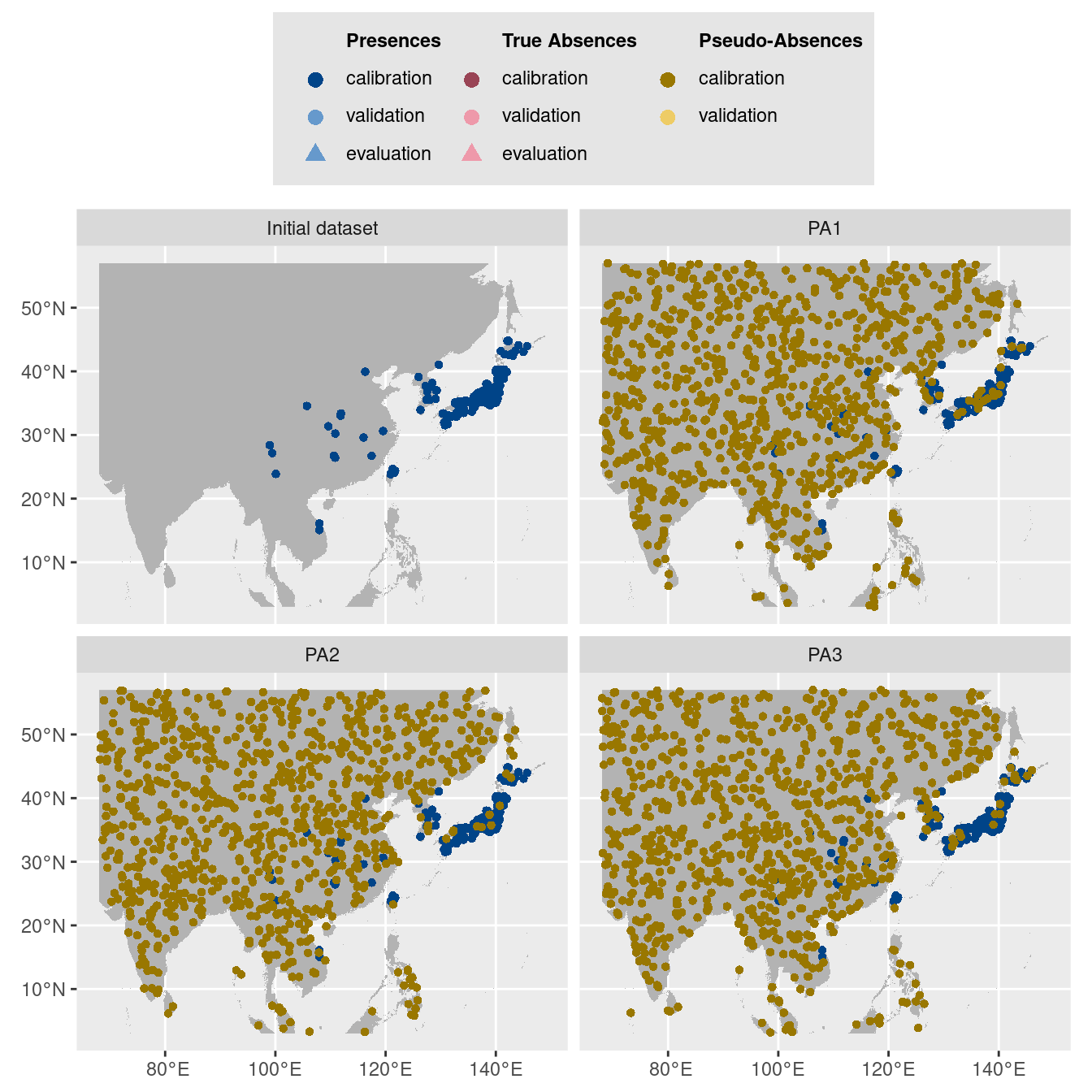
Figure 128.5: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
$data.vect
class : SpatVector
geometry : points
dimensions : 6228, 2 (geometries, attributes)
extent : 68.02083, 146.0208, 3.020833, 56.97917 (xmin, xmax, ymin, ymax)
coord. ref. :
names : resp dataset
type : <num> <chr>
values : 10 Initial dataset
10 Initial dataset
10 Initial dataset
$data.label
9 10
"**Presences**" "Presences (calibration)"
11 12
"Presences (validation)" "Presences (evaluation)"
19 20
"**True Absences**" "True Absences (calibration)"
21 22
"True Absences (validation)" "True Absences (evaluation)"
29 30
"**Pseudo-Absences**" "Pseudo-Absences (calibration)"
31 1
"Pseudo-Absences (validation)" "Background"
$data.plot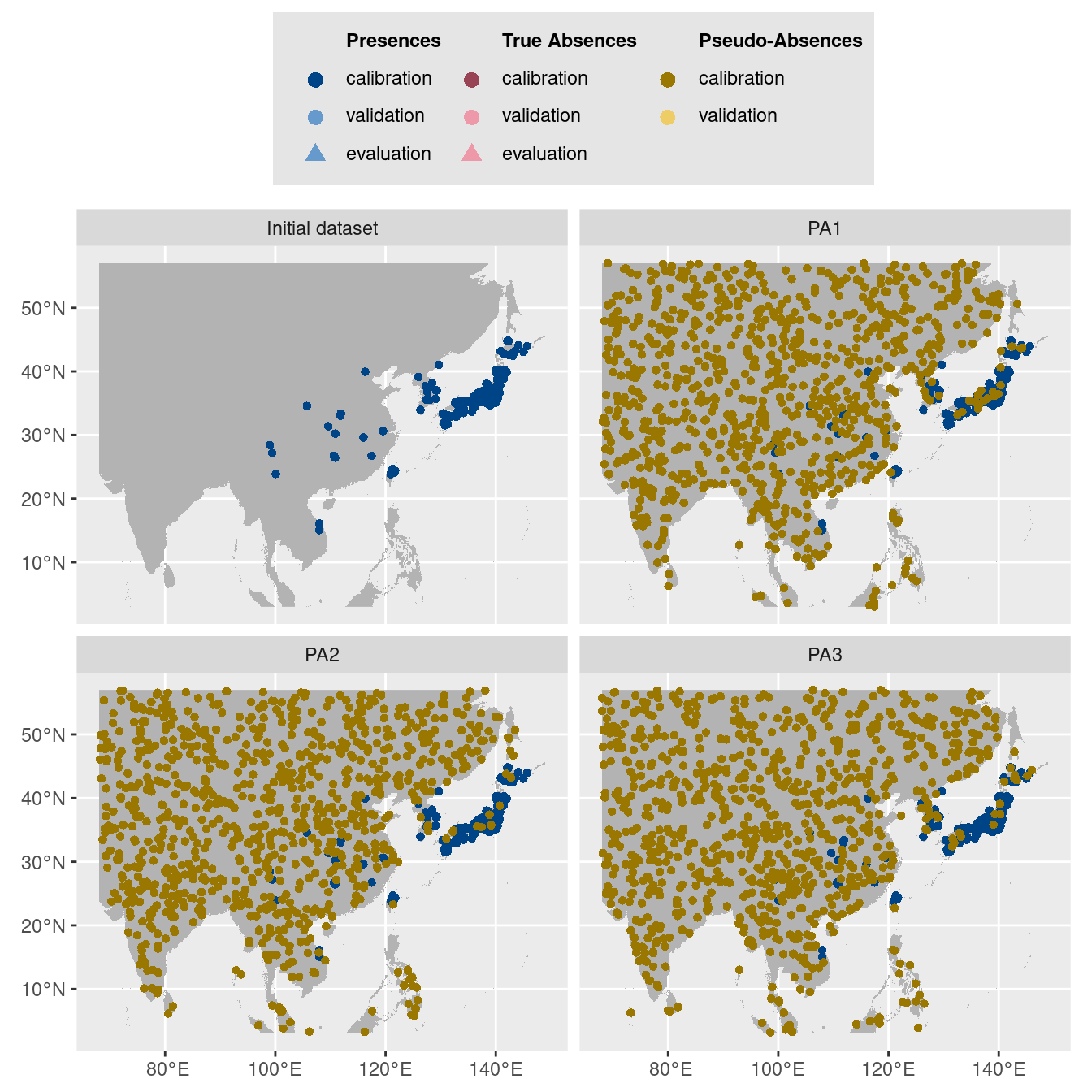
Figure 128.6: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
128.2.2 Modèles individuels de niche
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Build Single Models -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Checking Models arguments...
Creating suitable Workdir...
! Weights where automatically defined for coco_PA1 to rise a 0.5 prevalence !
! Weights where automatically defined for coco_PA2 to rise a 0.5 prevalence !
! Weights where automatically defined for coco_PA3 to rise a 0.5 prevalence !
-=-=-=-=-=-=-=-=-=-=-=-=-=-= coco Modeling Summary -=-=-=-=-=-=-=-=-=-=-=-=-=-=
6 environmental variables ( temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season )
Number of evaluation repetitions : 1
Models selected : GAM MARS MAXNET GBM ANN RF
Total number of model runs: 18
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=- Run : coco_PA1
-=-=-=--=-=-=- coco_PA1_RUN1
-=-=-=- Run : coco_PA2
-=-=-=--=-=-=- coco_PA2_RUN1
-=-=-=- Run : coco_PA3
-=-=-=--=-=-=- coco_PA3_RUN1
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=128.2.2.1 Évaluation de la performance
full.name PA run algo metric.eval cutoff sensitivity
1 coco_PA1_RUN1_GAM PA1 RUN1 GAM TSS 778 98.592
2 coco_PA1_RUN1_GAM PA1 RUN1 GAM ROC 783 98.592
3 coco_PA1_RUN1_MARS PA1 RUN1 MARS TSS 515 97.465
4 coco_PA1_RUN1_MARS PA1 RUN1 MARS ROC 519 97.465
5 coco_PA1_RUN1_MAXNET PA1 RUN1 MAXNET TSS 354 96.479
6 coco_PA1_RUN1_MAXNET PA1 RUN1 MAXNET ROC 354 96.479
specificity calibration validation evaluation
1 70.588 0.692 0.669 NA
2 70.728 0.766 0.761 NA
3 95.798 0.933 0.910 NA
4 95.798 0.989 0.989 NA
5 95.098 0.916 0.933 NA
6 95.098 0.988 0.988 NA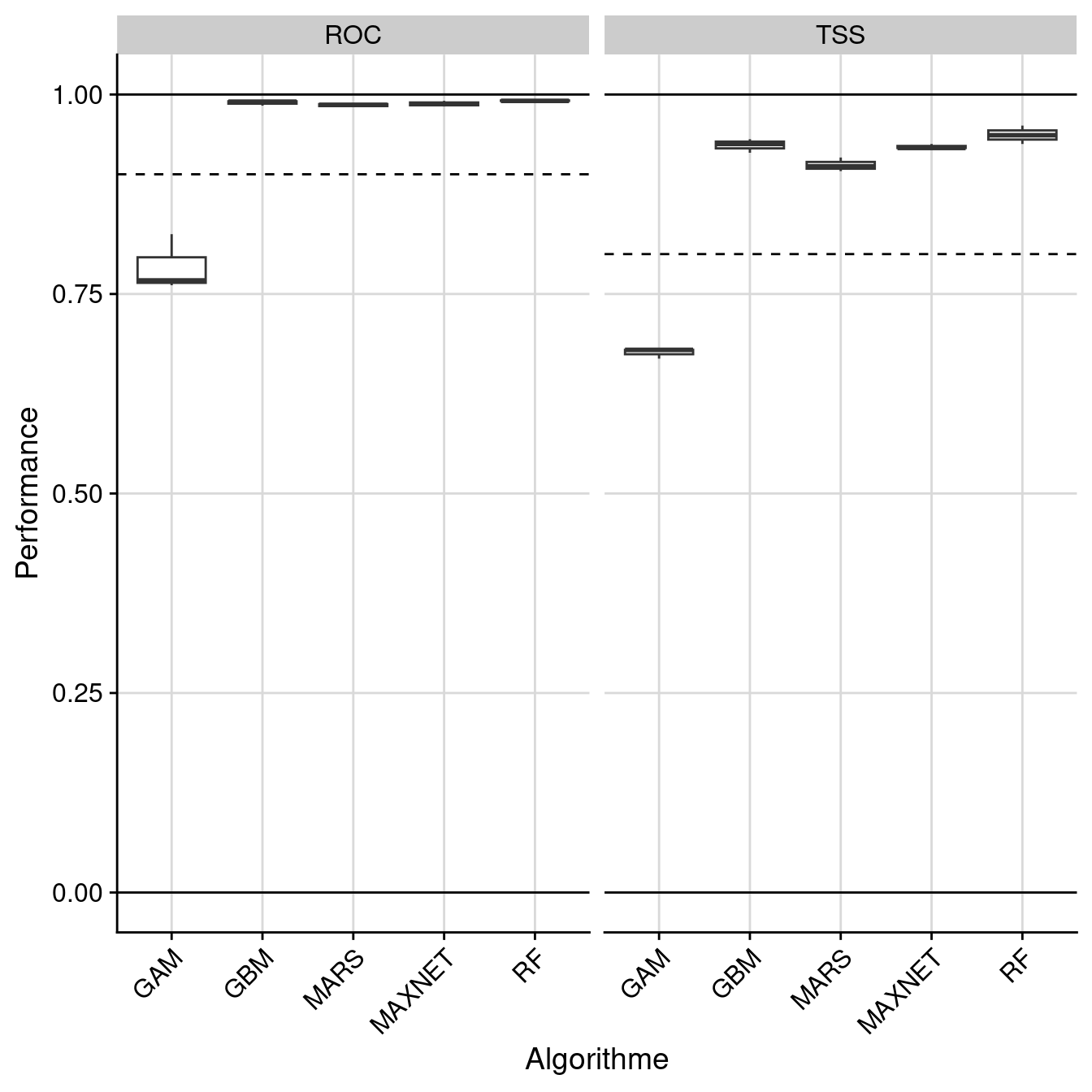
Figure 128.7: Évaluation de la performance des modèles individuels : True Skill Statistic & aire sous la courbe de ROC.
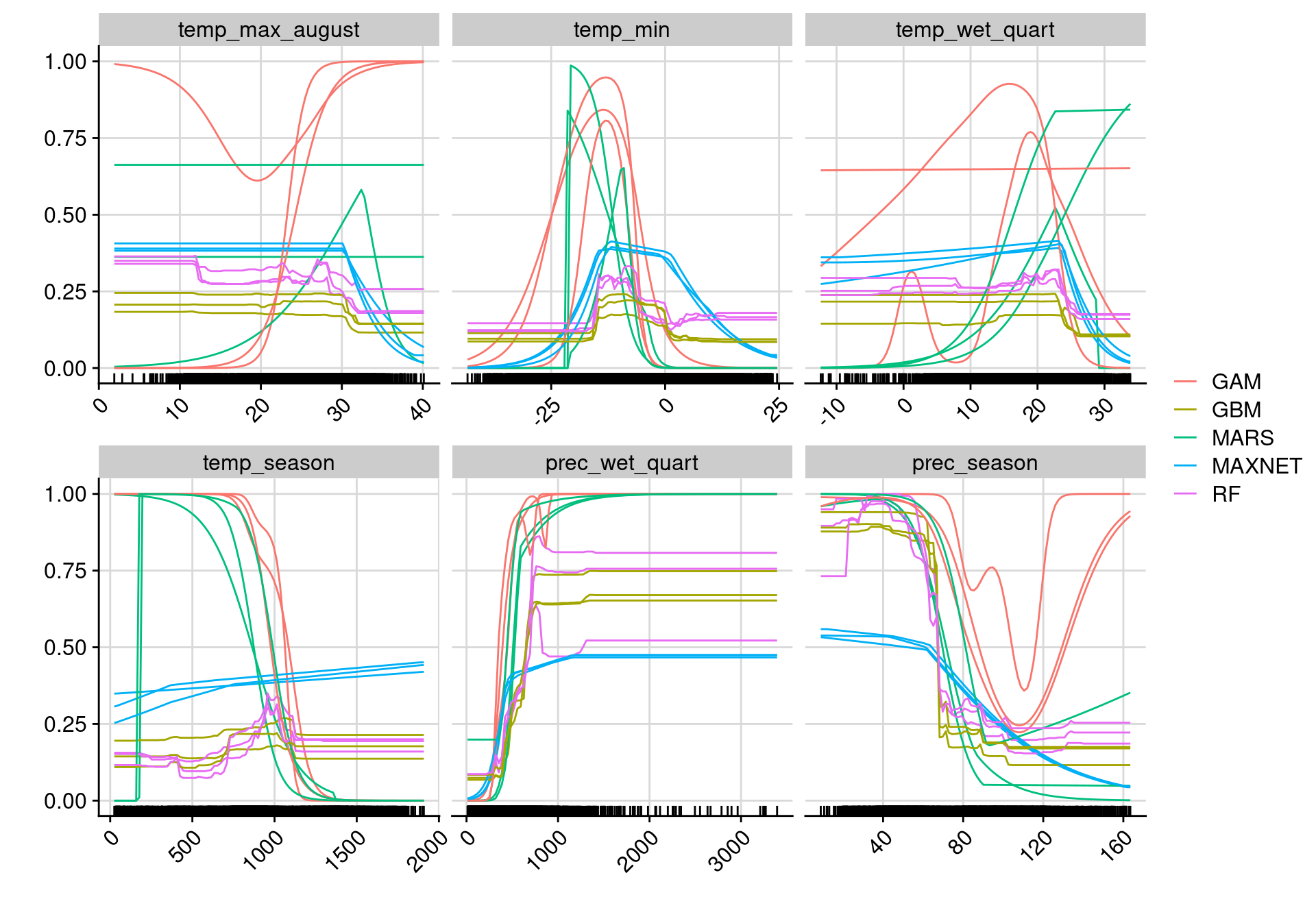
128.2.2.2 Importance des variables environnementales
full.name PA run algo expl.var rand var.imp
1 coco_PA1_RUN1_GAM PA1 RUN1 GAM temp_max_august 1 0.010466
2 coco_PA1_RUN1_GAM PA1 RUN1 GAM temp_min 1 0.094565
3 coco_PA1_RUN1_GAM PA1 RUN1 GAM temp_wet_quart 1 0.046847
4 coco_PA1_RUN1_GAM PA1 RUN1 GAM temp_season 1 0.426281
5 coco_PA1_RUN1_GAM PA1 RUN1 GAM prec_wet_quart 1 0.380761
6 coco_PA1_RUN1_GAM PA1 RUN1 GAM prec_season 1 0.129268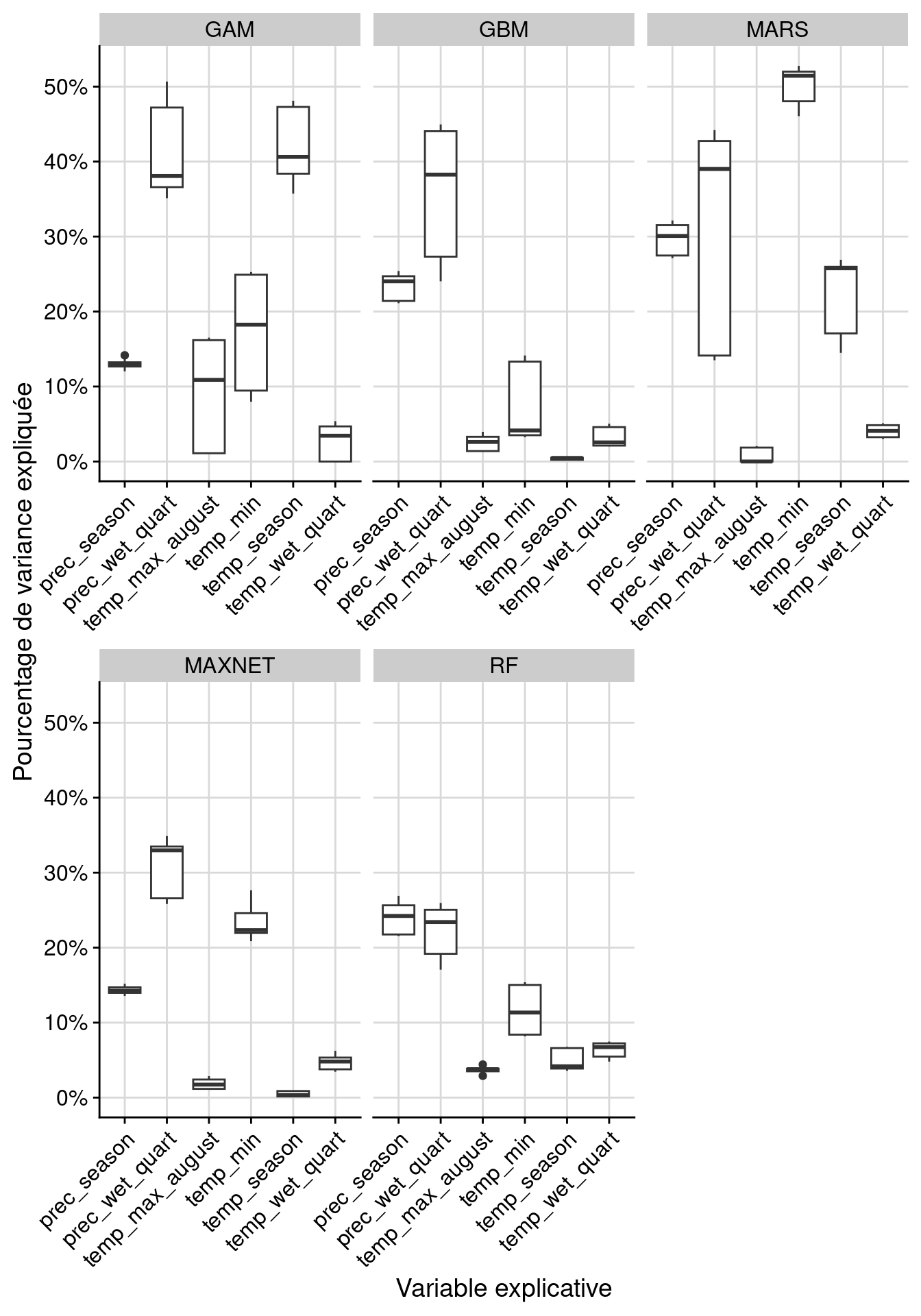
Figure 128.8: Pourcentage de variance expliquée pour chaque variable, décomposé par algorithme.
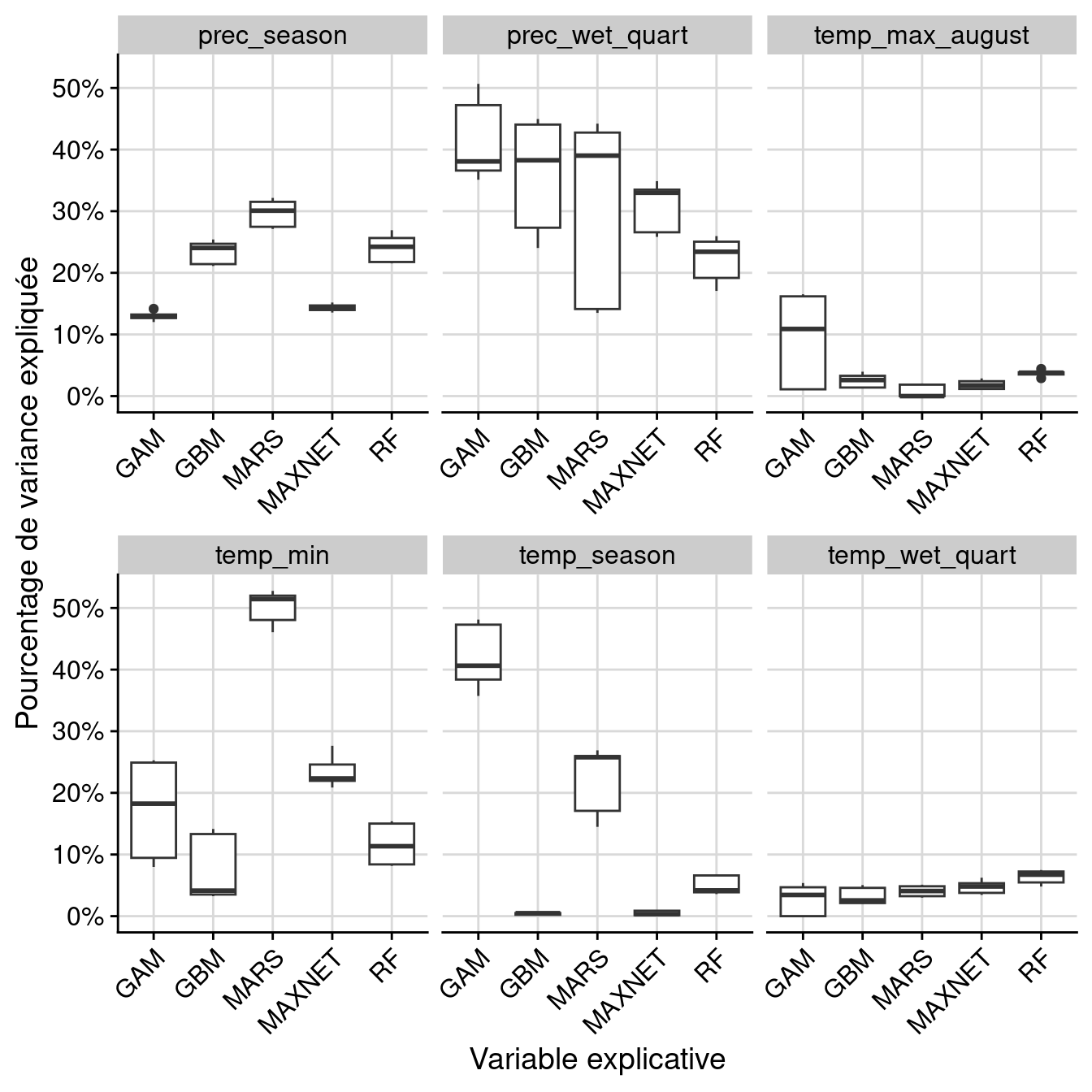
Figure 128.9: Pourcentage de variance expliquée pour chaque variable, décomposé par variable.
128.2.3 Modèles d’ensemble de niche
Seulement modèles avec TSS > 0.8
-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.ensemble.models.out -=-=-=-=-=-=-=-=-=-=-=-=-=
sp.name : coco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
models computed:
coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, coco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
models failed: none
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=128.2.3.1 Évaluation de la qualité
full.name merged.by.PA merged.by.run
1 coco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
2 coco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
merged.by.algo filtered.by algo metric.eval cutoff sensitivity specificity
1 mergedAlgo TSS EMwmean TSS 538.0 97.523 97.048
2 mergedAlgo TSS EMwmean ROC 569.5 97.410 97.235
calibration validation evaluation
1 0.946 NA NA
2 0.996 NA NA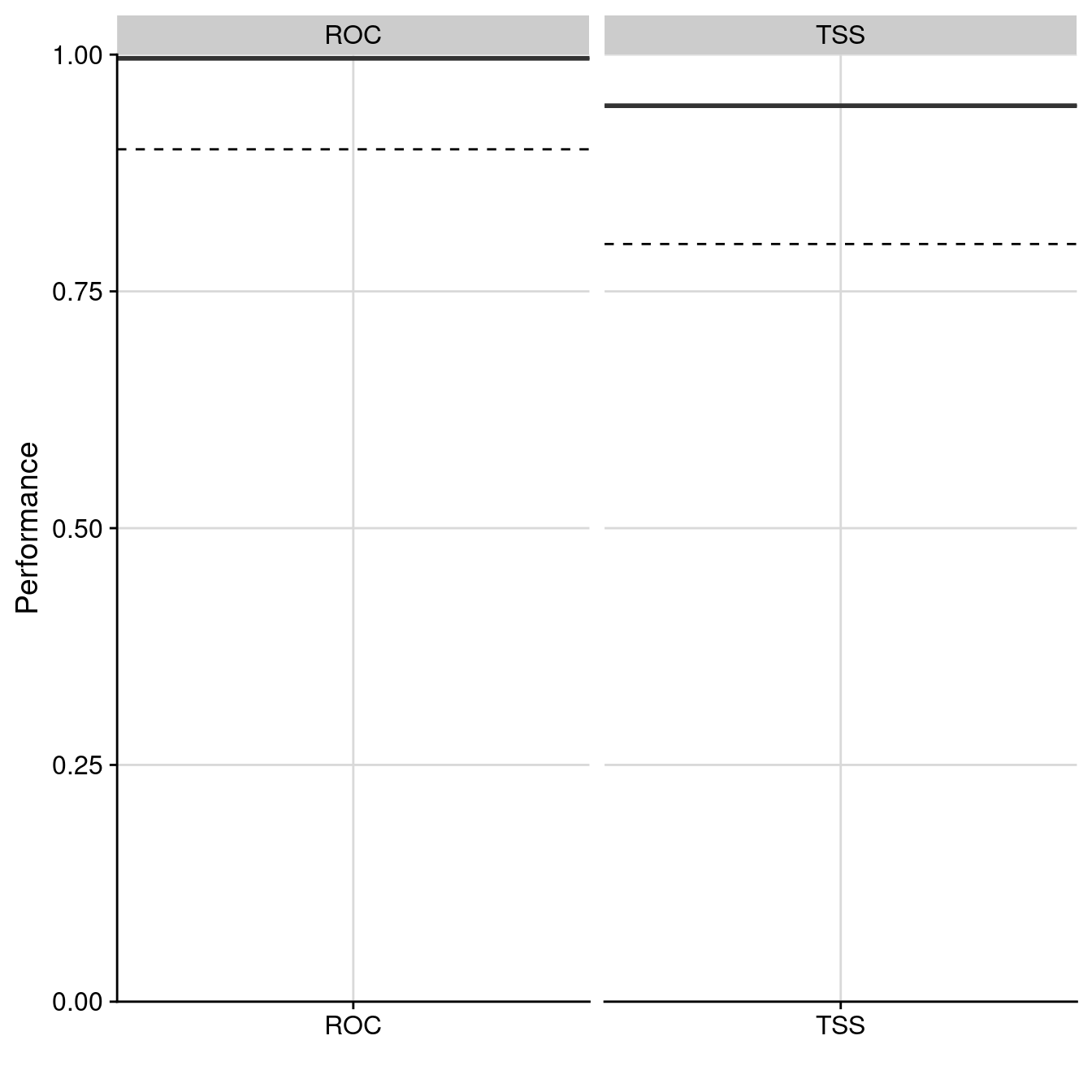
Figure 128.11: Évaluation de la qualité du modèle d’ensemble : Aire sous la courbe de ROC & True Skill Statistic.
full.name merged.by.PA merged.by.run
1 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
2 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
3 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
4 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
5 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
6 coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
merged.by.algo filtered.by algo expl.var rand var.imp
1 mergedAlgo TSS EMcv temp_max_august 1 0.061001
2 mergedAlgo TSS EMcv temp_min 1 0.362329
3 mergedAlgo TSS EMcv temp_wet_quart 1 0.101258
4 mergedAlgo TSS EMcv temp_season 1 0.154075
5 mergedAlgo TSS EMcv prec_wet_quart 1 0.337738
6 mergedAlgo TSS EMcv prec_season 1 0.128728Par variable :
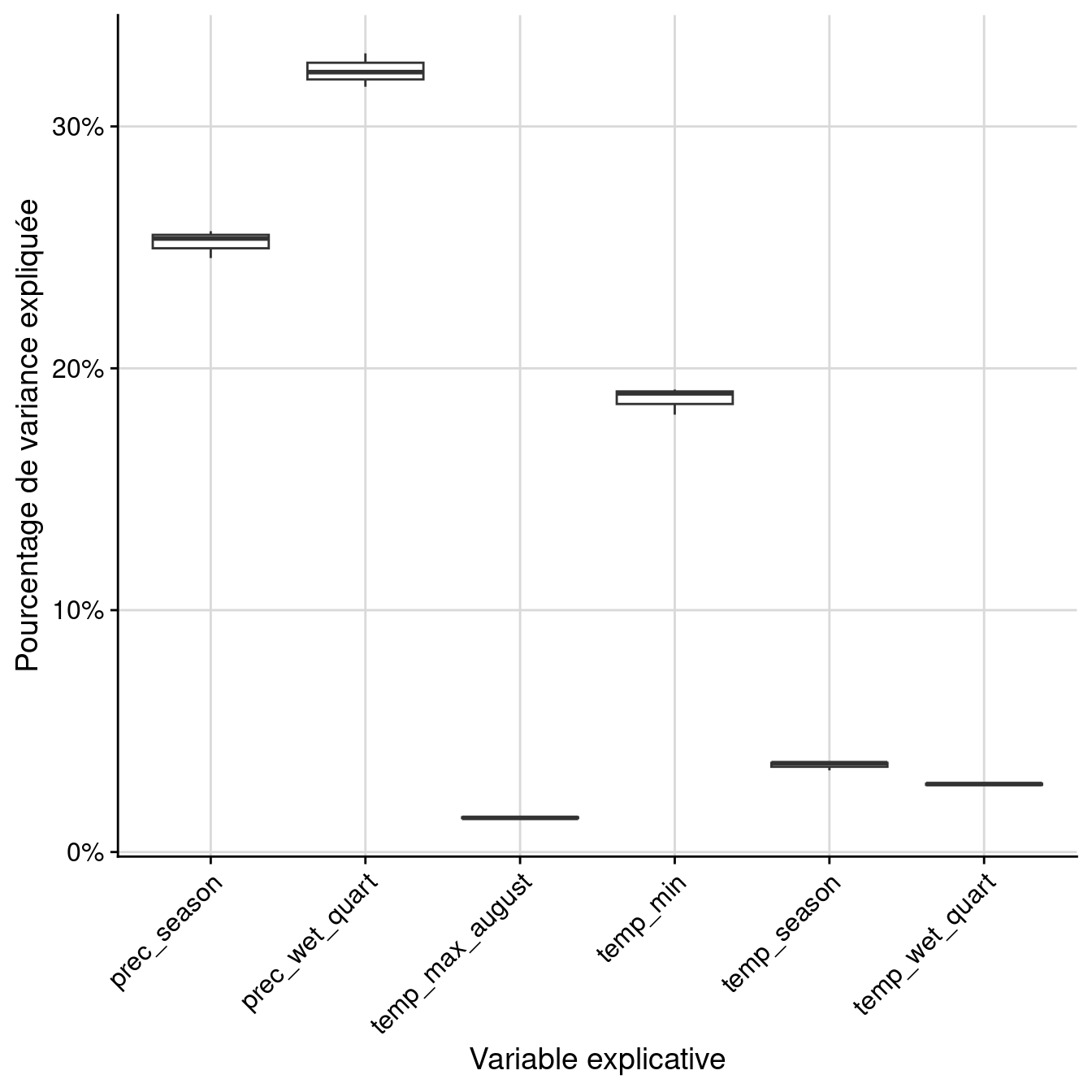
Figure 128.12: Pourcentage de variance expliquée par variable dans le modèle d’ensemble.
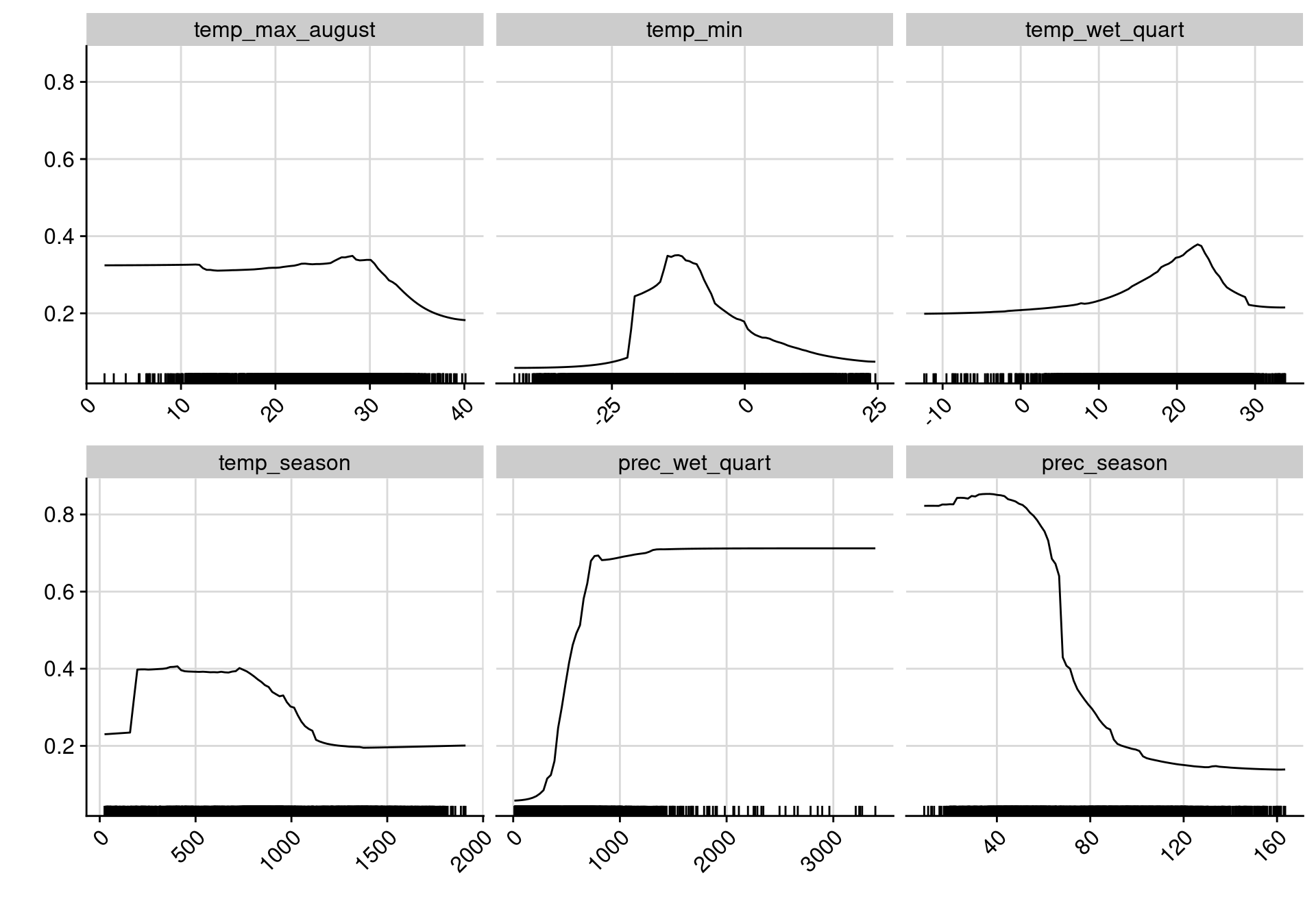
Figure 128.13: Courbes de réponse de chaque variable climatique dans le modèle d’ensemble.
128.3 Projections
128.3.1 Distribution potentielle contemporaine
Contemporaine : Cartographie des observations sur couches climatiques Extraction des conditions climatiques favorables
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/coco/current
sp.name : coco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/coco/coco.AllModels.models.out )
models.projected :
coco_PA1_RUN1_GAM, coco_PA1_RUN1_MARS, coco_PA1_RUN1_MAXNET, coco_PA1_RUN1_GBM, coco_PA1_RUN1_RF, coco_PA2_RUN1_GAM, coco_PA2_RUN1_MARS, coco_PA2_RUN1_MAXNET, coco_PA2_RUN1_GBM, coco_PA2_RUN1_RF, coco_PA3_RUN1_GAM, coco_PA3_RUN1_MARS, coco_PA3_RUN1_MAXNET, coco_PA3_RUN1_GBM, coco_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=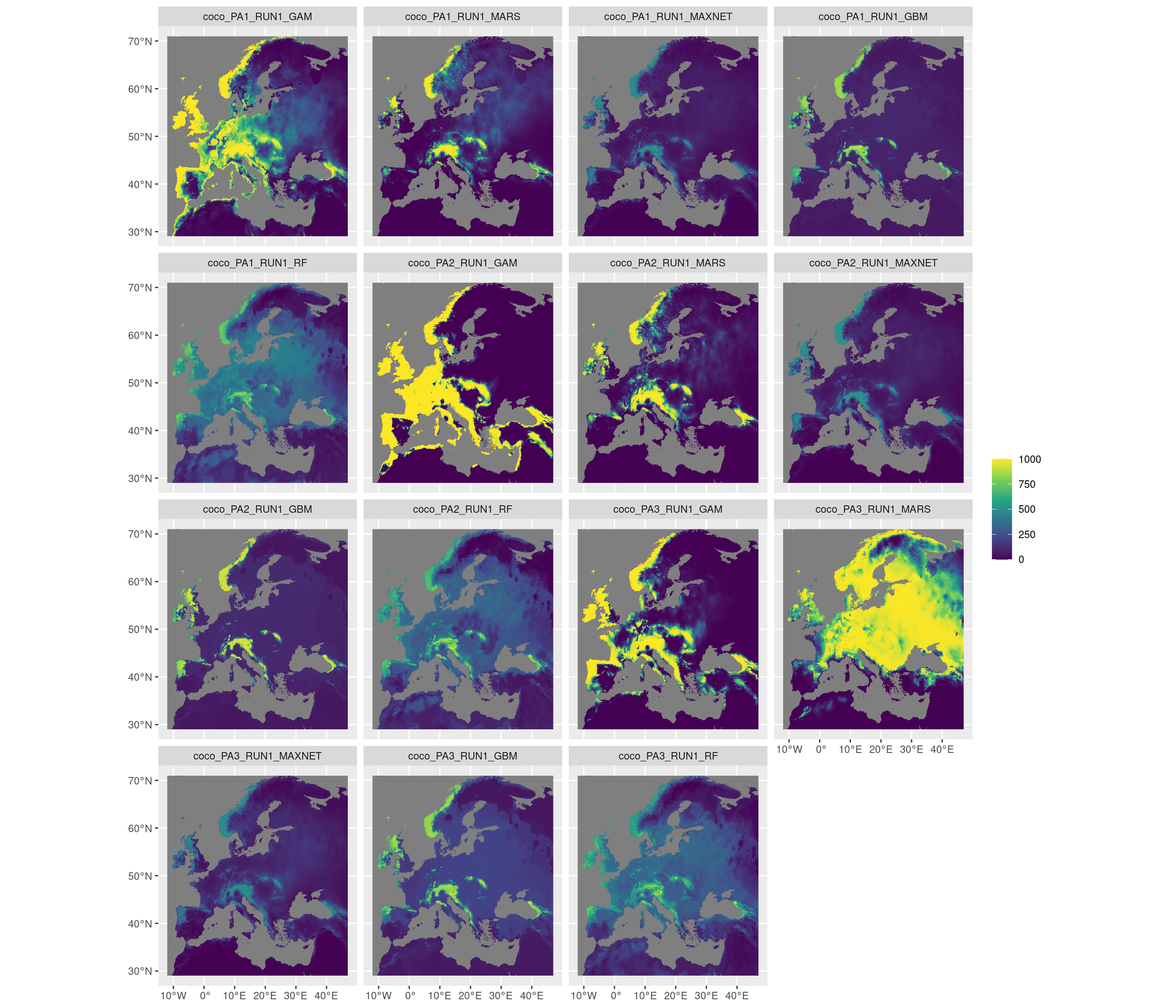
Figure 128.14: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
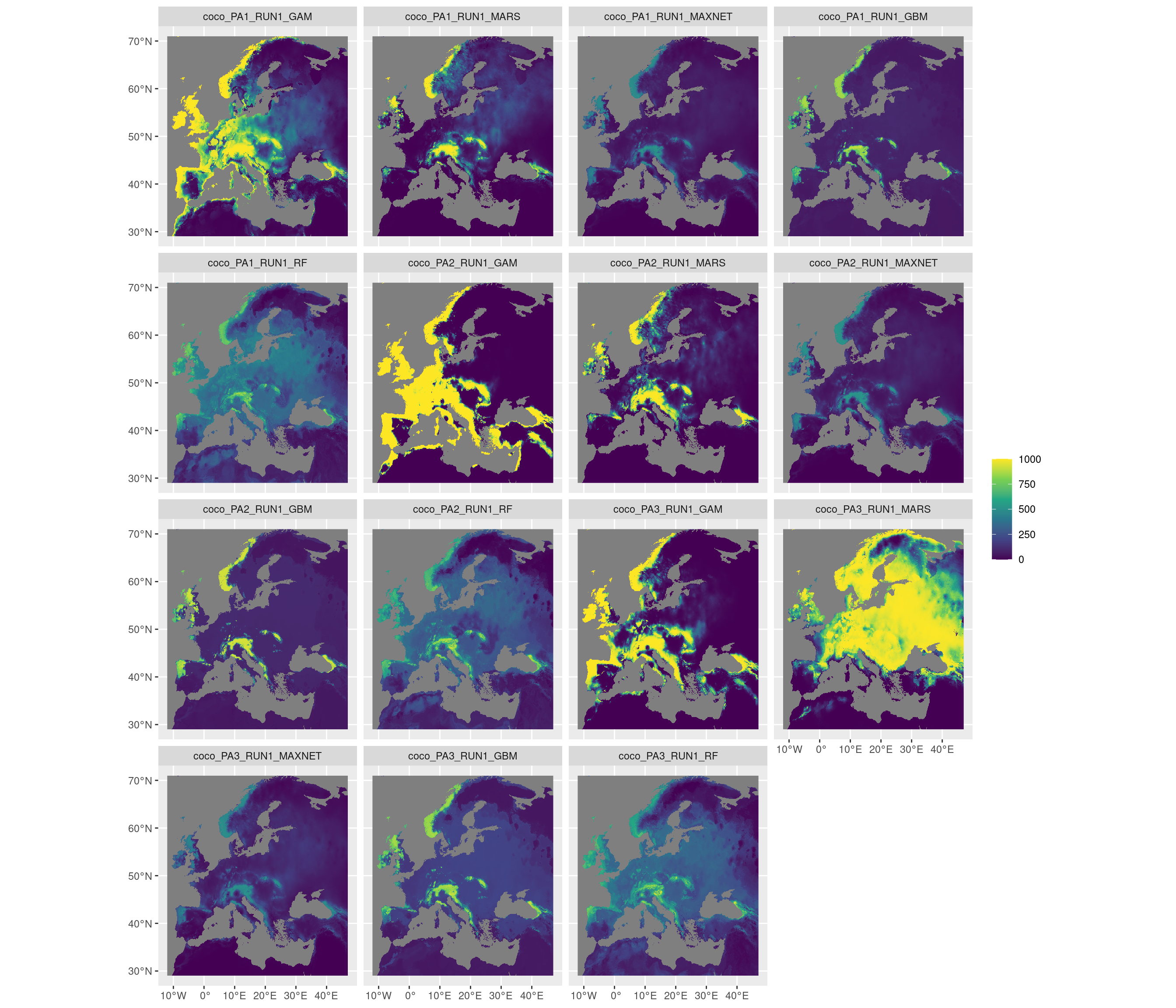
Figure 128.15: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/coco/current
sp.name : coco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/coco/coco.AllModels.ensemble.models.out )
models.projected :
coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, coco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=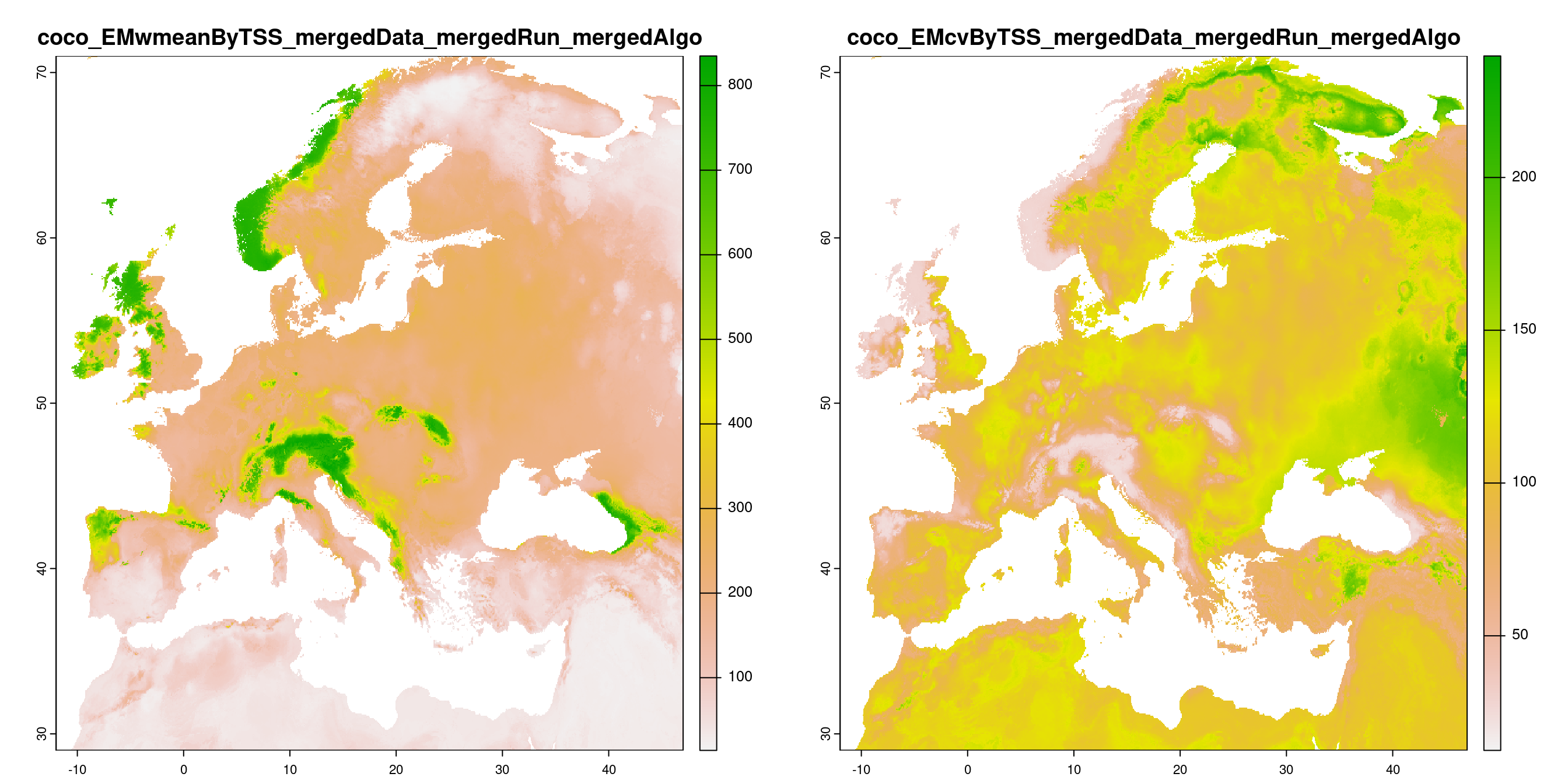
Figure 128.16: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) ; B) Incertitude associée à la projection.
128.3.2 Interpolation à l’échelle de la métropole
- → conditions climatiques favorables à fine échelle
On projette la niche climatique sur ce raster à fine échelle.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/coco/cont_gre
sp.name : coco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/coco/coco.AllModels.models.out )
models.projected :
coco_PA1_RUN1_GAM, coco_PA1_RUN1_MARS, coco_PA1_RUN1_MAXNET, coco_PA1_RUN1_GBM, coco_PA1_RUN1_RF, coco_PA2_RUN1_GAM, coco_PA2_RUN1_MARS, coco_PA2_RUN1_MAXNET, coco_PA2_RUN1_GBM, coco_PA2_RUN1_RF, coco_PA3_RUN1_GAM, coco_PA3_RUN1_MARS, coco_PA3_RUN1_MAXNET, coco_PA3_RUN1_GBM, coco_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=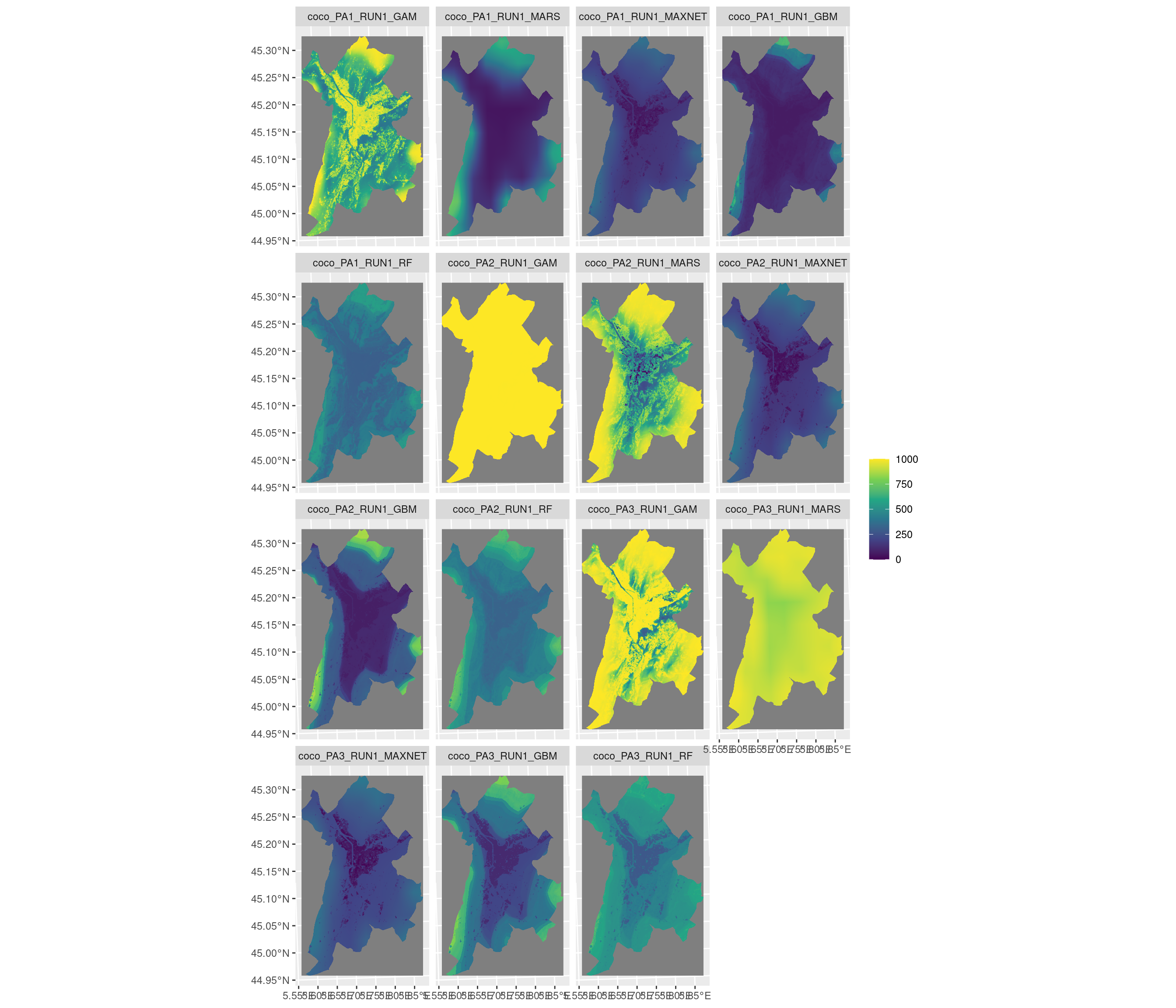
Figure 128.17: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
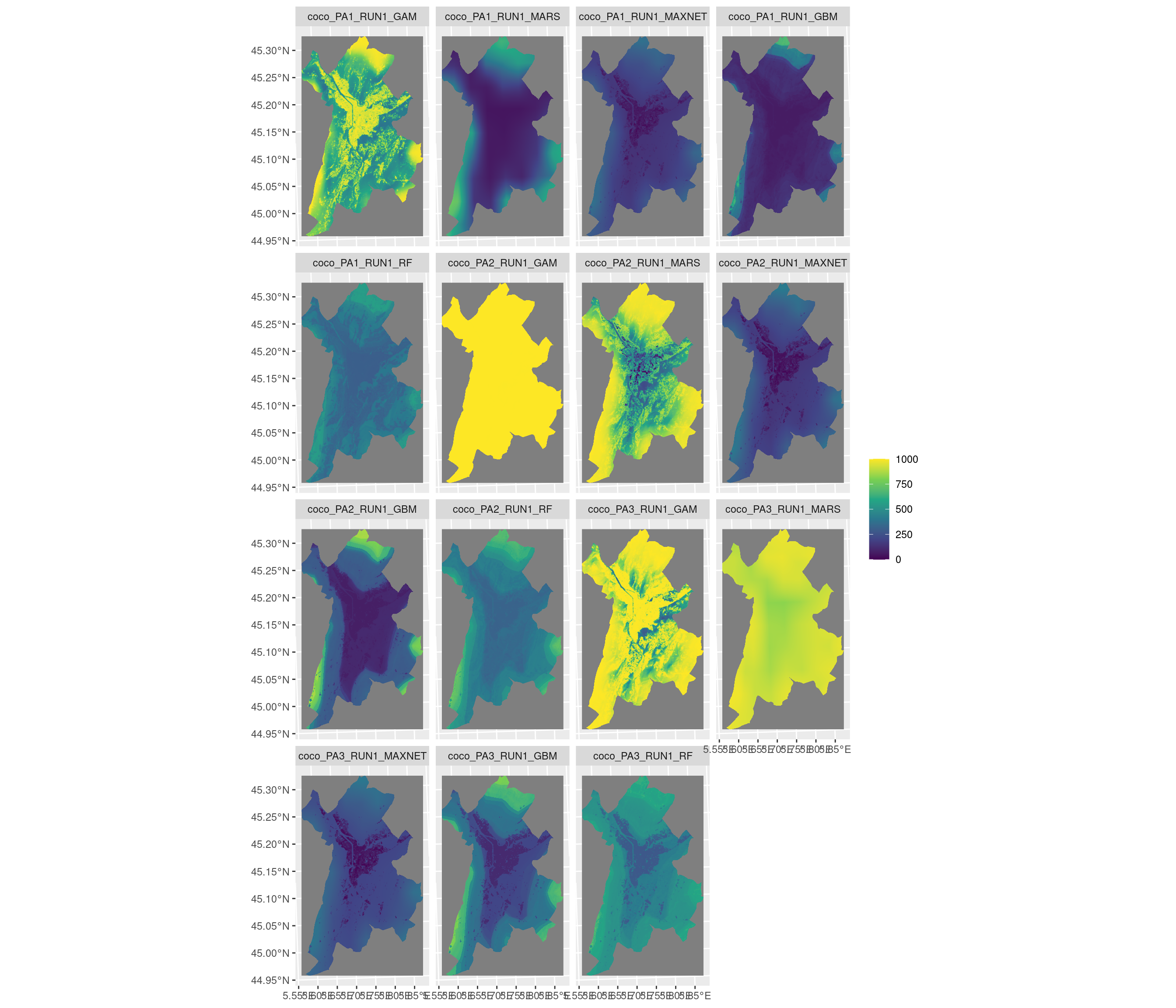
Figure 128.18: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/coco/cont_gre
sp.name : coco
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/coco/coco.AllModels.ensemble.models.out )
models.projected :
coco_EMcvByTSS_mergedData_mergedRun_mergedAlgo, coco_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=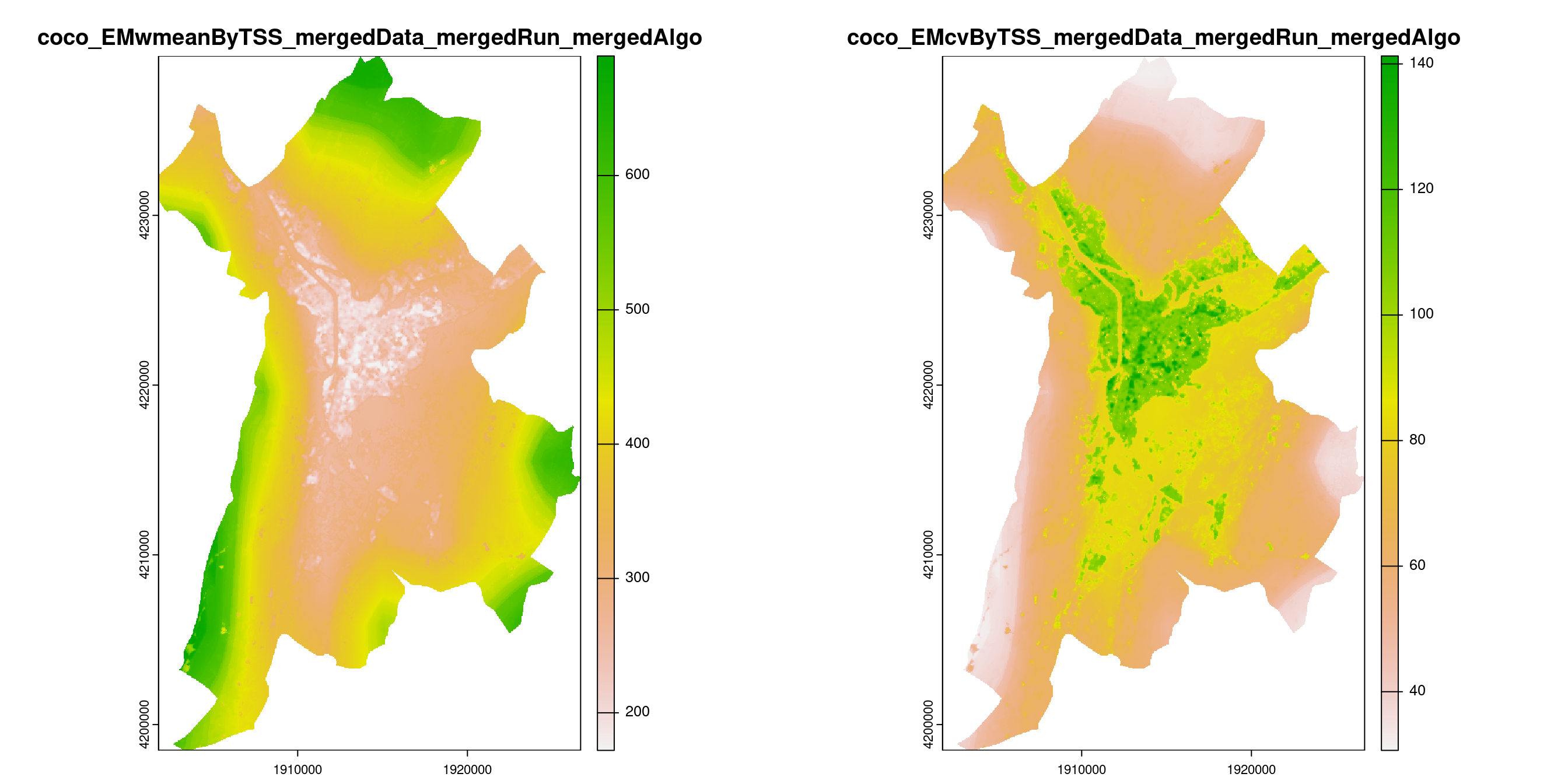
Figure 128.19: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) sur la métropole de Grenoble ; B) Incertitude associée à la projection.
128.3.3 Distributions potentielles futures
Futur : Projection à l’échelle de l’Europe jusqu’à l’horizon 2100
Toutes les fenêtres temporelles × scénarios pour le GCM « IPSL-CM6A-LR »
- SSP1-2.6
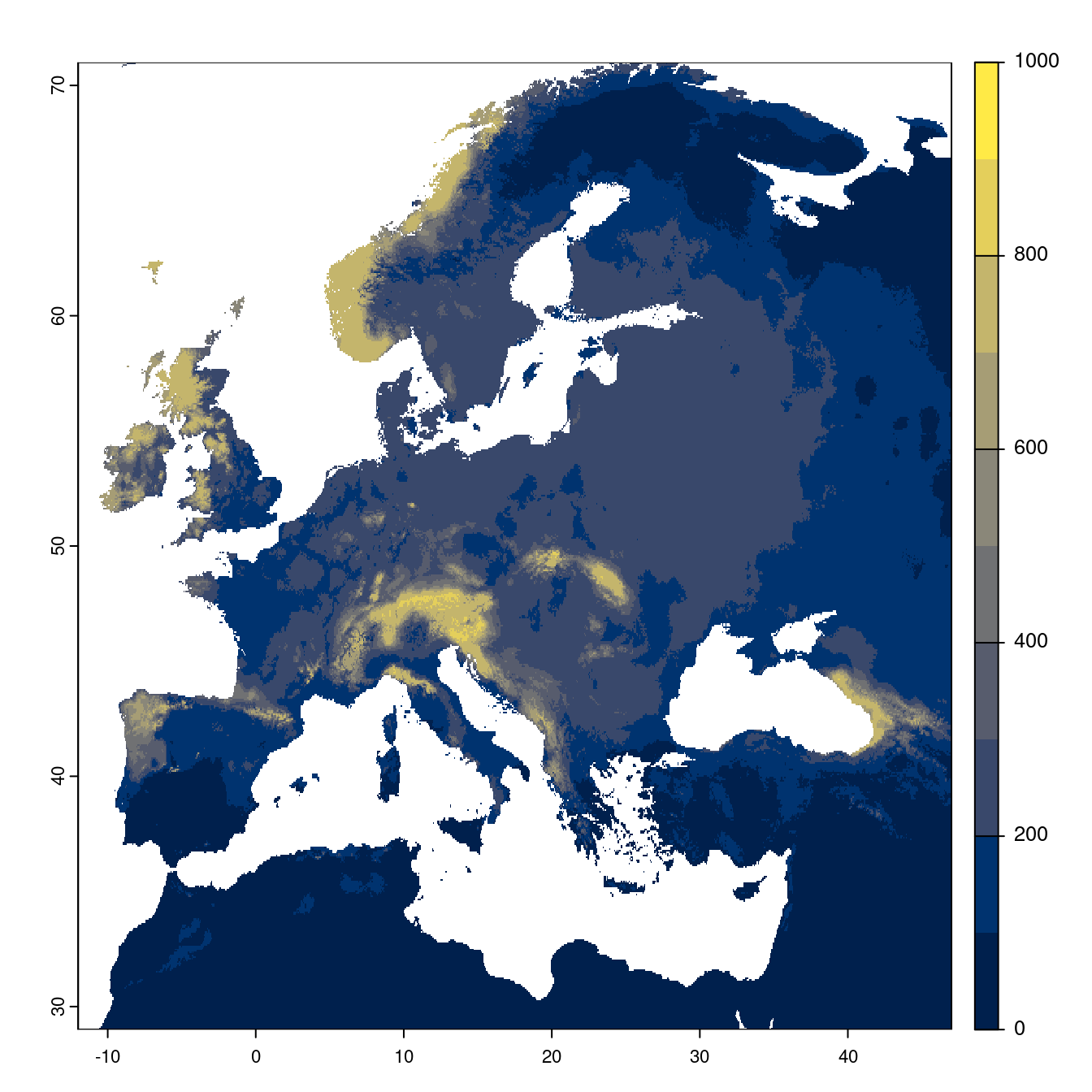
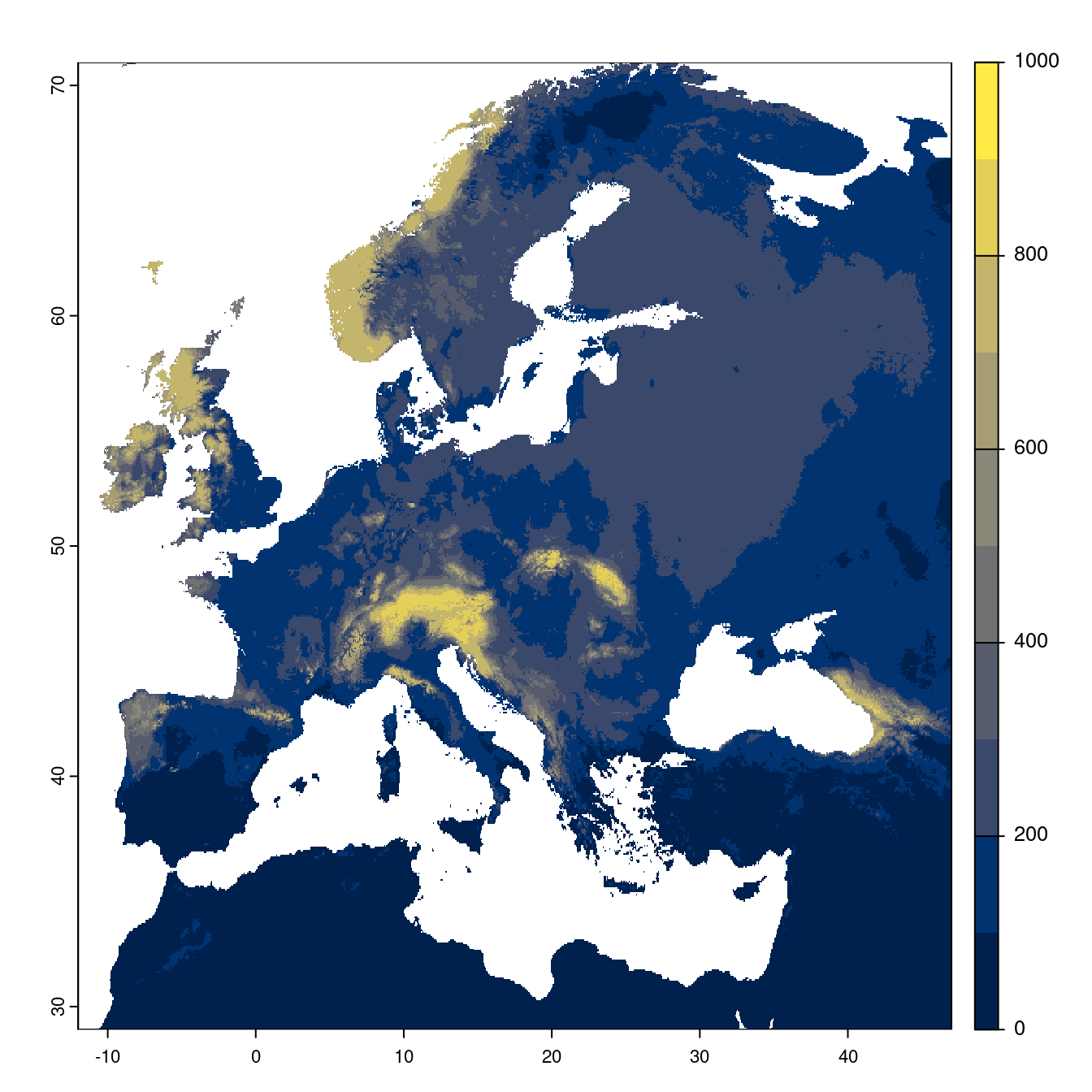
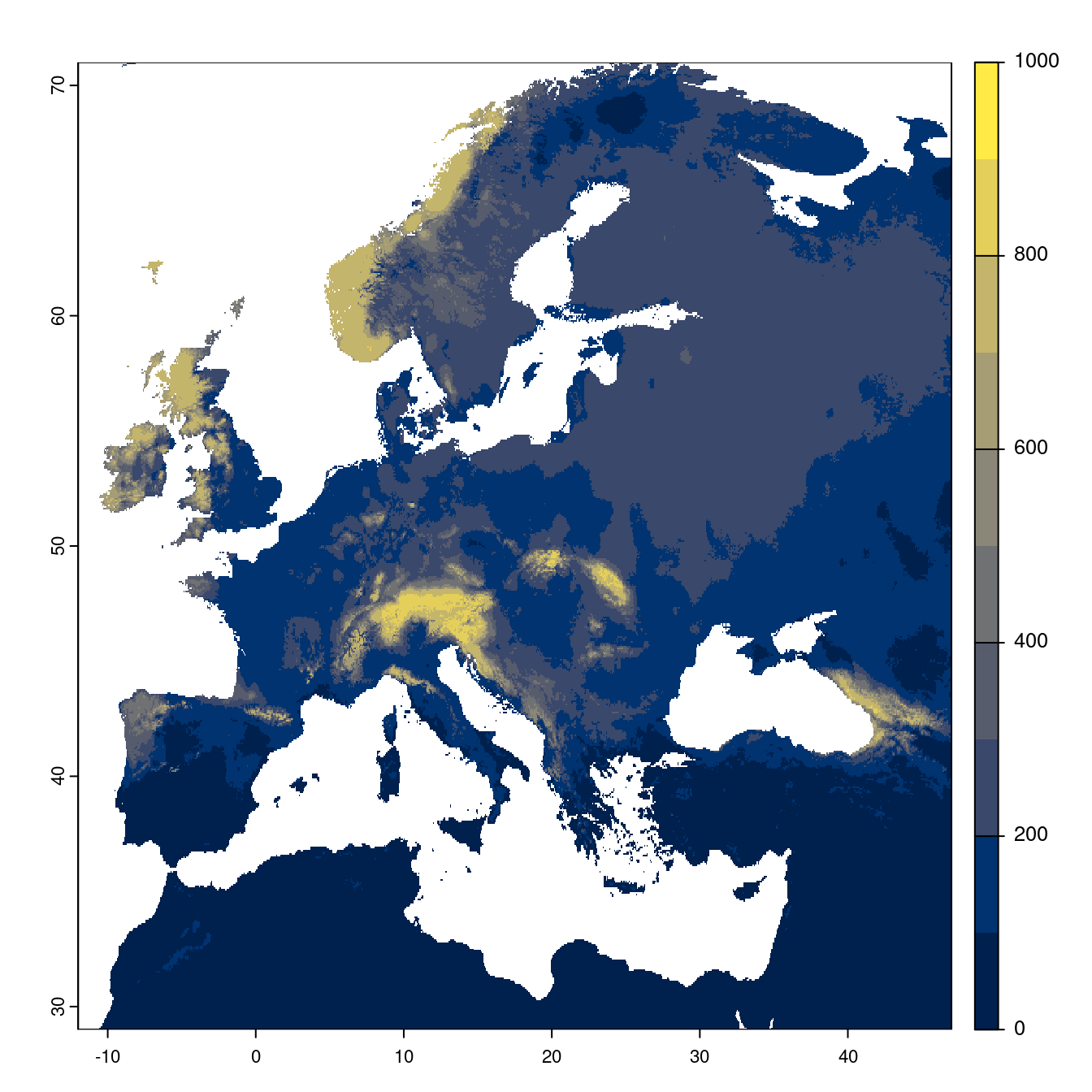
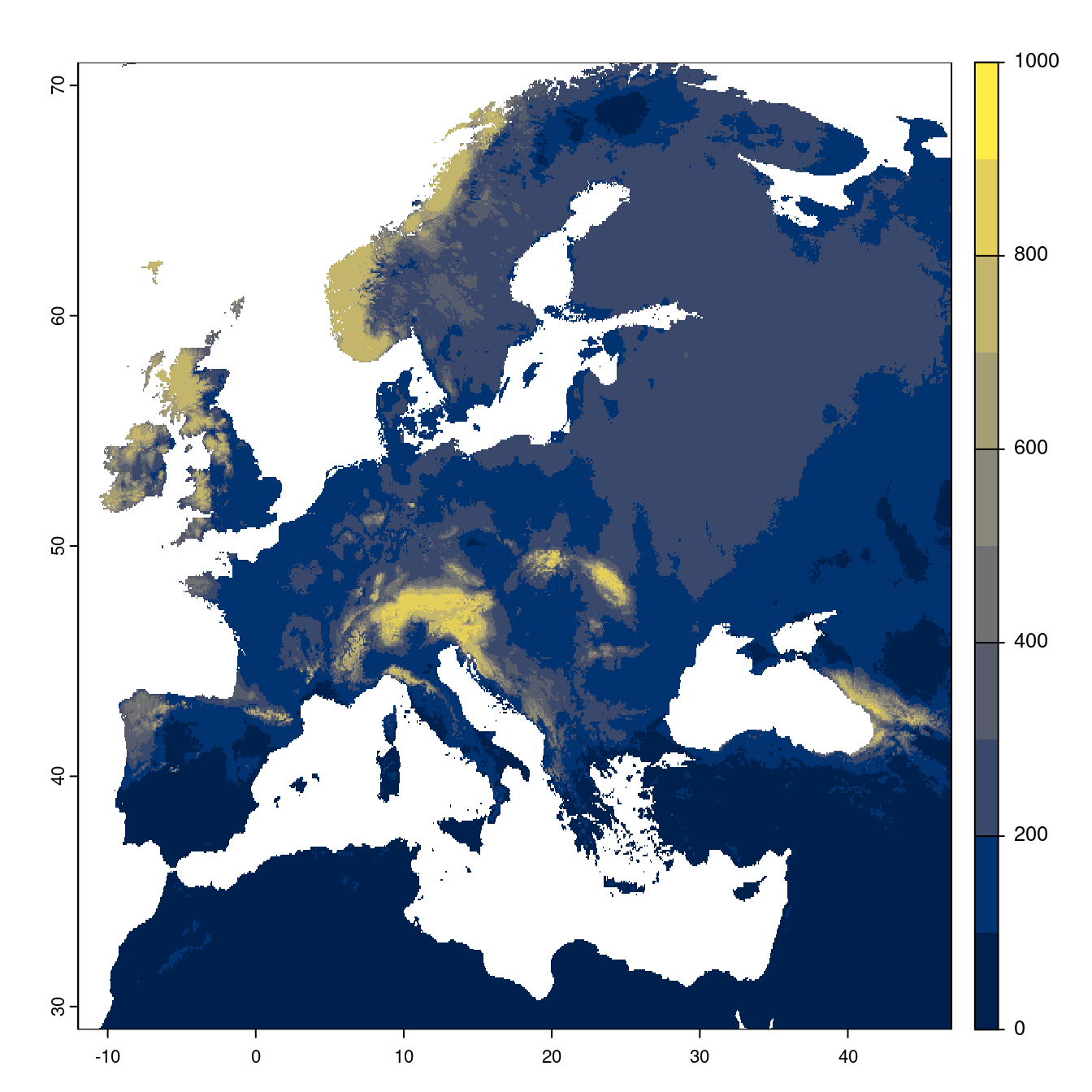
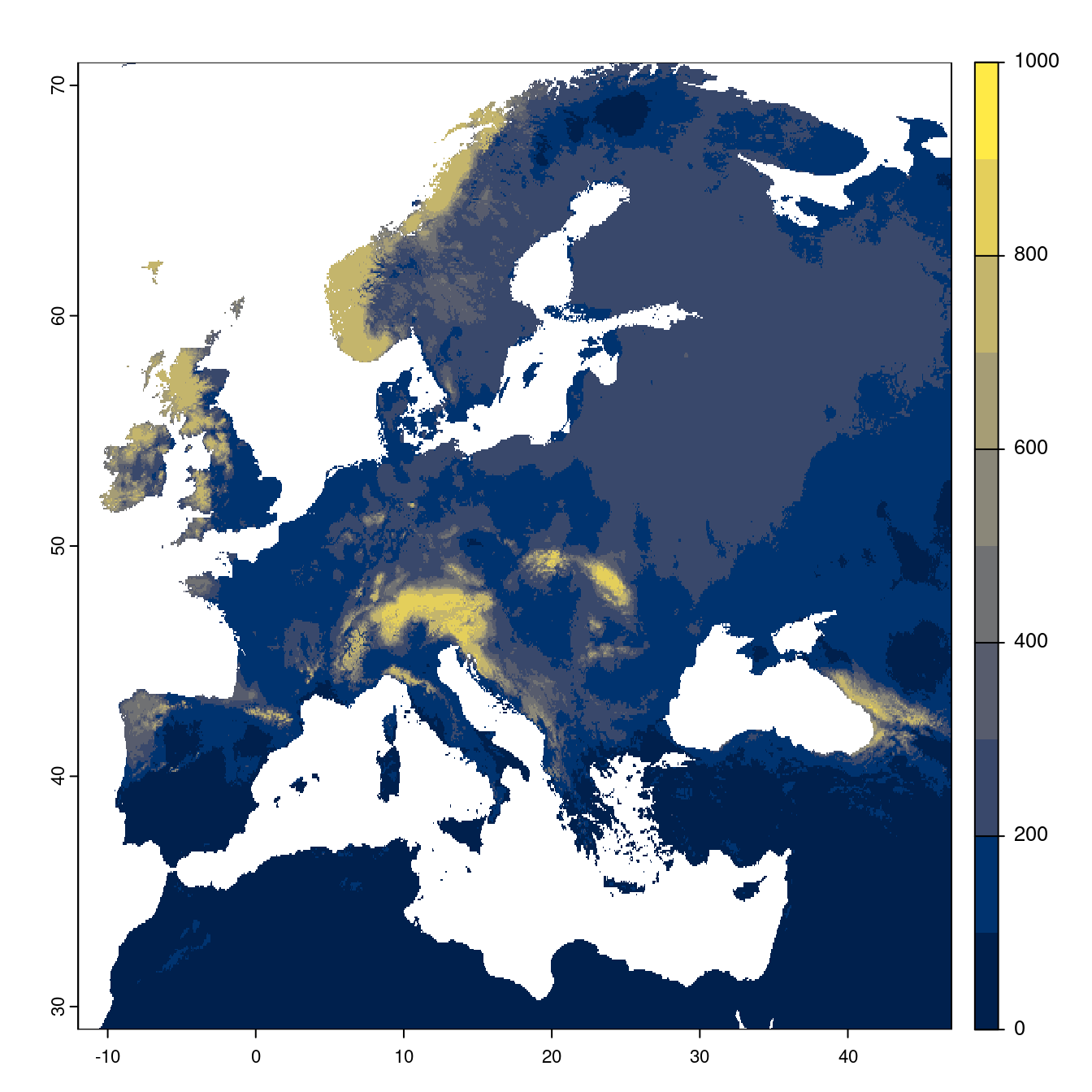
Figure 128.20: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP1-2.6 (modèle d’ensemble).
On obtient pour la métrique TSS une carte unique de moyenne pondérée sur les algorithmes et sur les jeux de pseudo-absences.
- SSP2-4.5
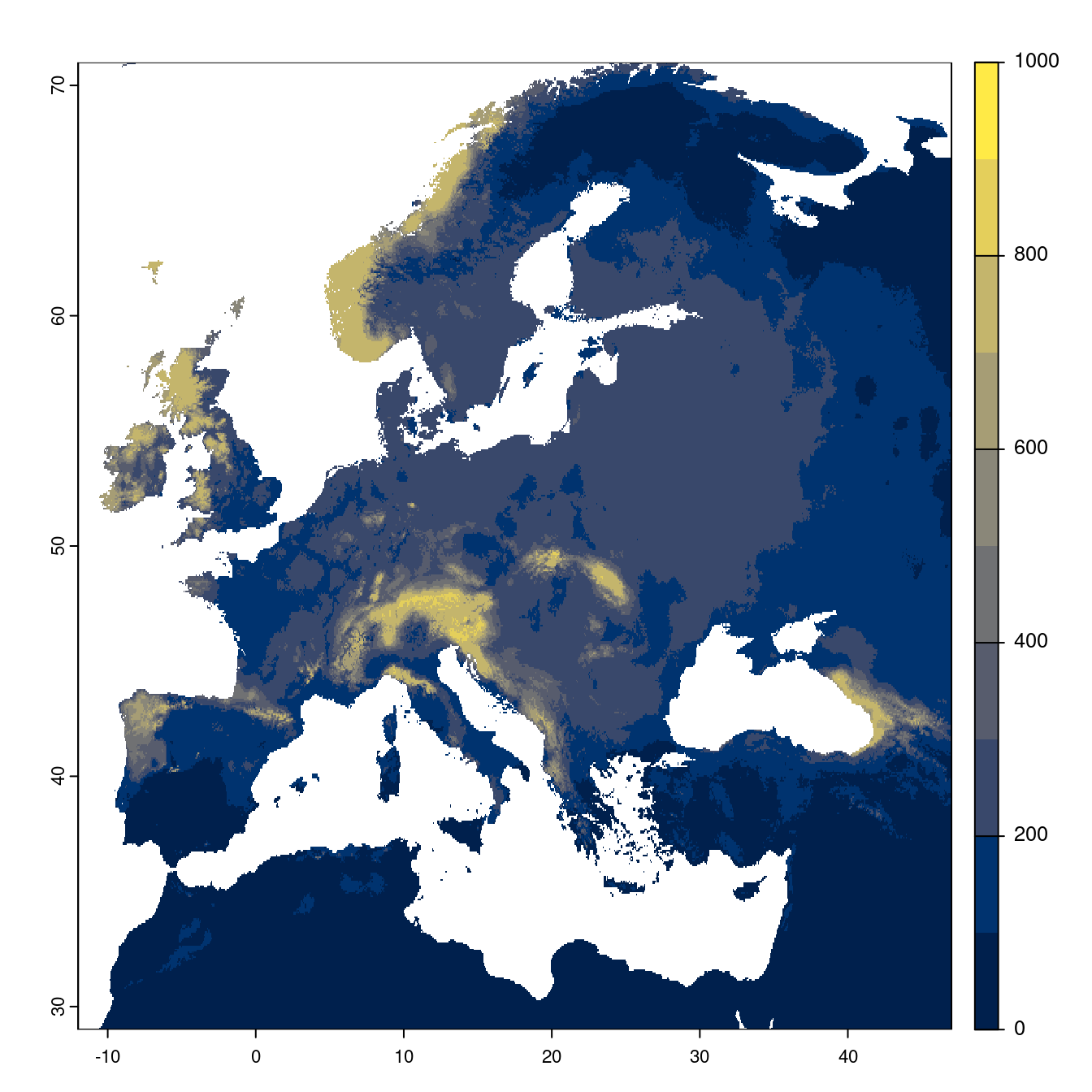
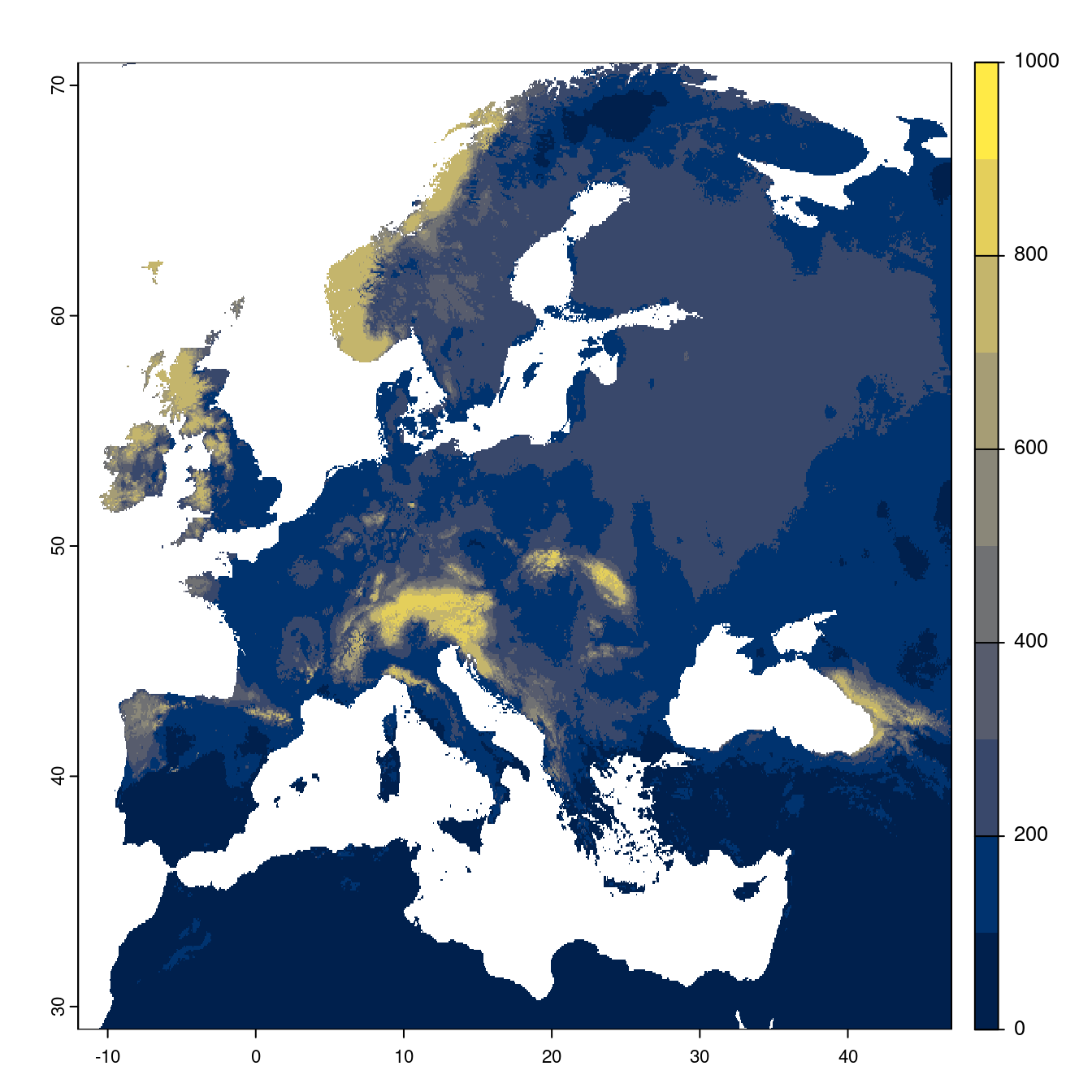
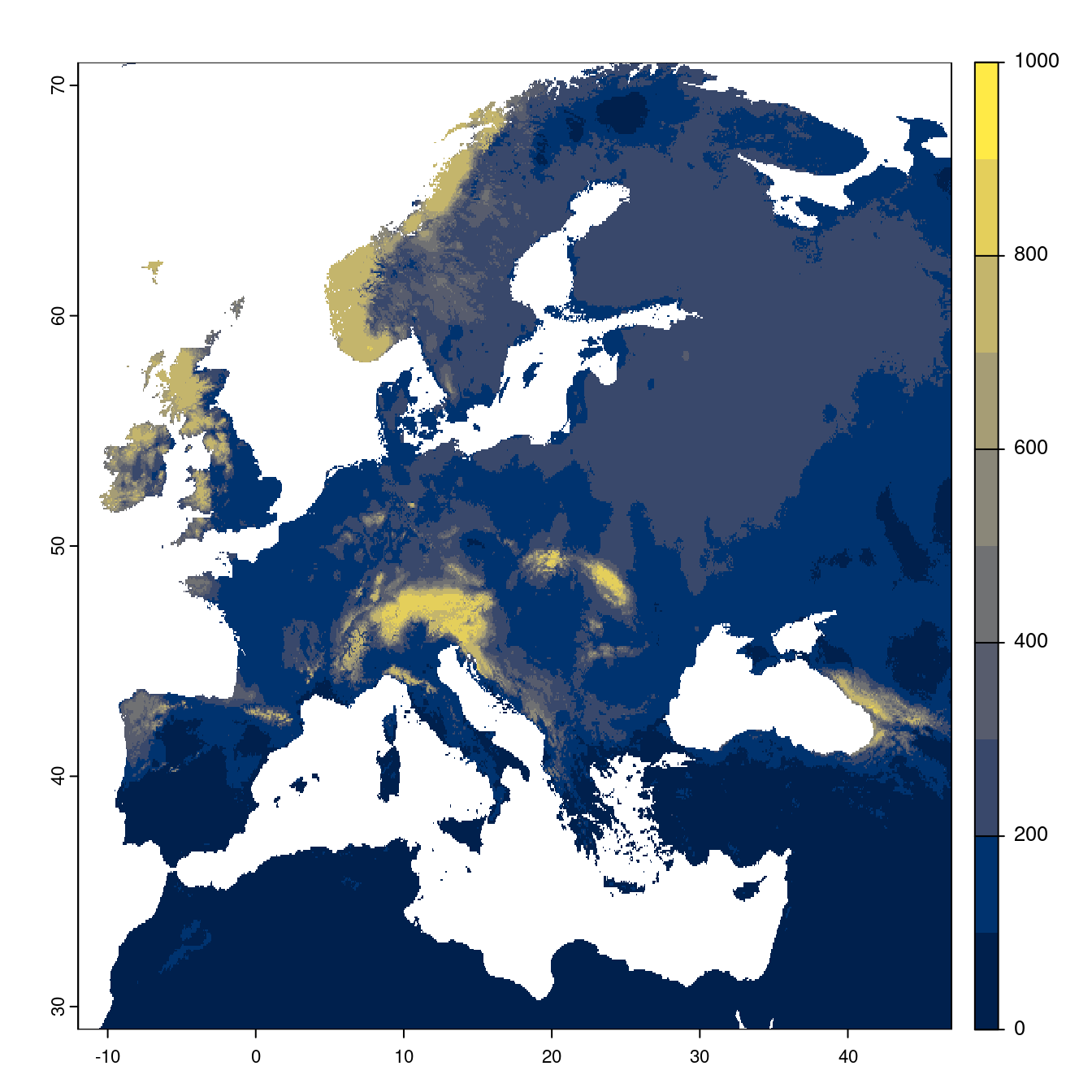
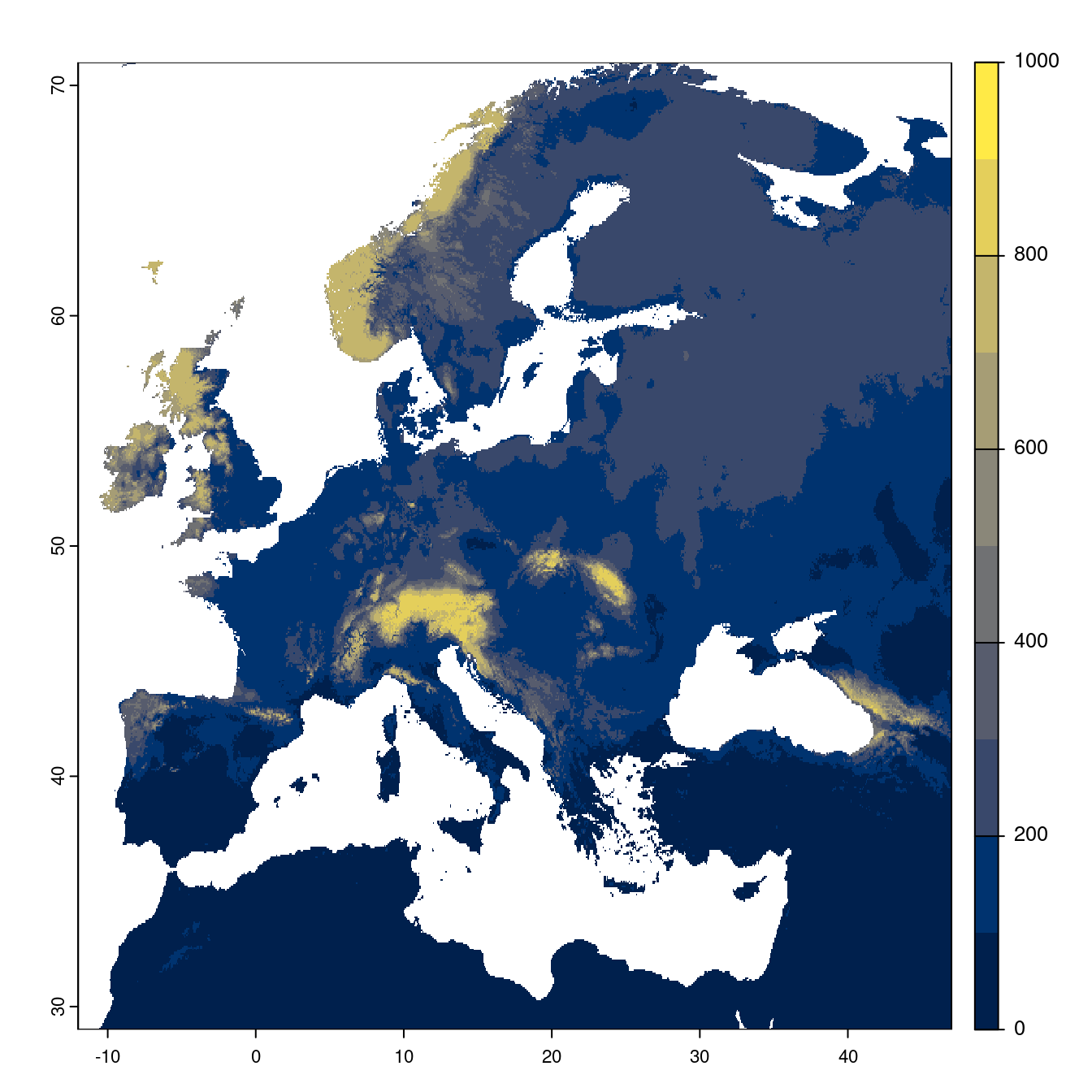
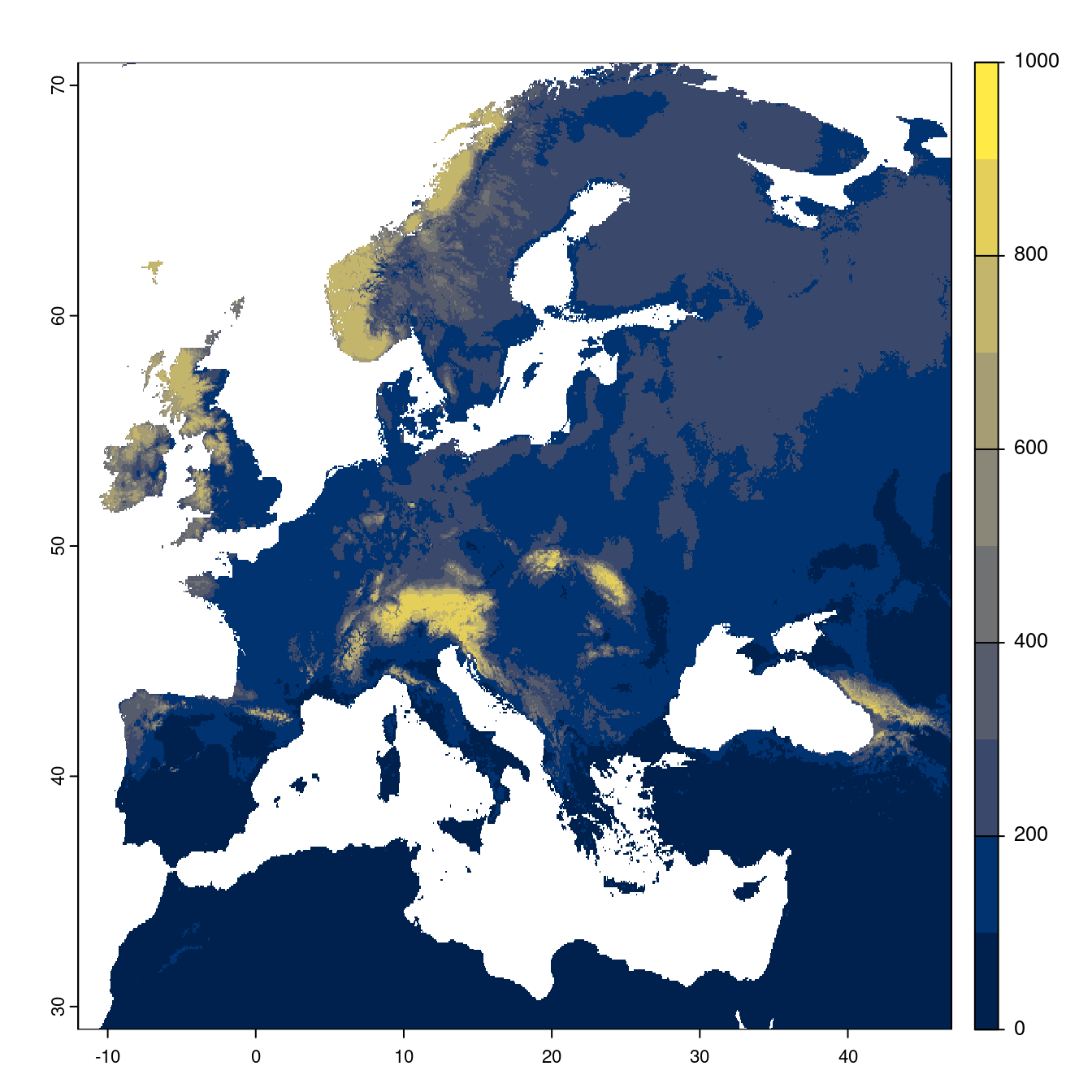
Figure 128.21: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP2-4.5 (modèle d’ensemble).
- SSP3-7.0
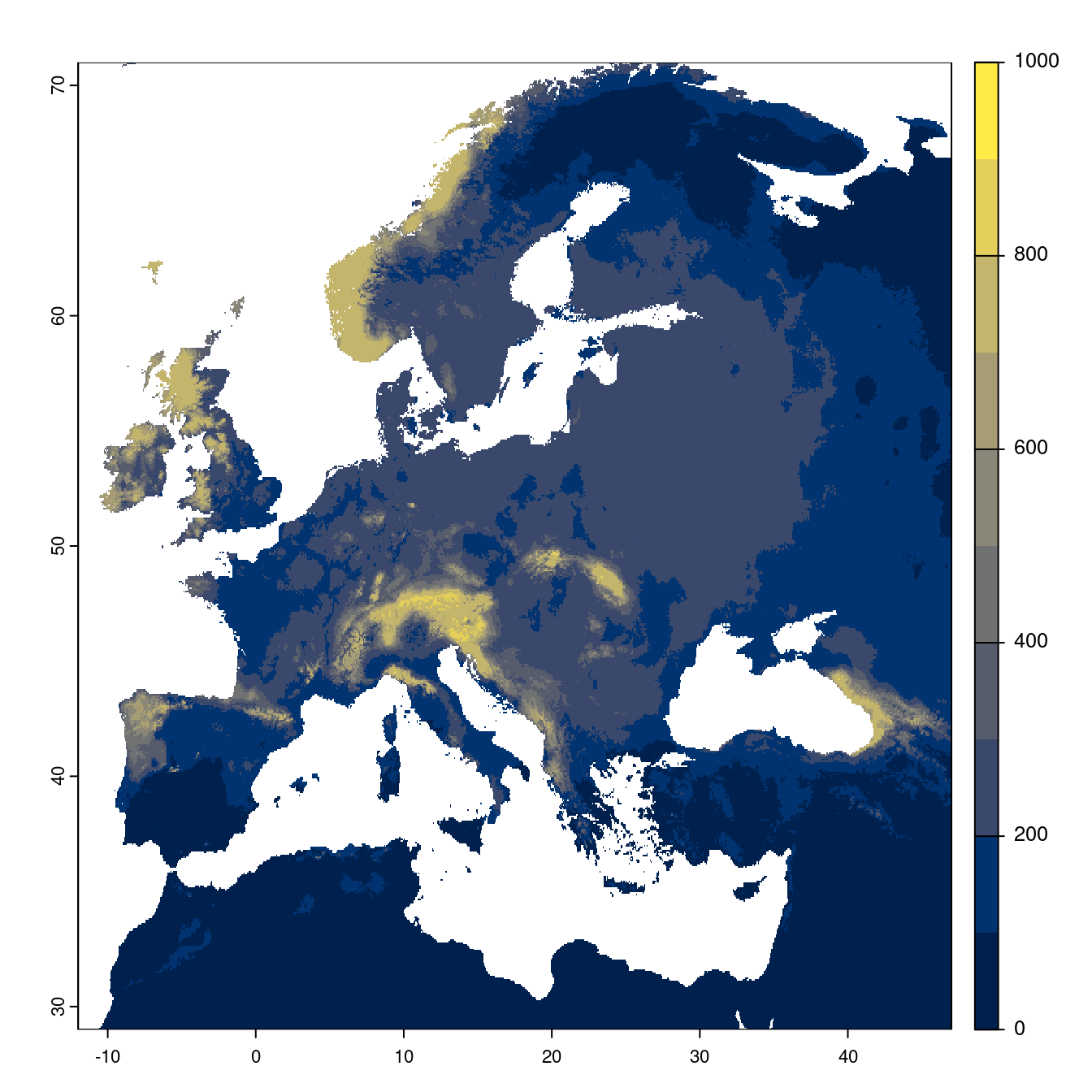
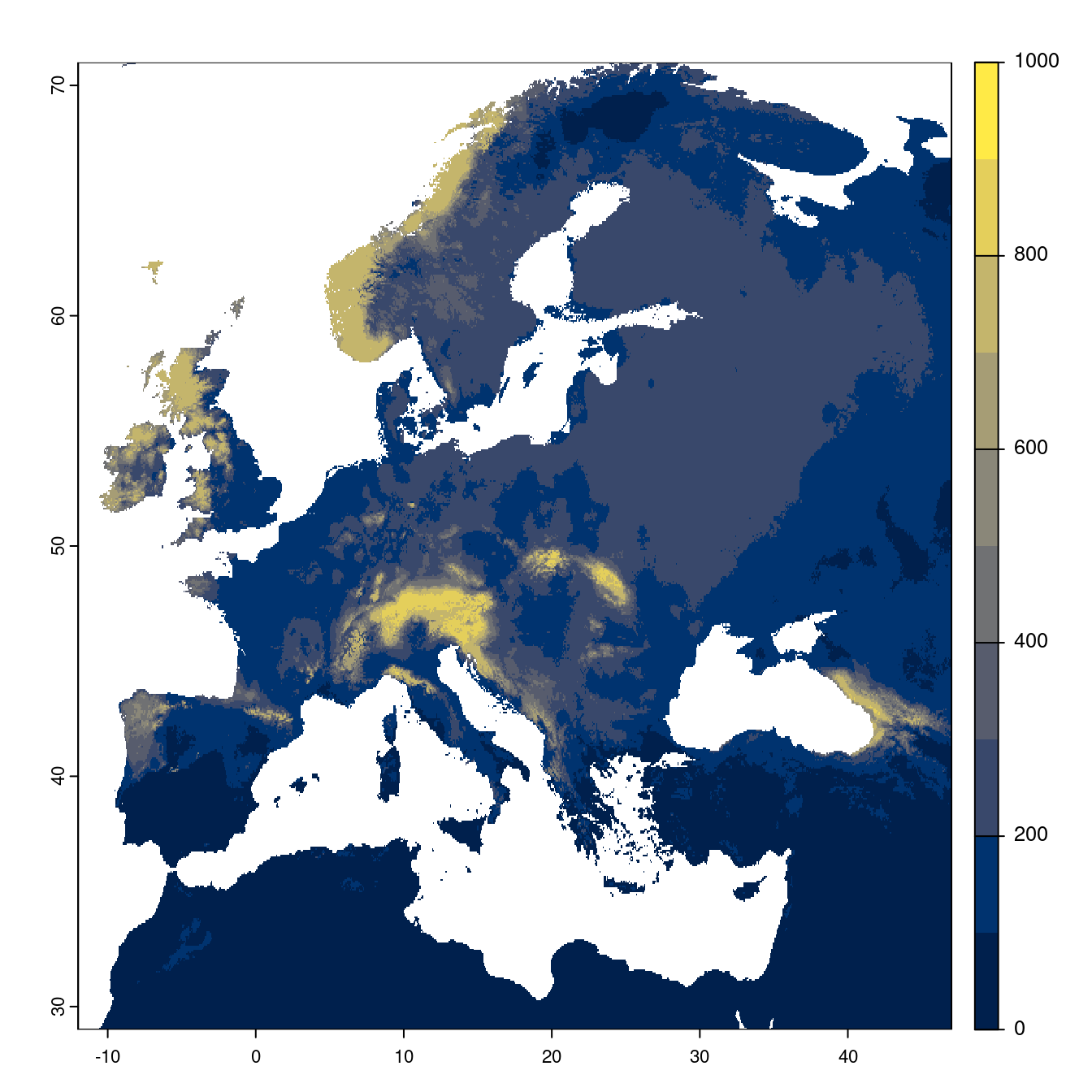
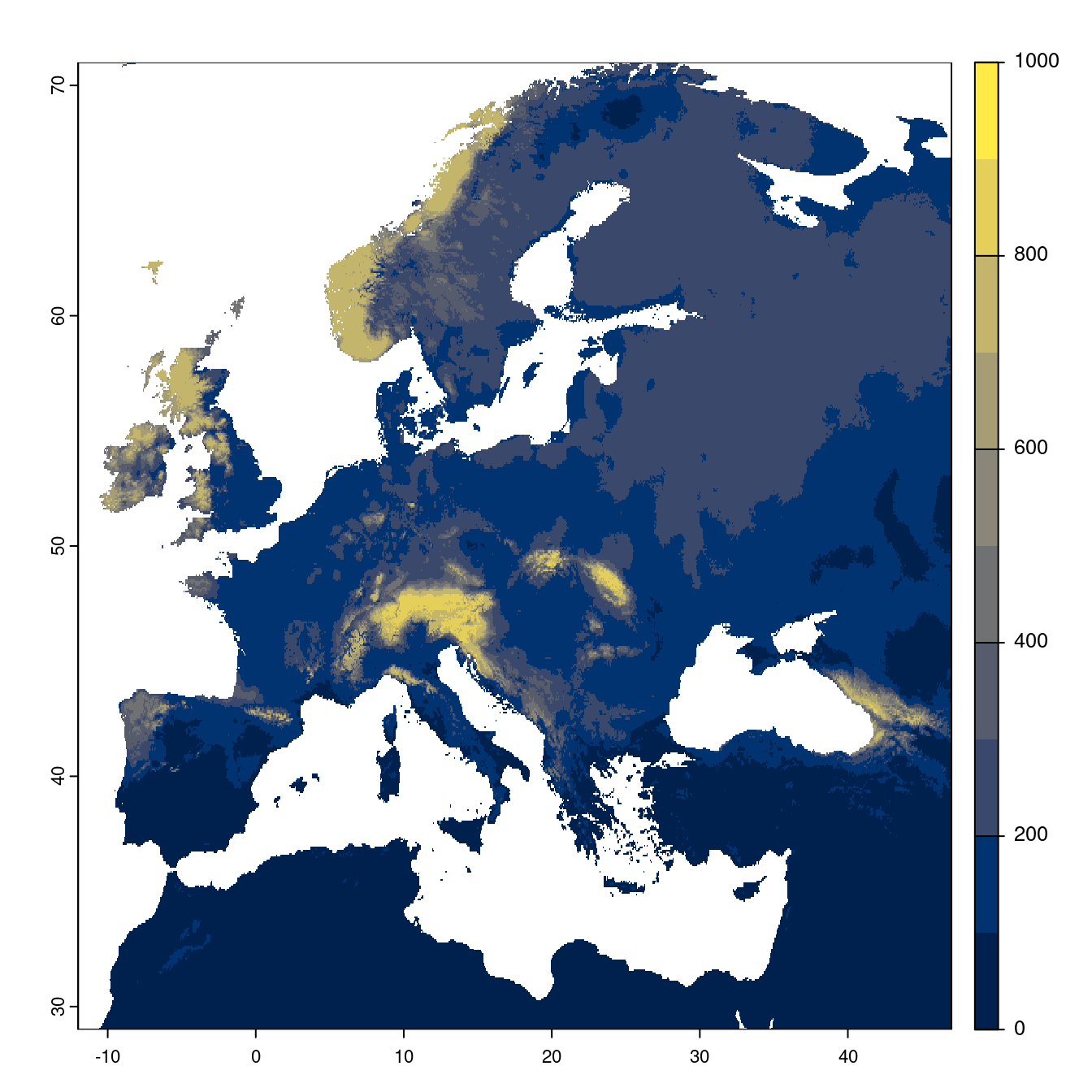
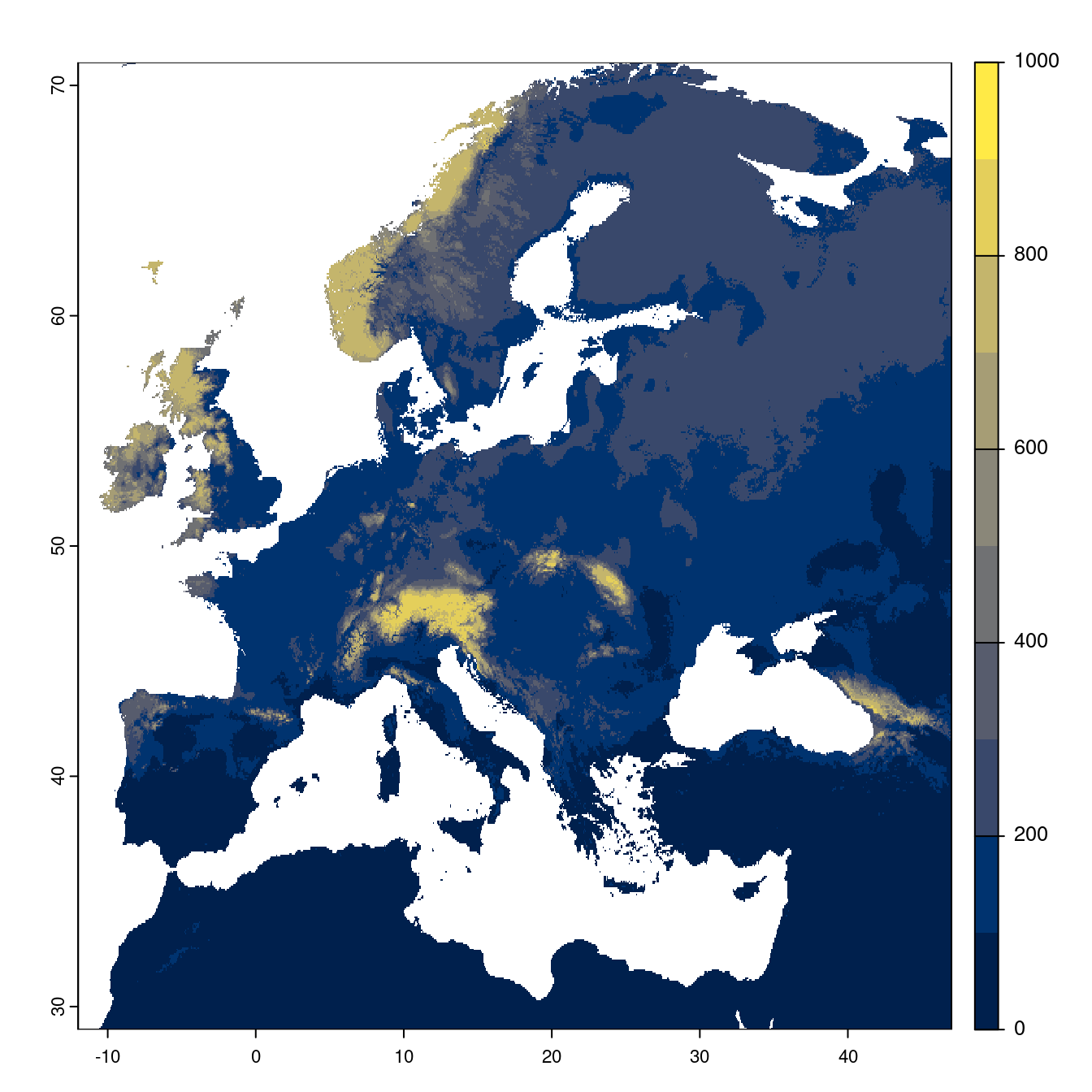
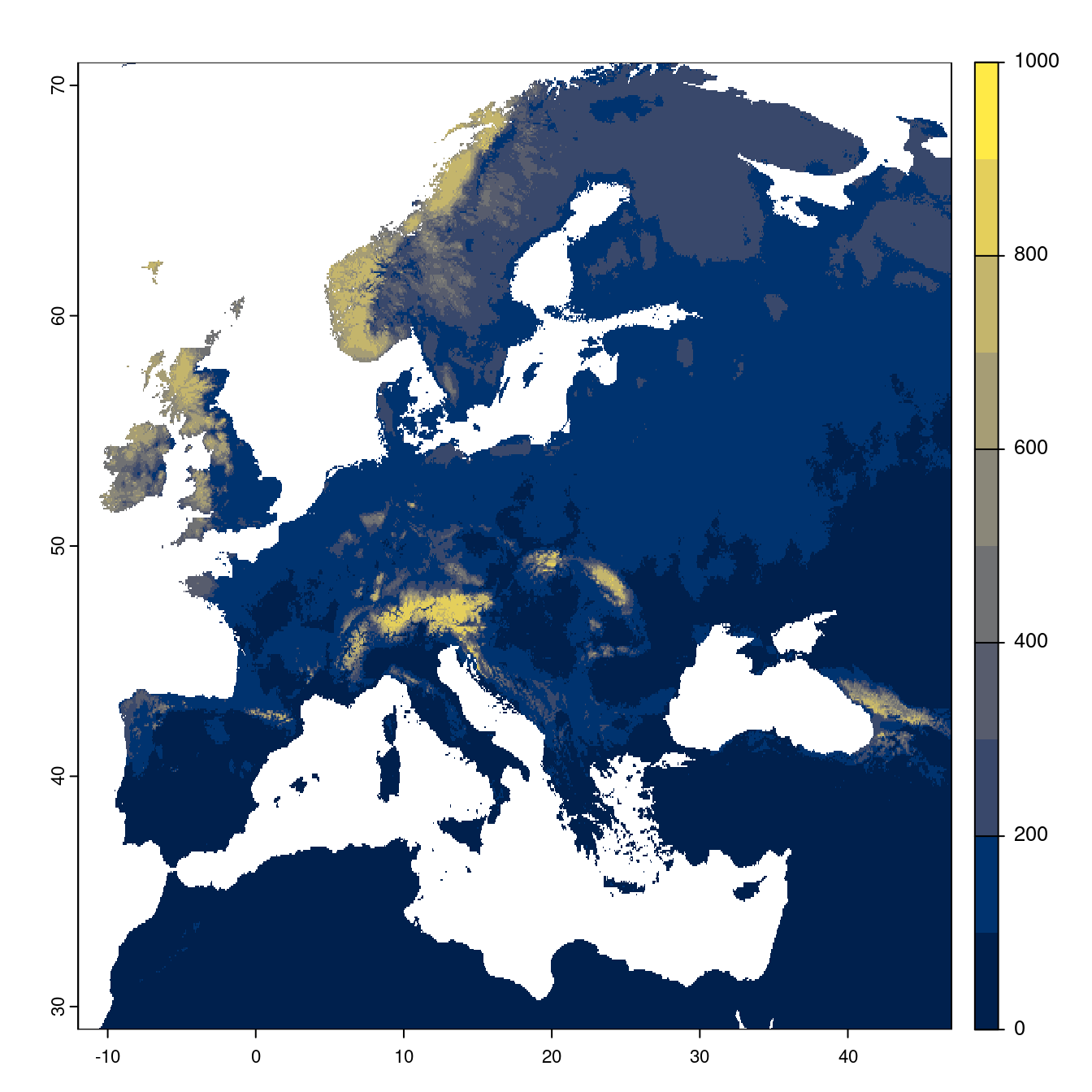
Figure 128.22: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP3-7.0 (modèle d’ensemble).
- SSP5-8.5
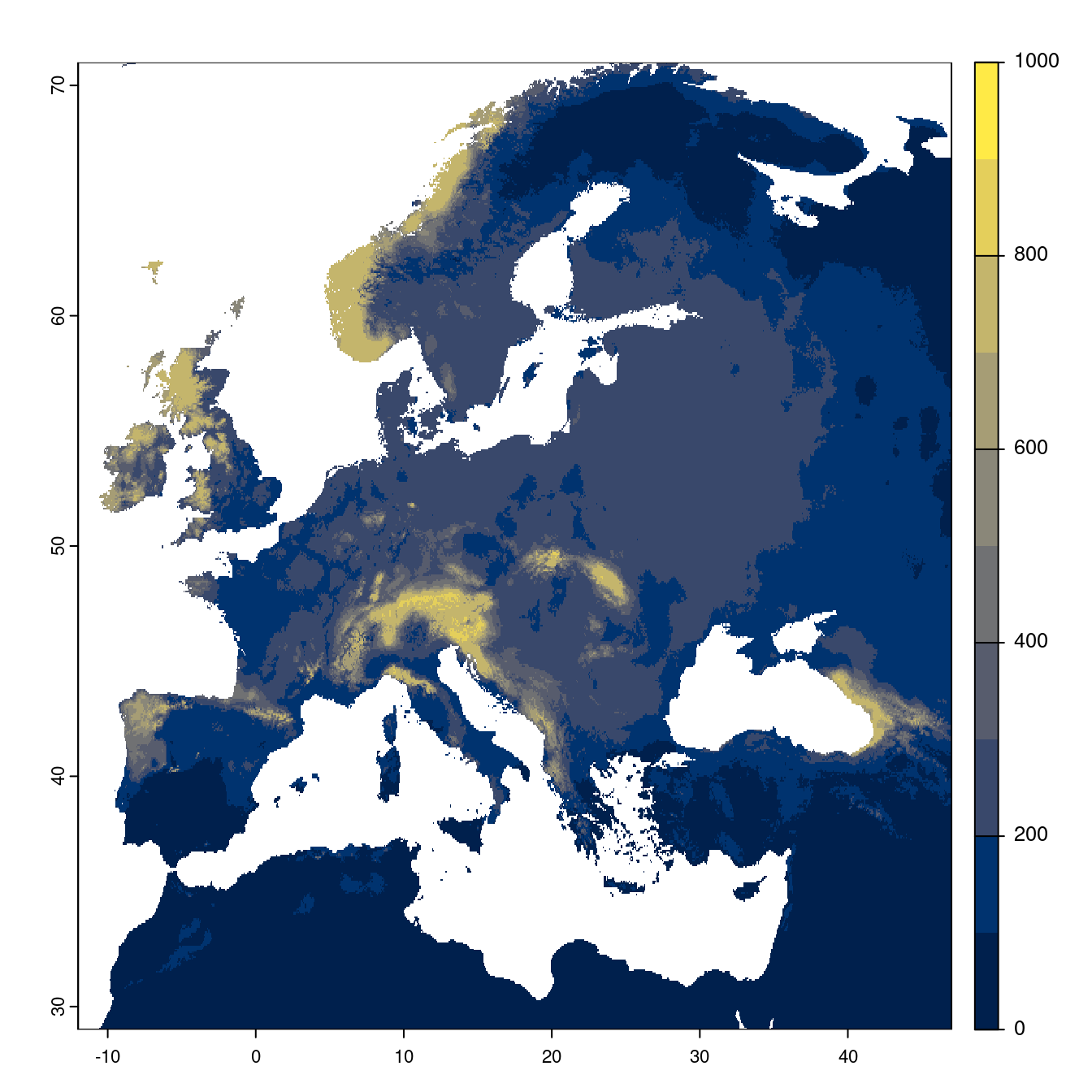
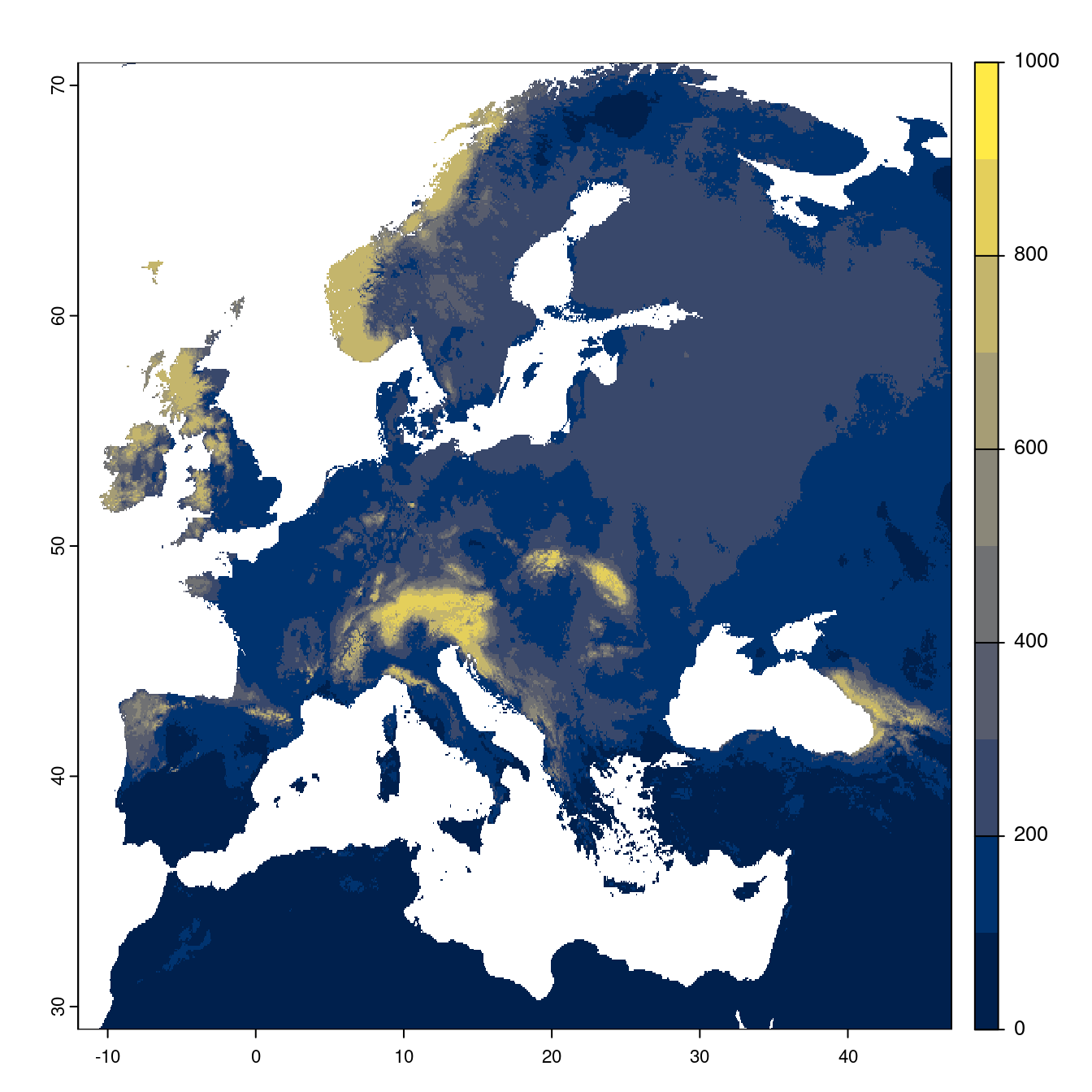
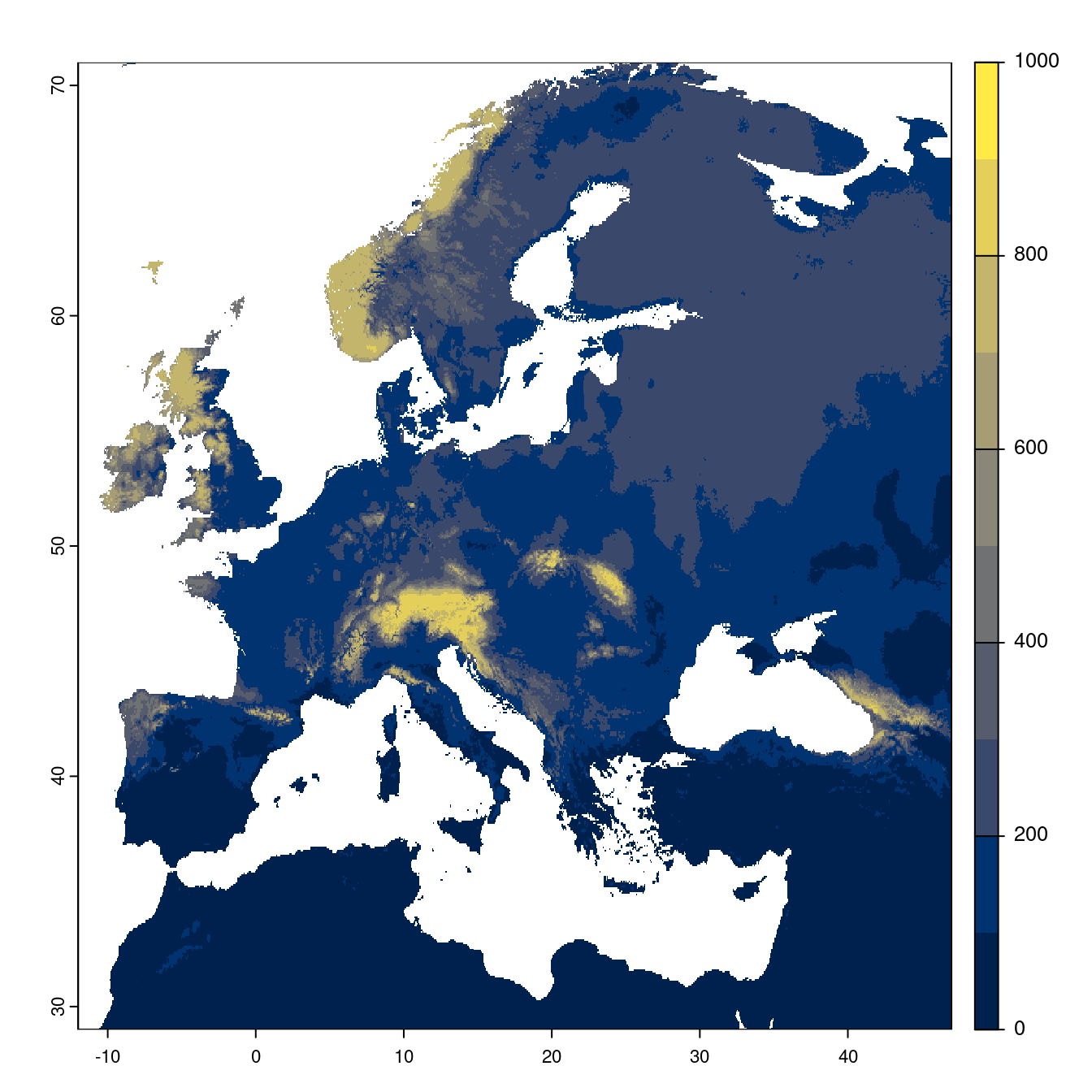
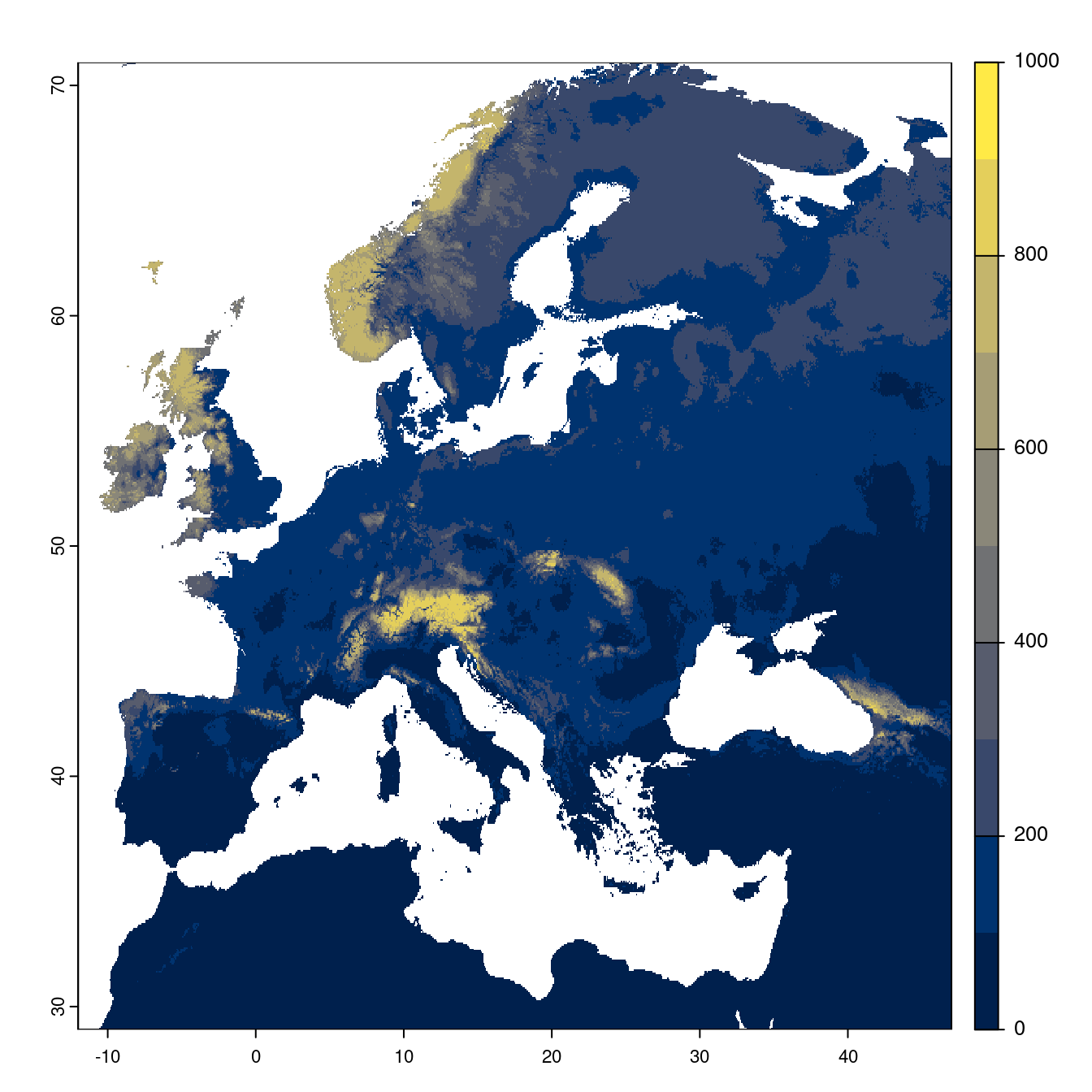
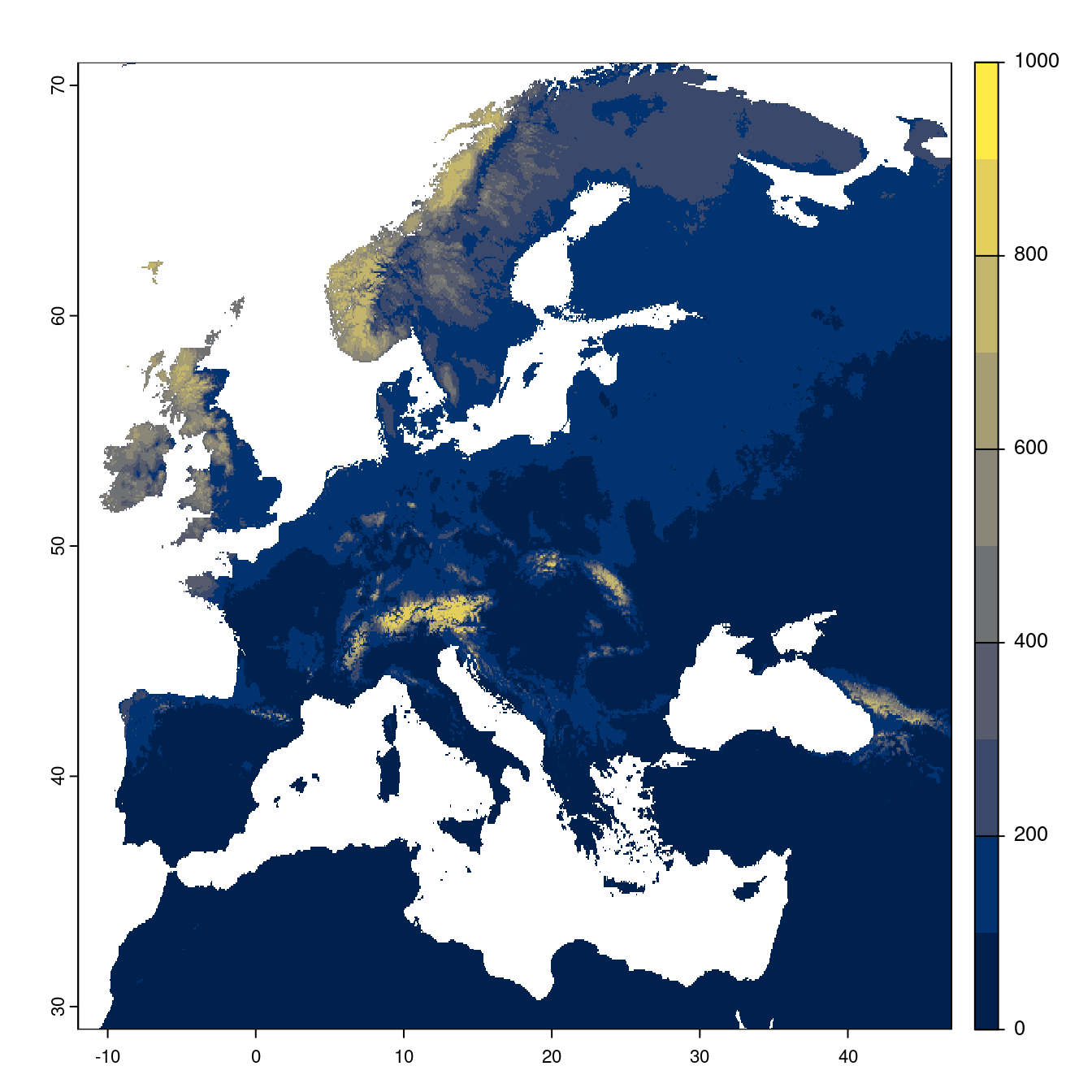
Figure 128.23: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble).
128.4 Inférence à l’échelle de la métropole
- Croisement avec températures de surface 2019 (fournie par la métropole)
- → conditions climatiques favorables à fine échelle
- à peu près 7 pixels × 9 (WorldClim)
128.4.1 Distributions potentielles futures
Même chose que précédemment mais sur les rasters de Grenoble
- SSP1-2.6
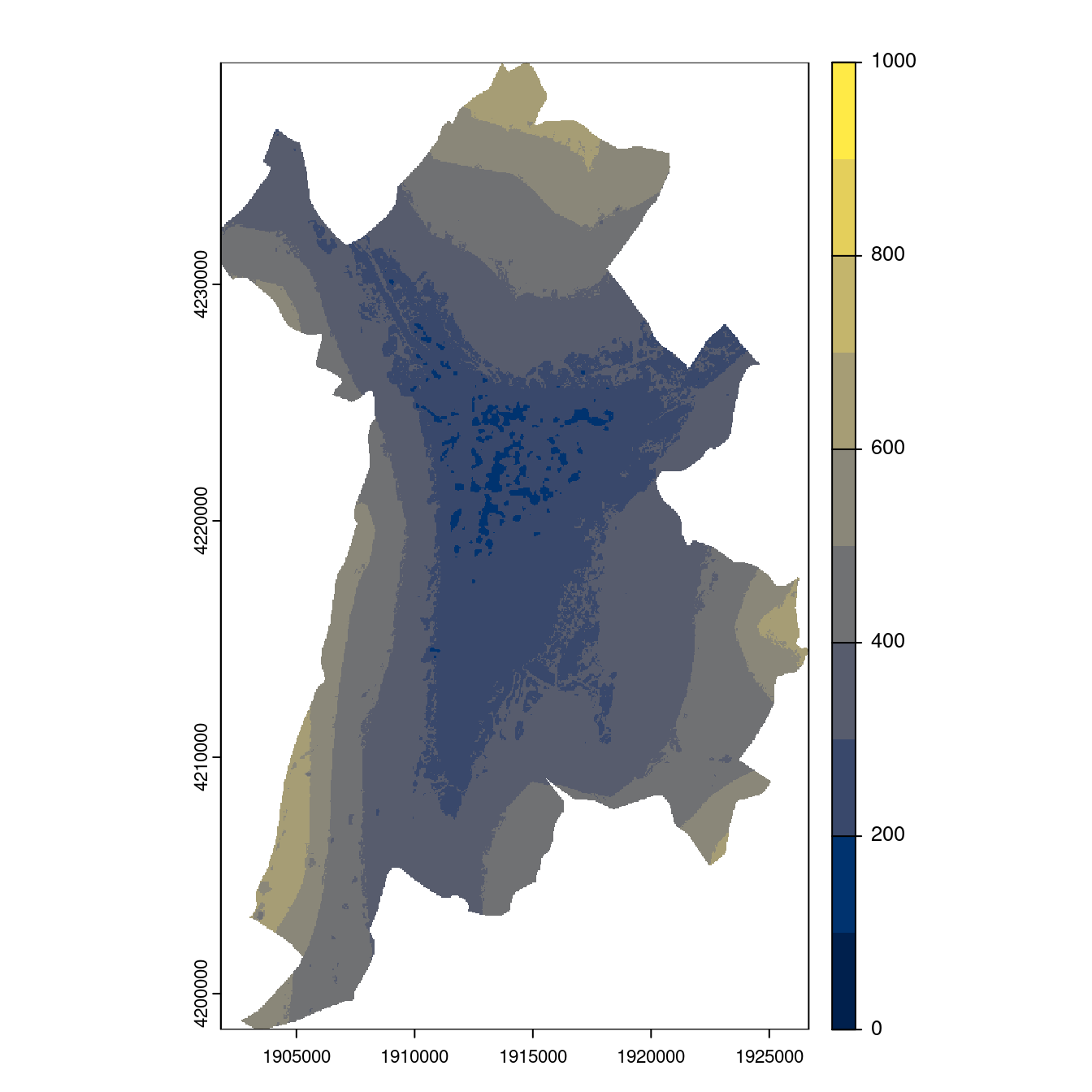
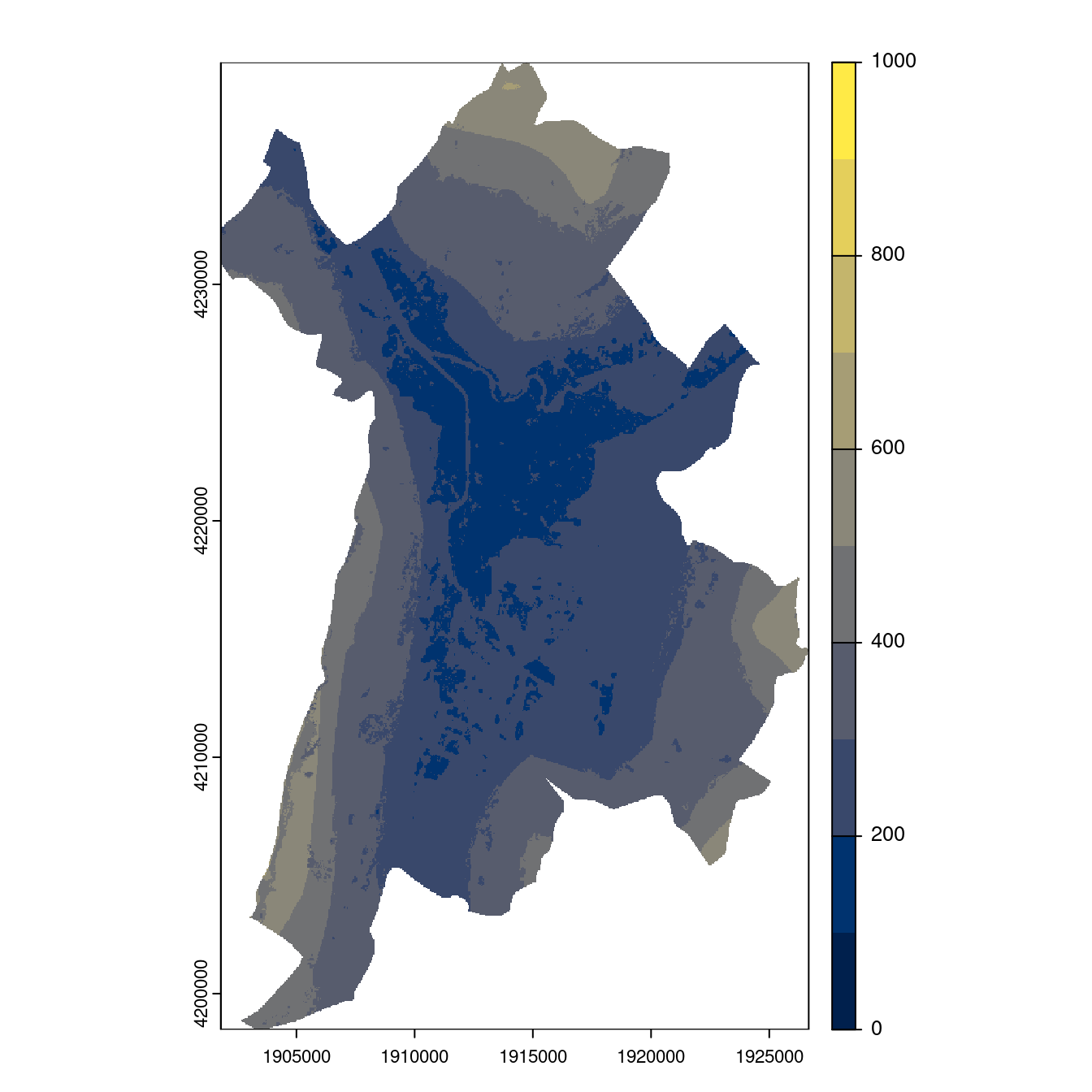
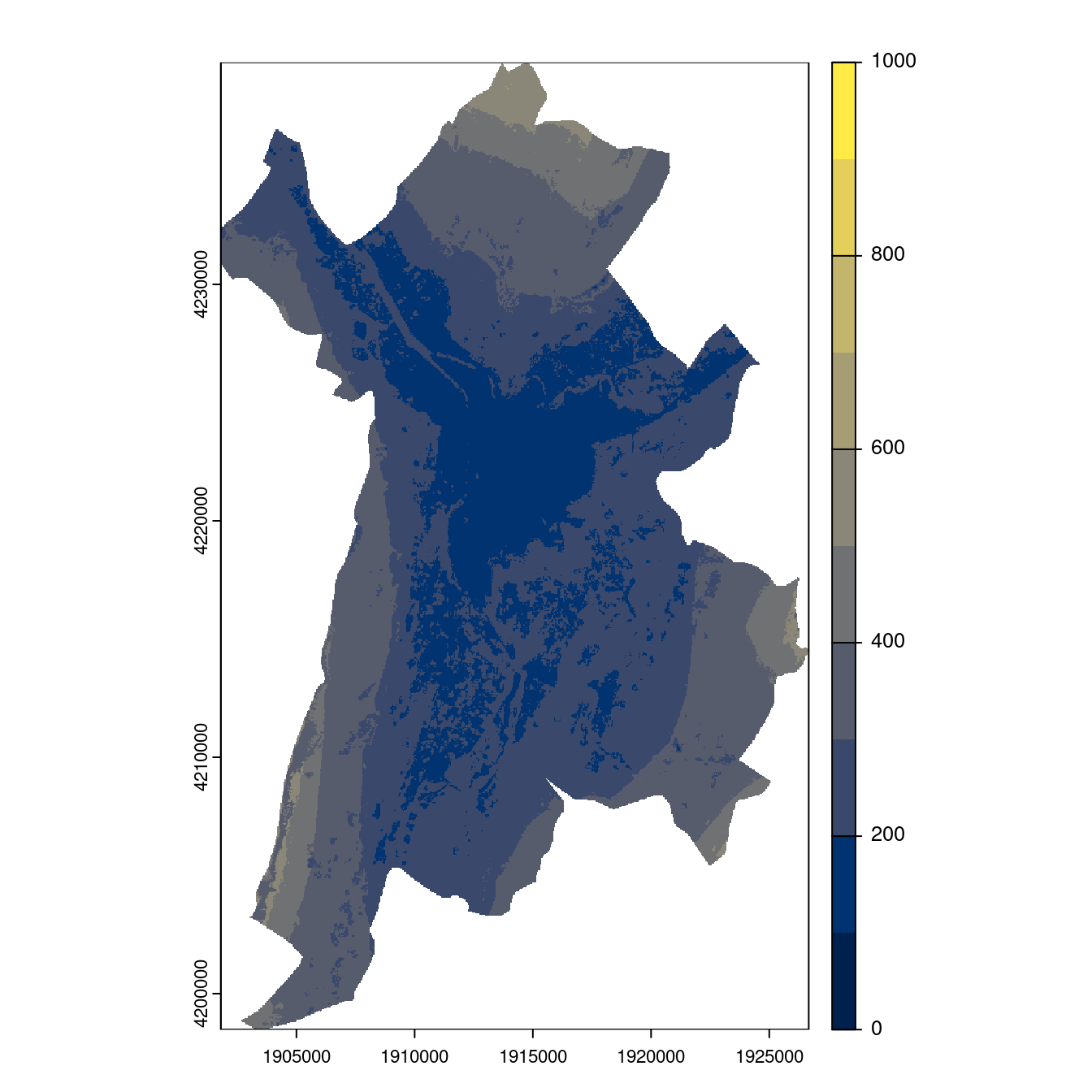
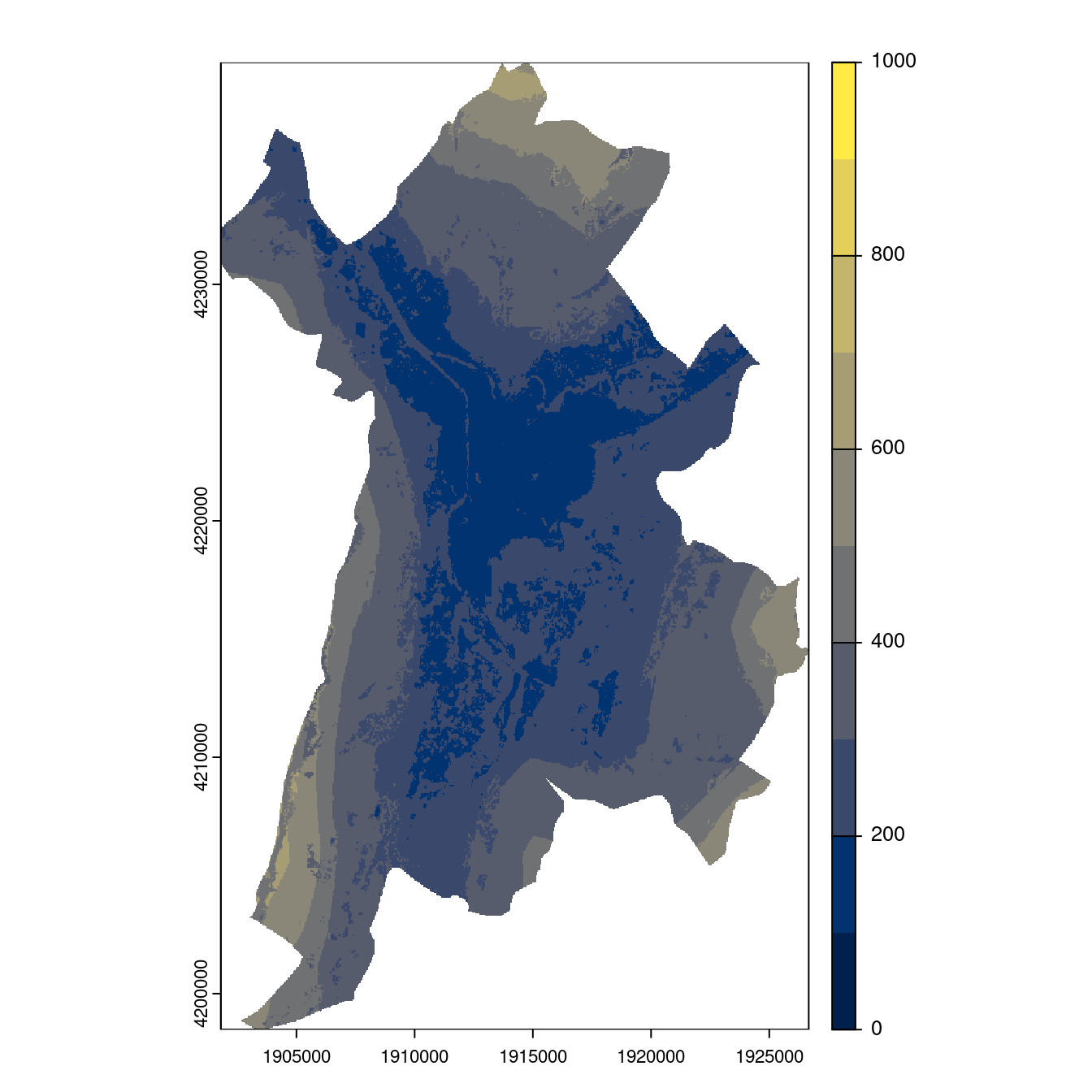
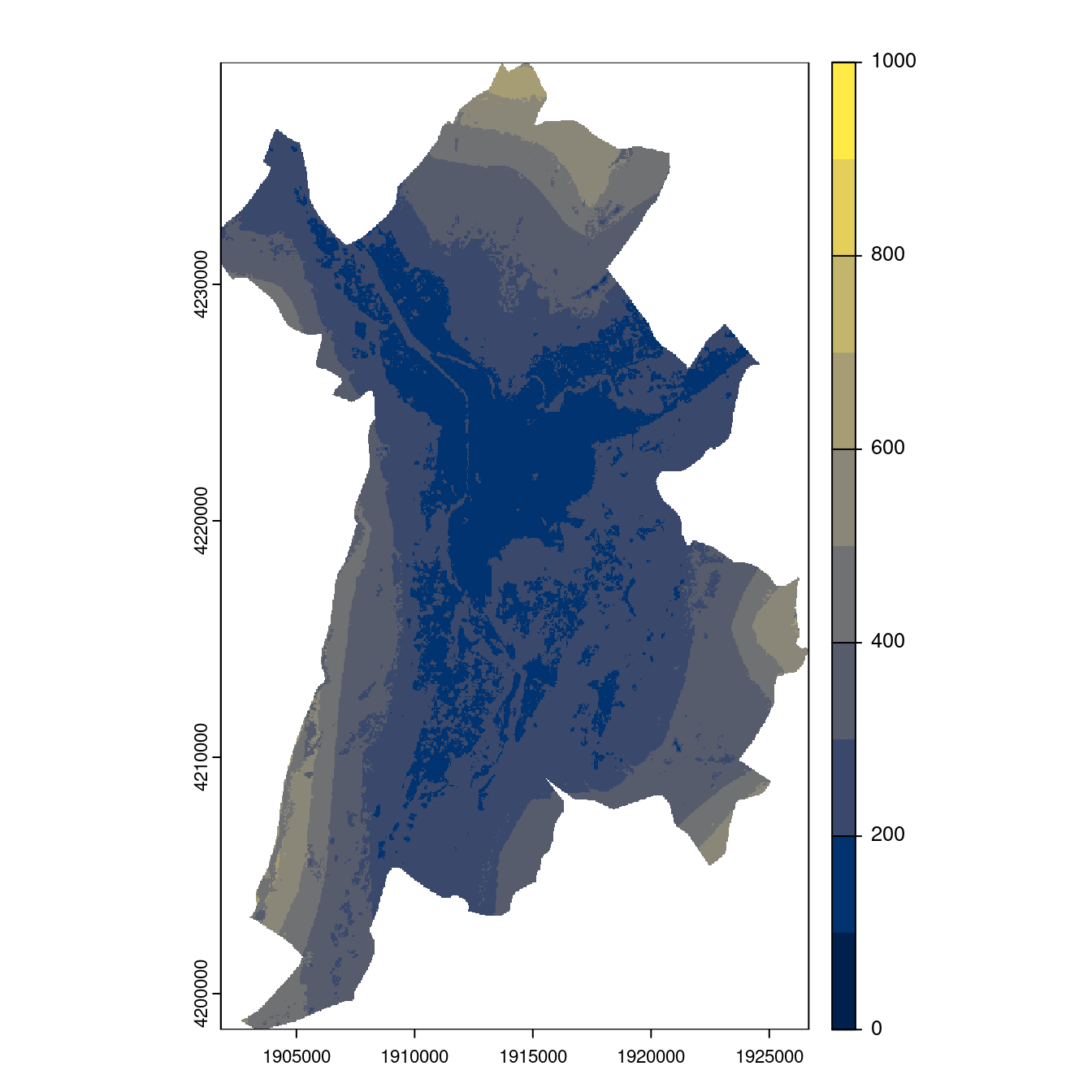
Figure 128.24: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP2-4.5
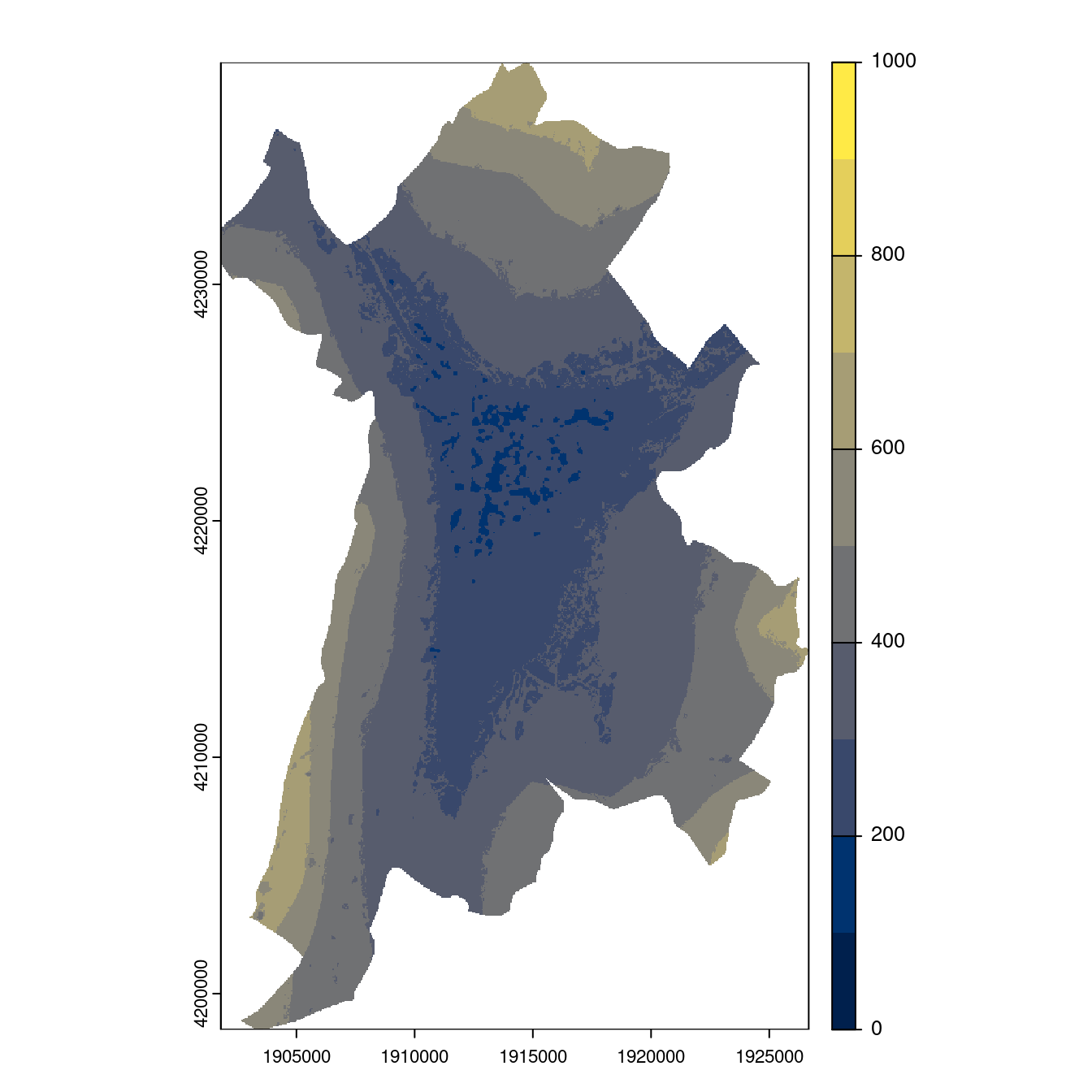
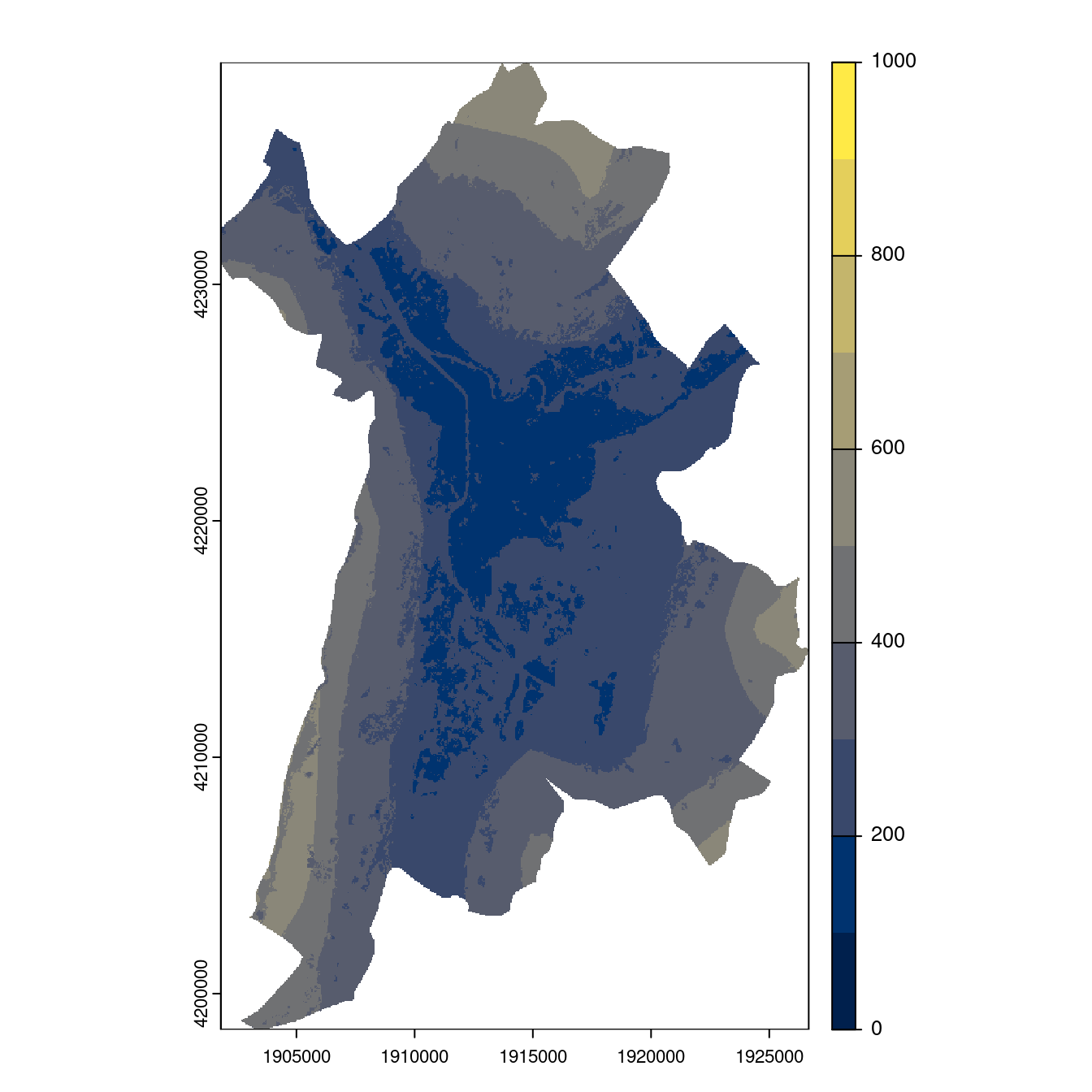
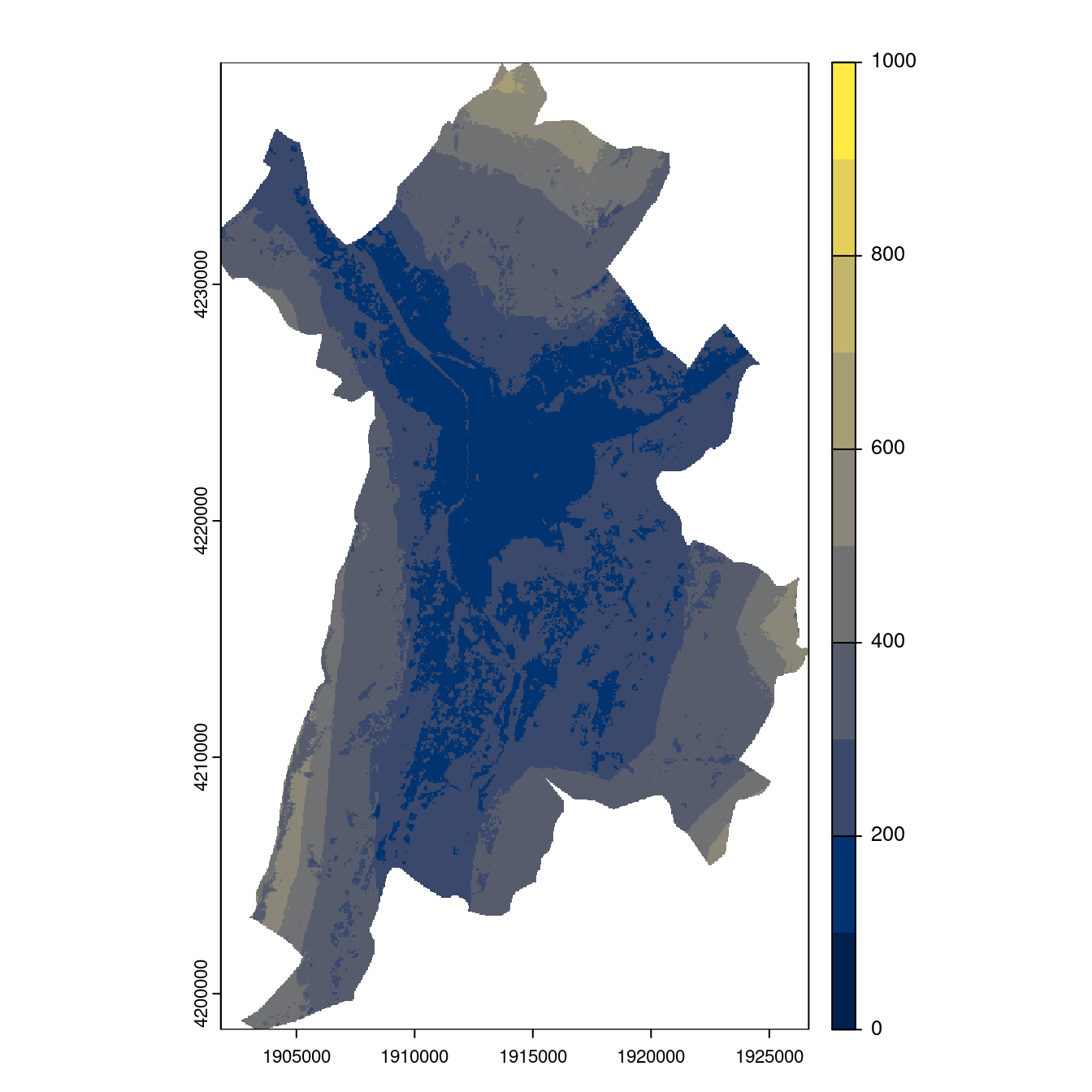
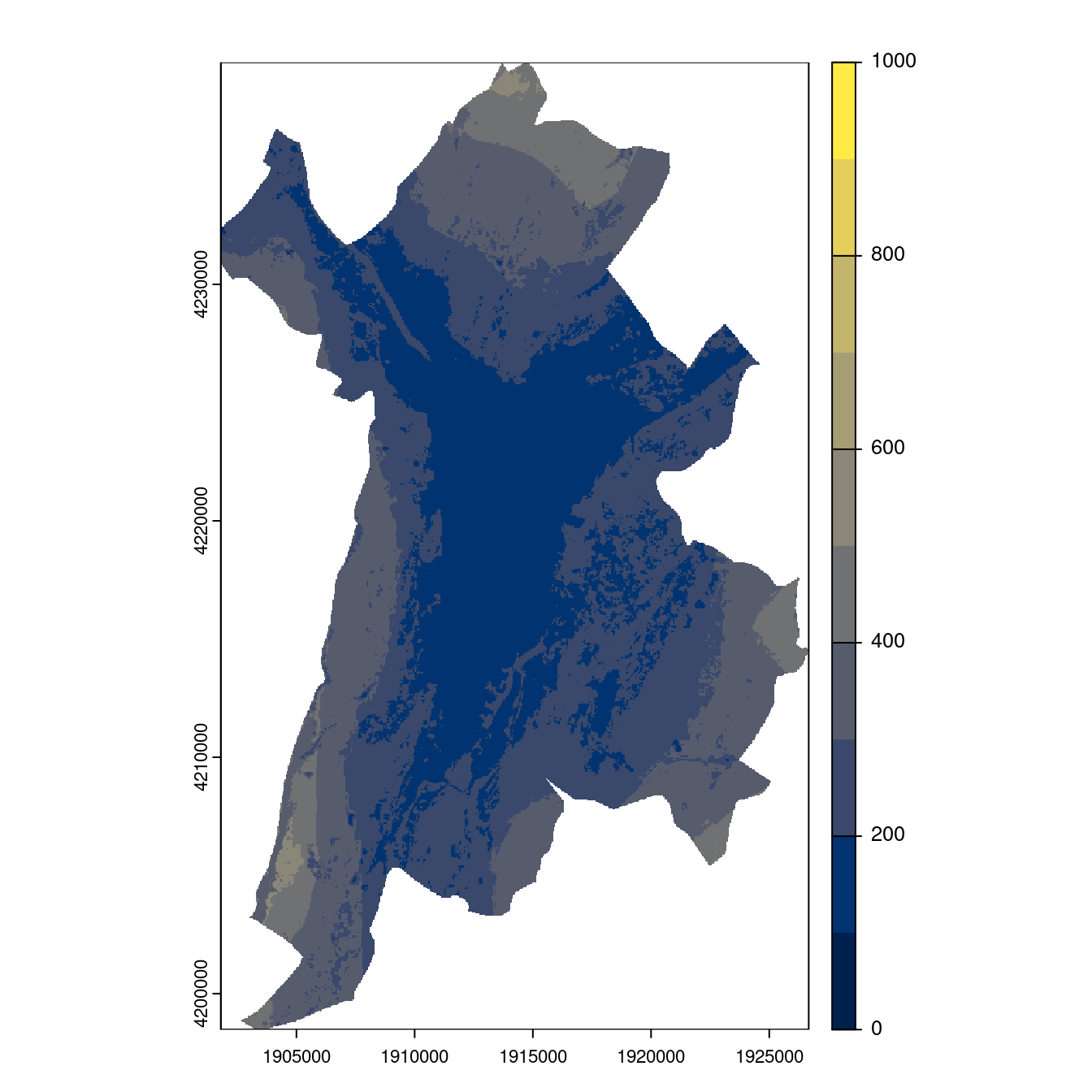
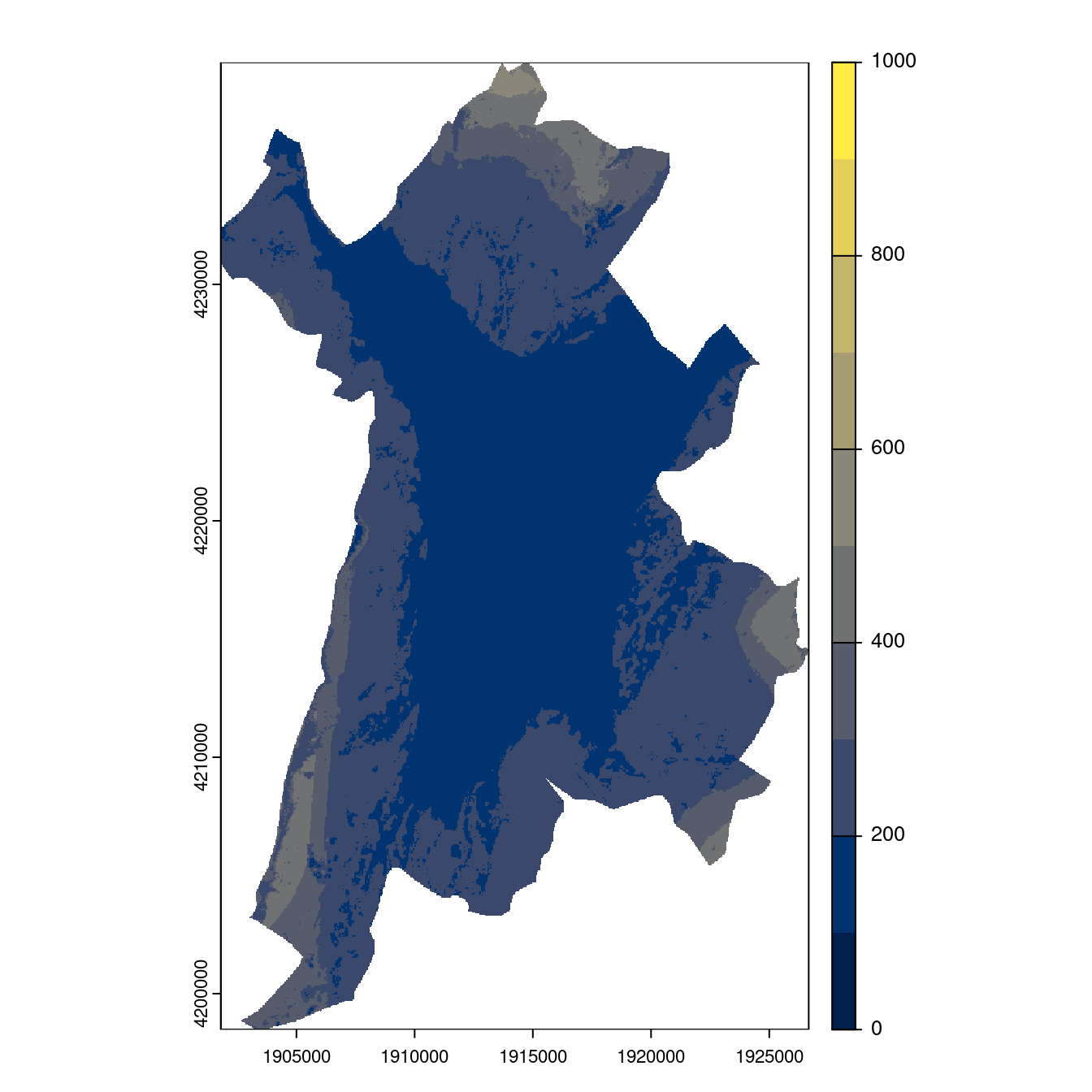
Figure 128.25: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP3-7.0
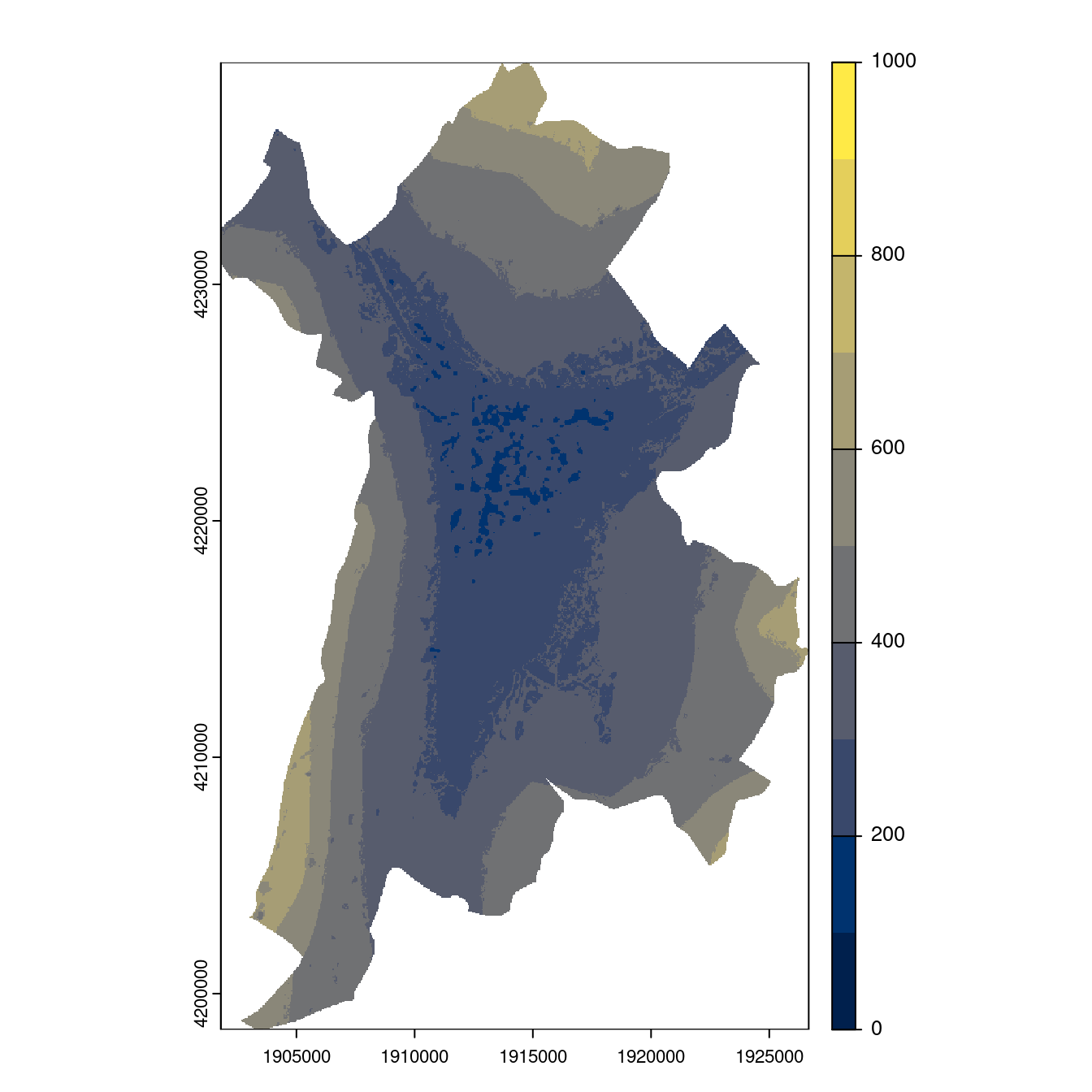
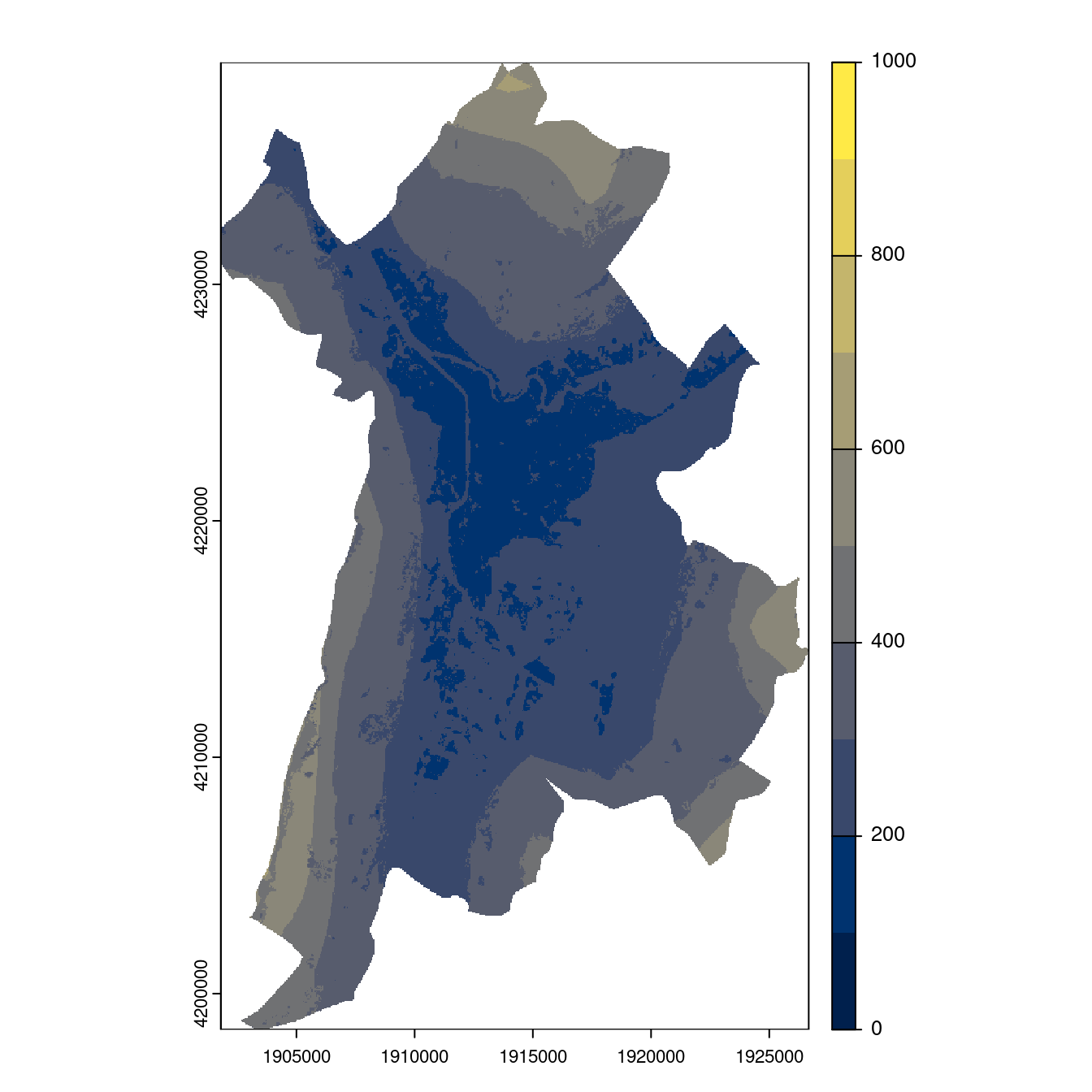
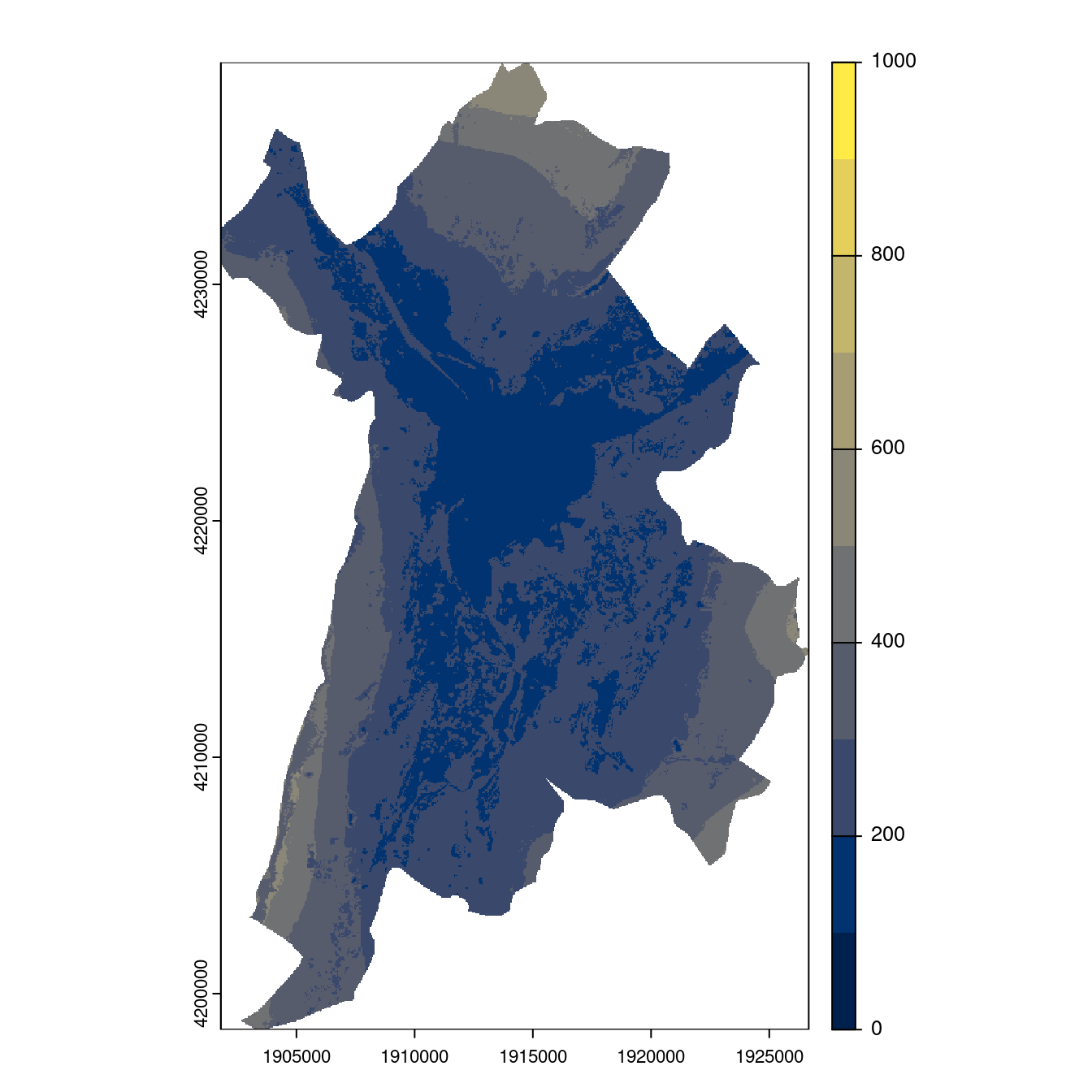
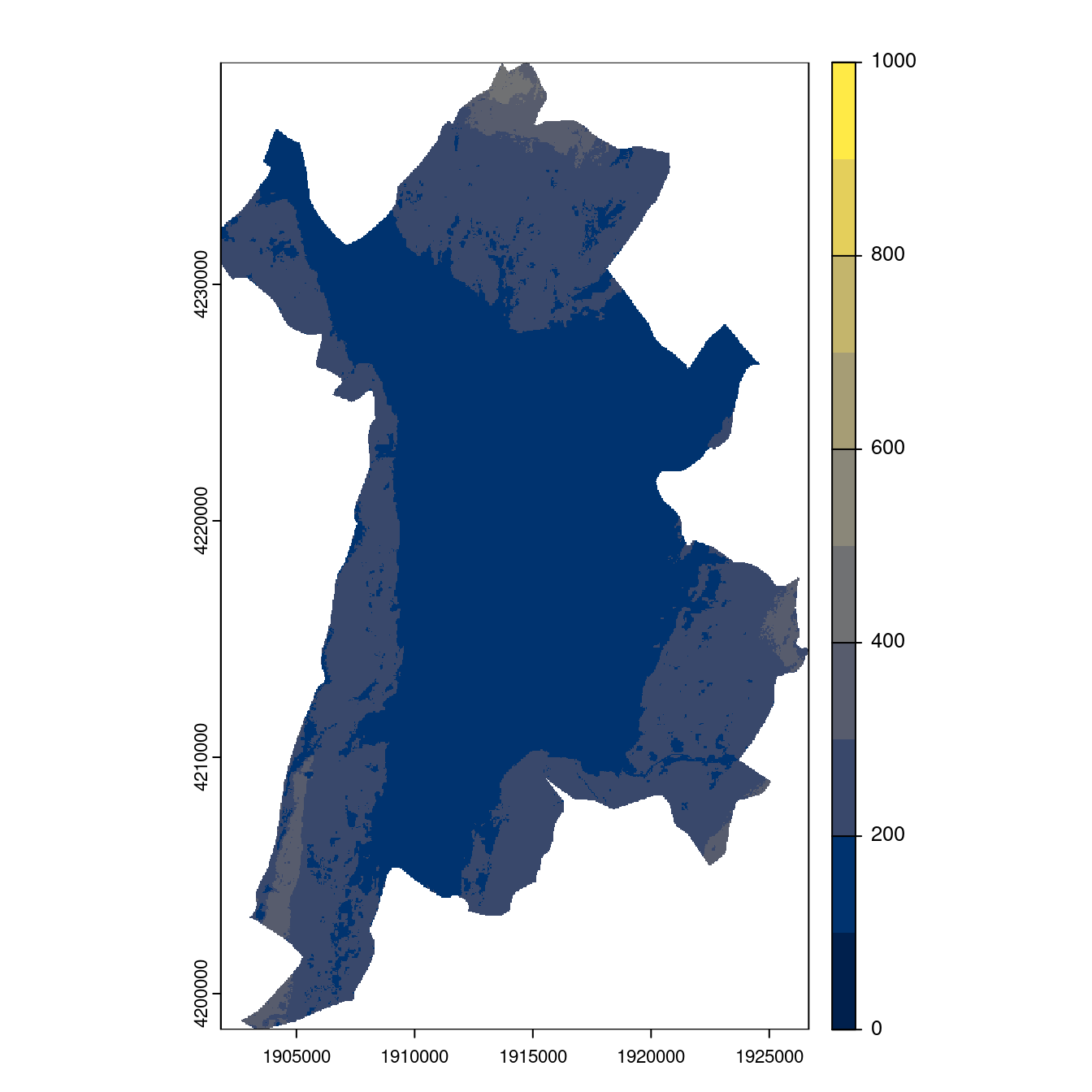
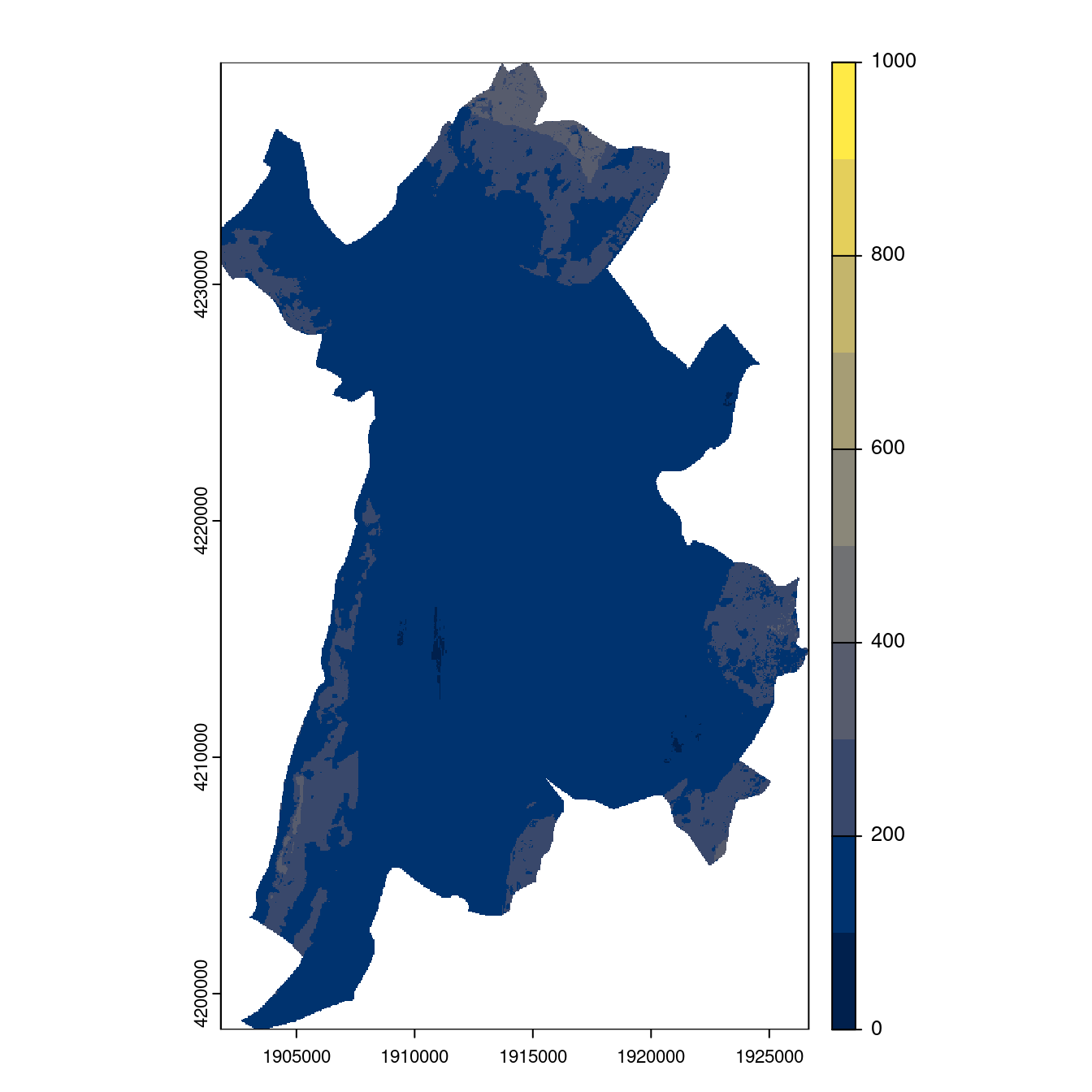
Figure 128.26: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP5-8.5
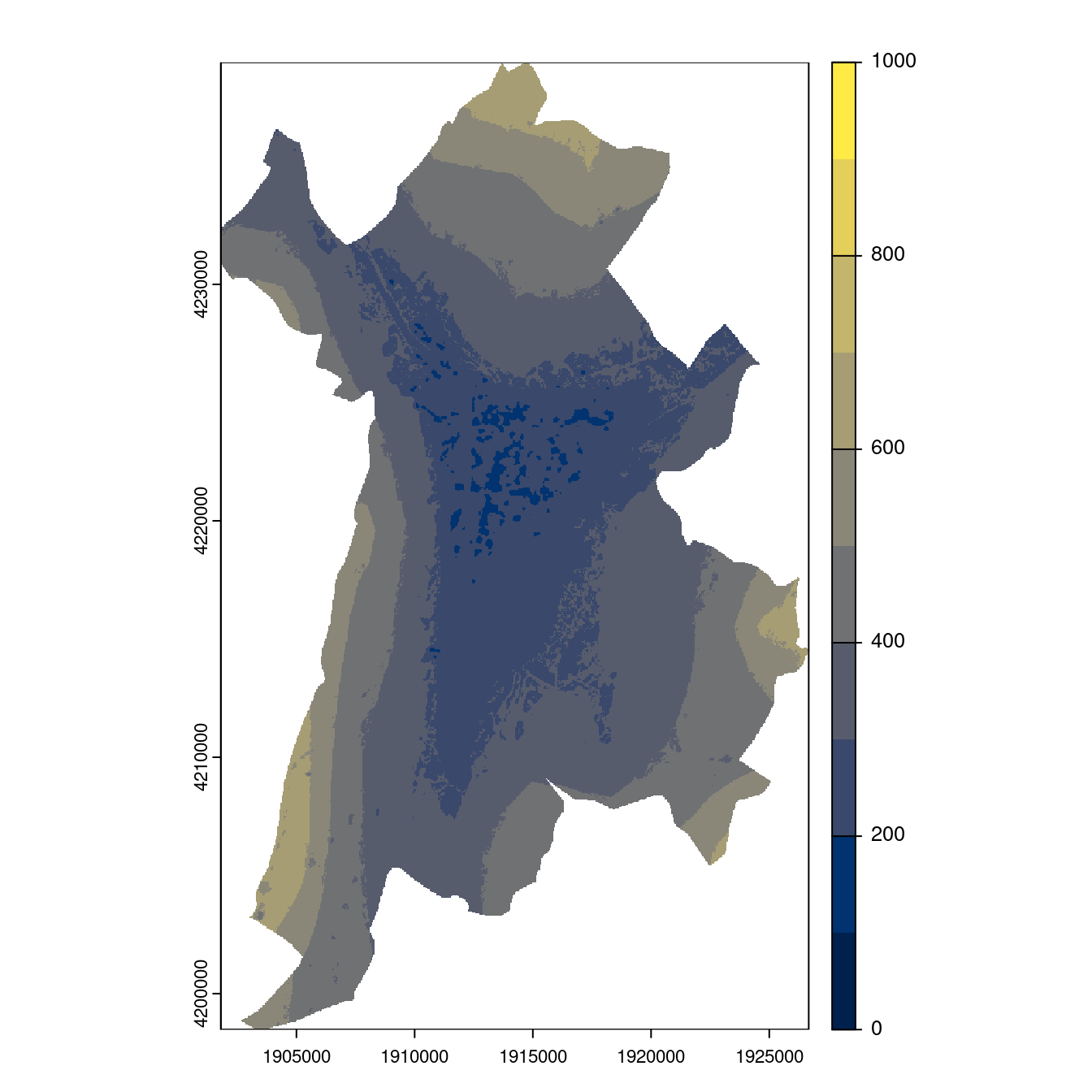
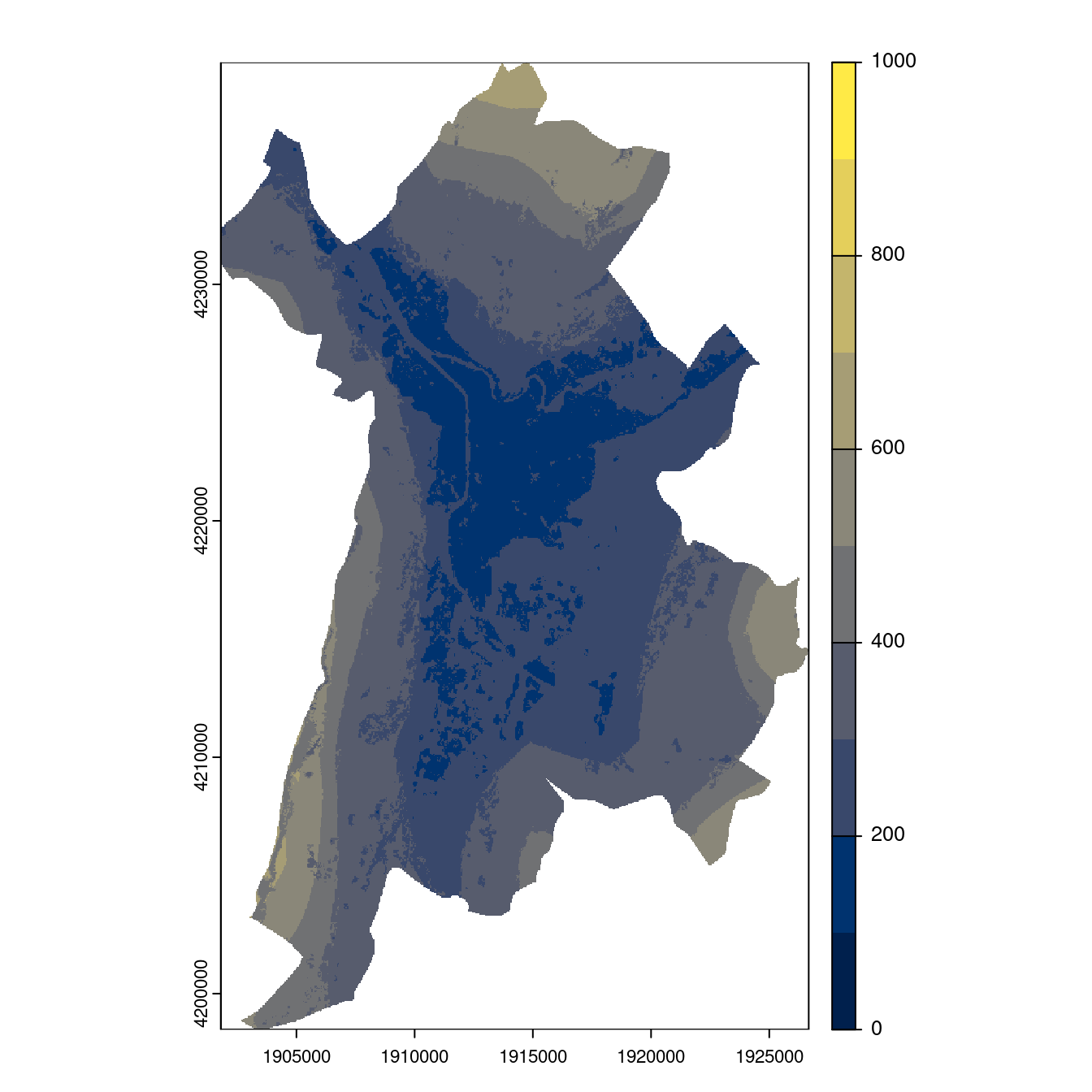
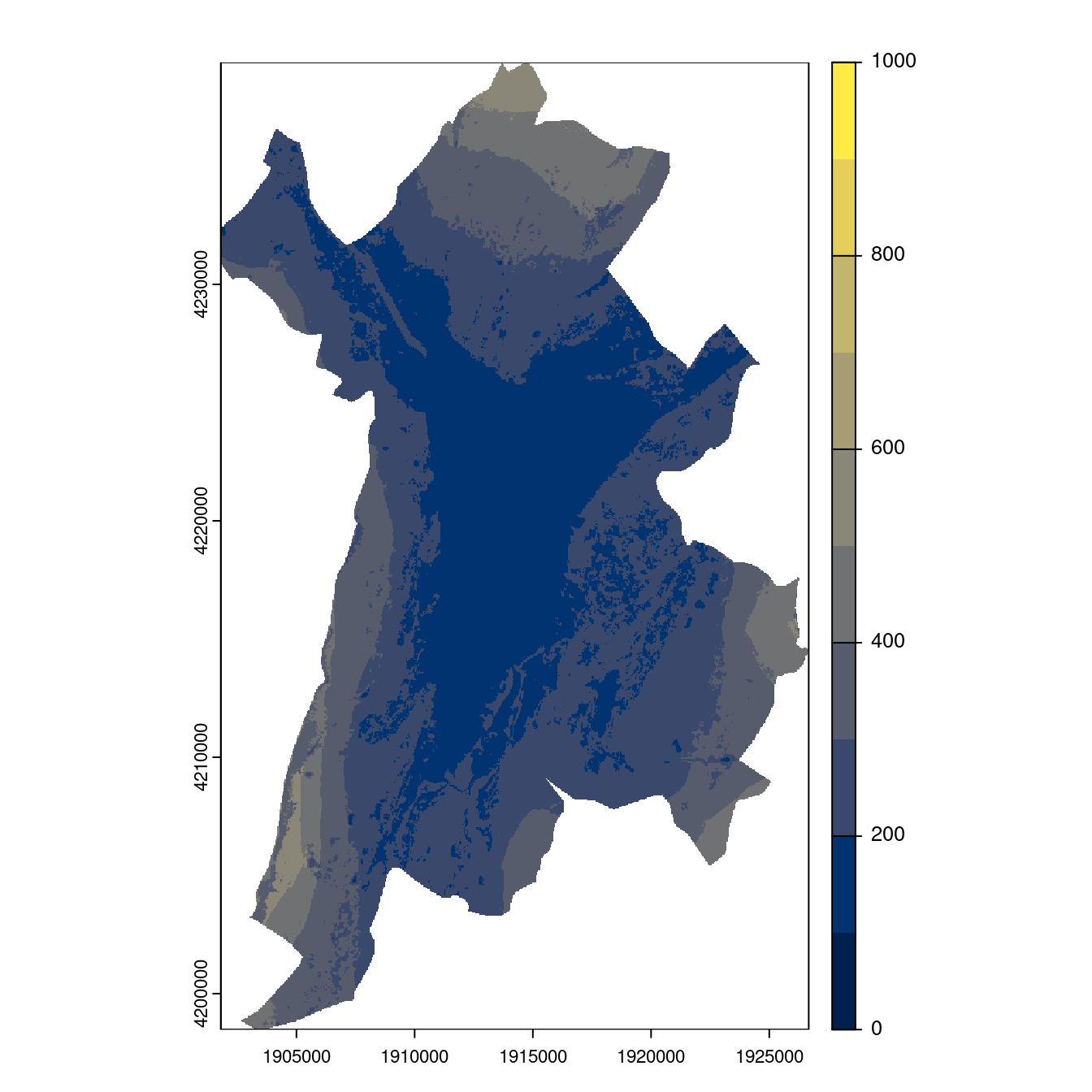
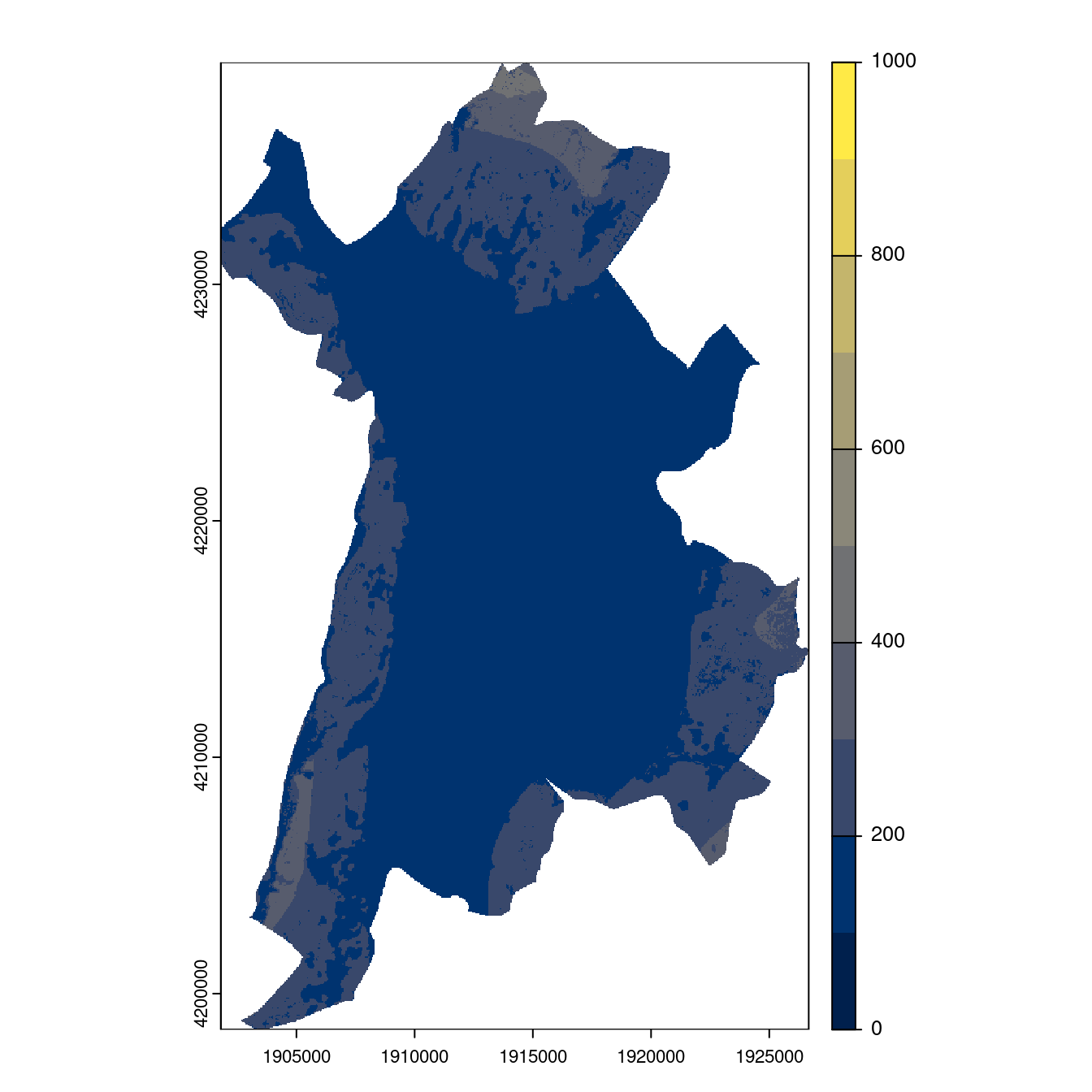
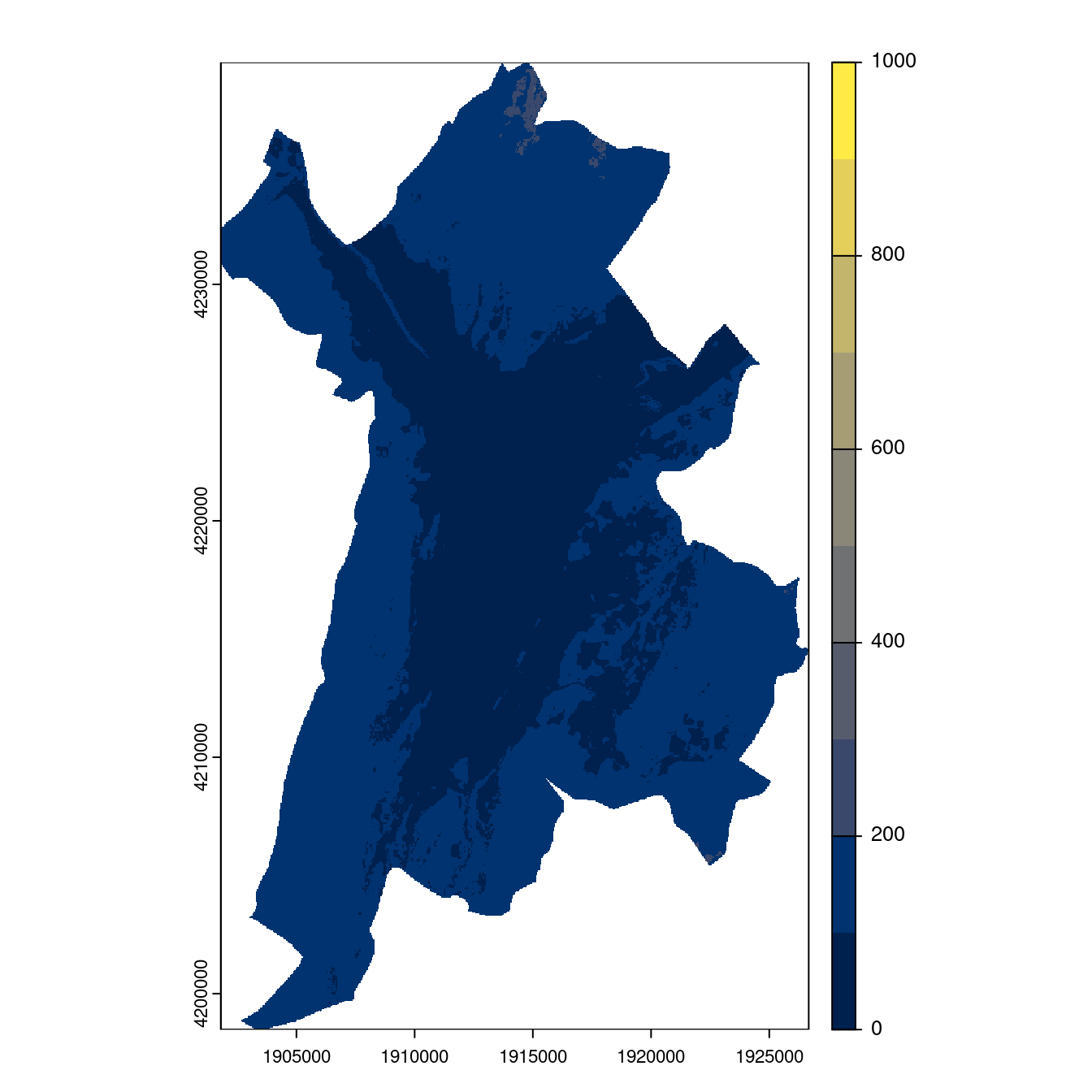
Figure 128.27: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
128.4.2 Évolution temporelle
# A tibble: 17 × 8
min q1 median mean q3 max ssp year
<table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <tab> <fct> <dbl>
1 172 298 373 380.2882 437 689 0 2000
2 148 238 294 309.9285 374 644 126 2040
3 138 205 244 269.3085 331 603 126 2060
4 142 221 293 301.5514 365 650 126 2080
5 140 211 271 285.3777 339 633 126 2100
6 143 230 299 307.1973 376 625 245 2040
7 140 211 269 285.0214 349 621 245 2060
8 126 179 232 253.1473 313 539 245 2080
9 117 153 206 219.3445 260 533 245 2100
10 149 239 295 311.2122 376 651 370 2040
11 132 197 238 260.4873 315 594 370 2060
12 113 141 189 193.4390 235 425 370 2080
13 94 107 129 145.9388 171 383 370 2100
14 144 233 308 314.7354 377 659 585 2040
15 121 181 236 254.2124 307 597 585 2060
16 103 117 159 172.0547 217 464 585 2080
17 88 96 103 109.7385 120 303 585 2100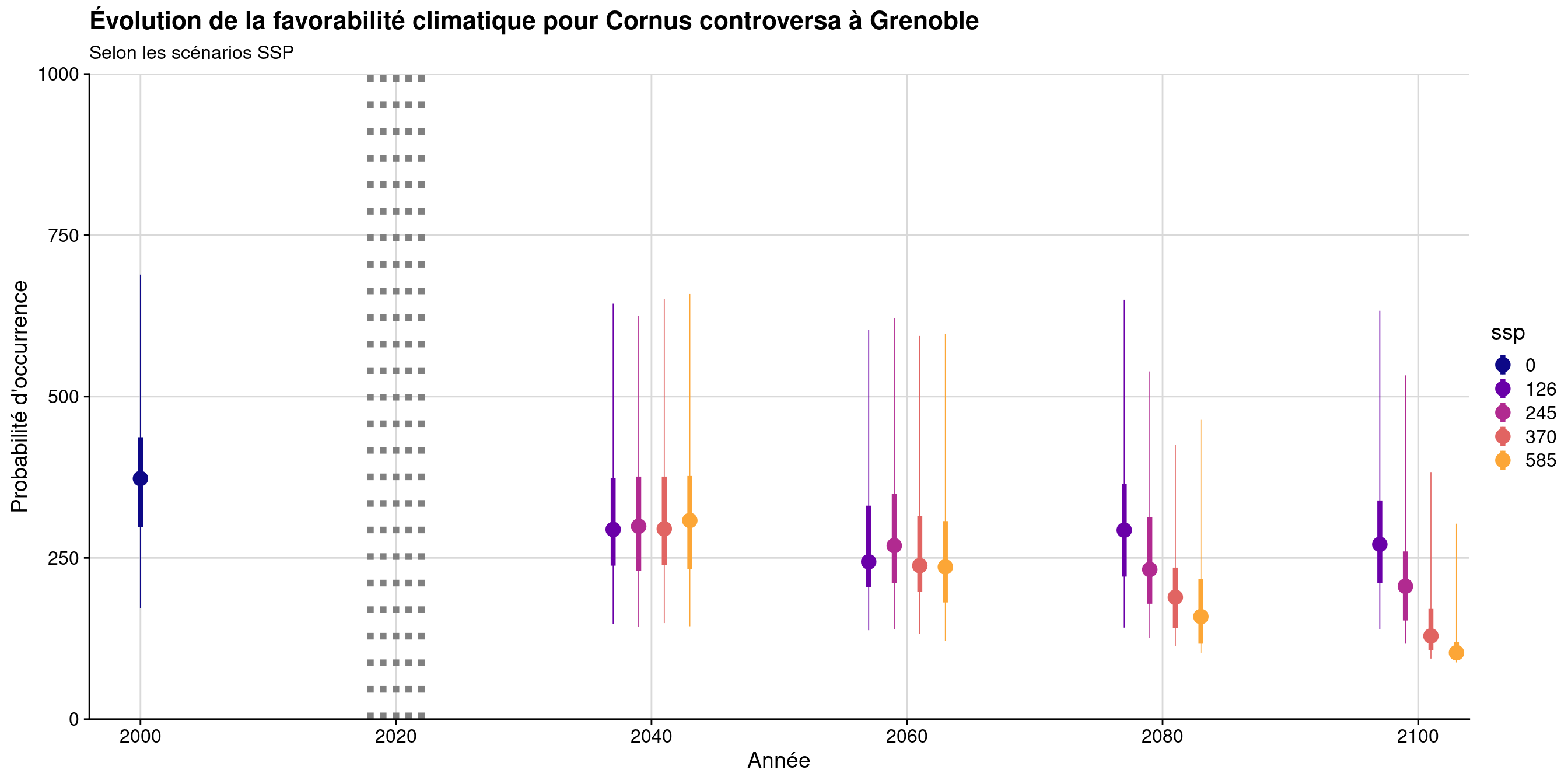
Figure 128.28: Évolution de la favorabilité climatique au cours du XXIème siècle. Le point représente la médiane sur l’étendue de la métropole (i.e. tous les pixels de la carte) ; le trait gras représente 50 % des données intermédiaires (espace inter-quartile) ; le trait fin représente le reste des données du minimum au maximum.