180 Sorbus aucuparia
180.1 Données d’occurrences GBIF
Collecte des données du GBIF :
<<gbif download metadata>>
Status: SUCCEEDED
DOI: 10.15468/dl.esyw9j
Format: SIMPLE_CSV
Download key: 0260104-230224095556074
Created: 2023-05-24T19:50:15.458+00:00
Modified: 2023-05-24T19:55:52.732+00:00
Download link: https://api.gbif.org/v1/occurrence/download/request/0260104-230224095556074.zip
Total records: 474154Conversion en objets géographiques (format Simple feature de sf) et
exploration rapide des données (classe du jeu de données et premières lignes du
tableau) :
Simple feature collection with 474154 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -152.403495 ymin: -51.806111 xmax: 176.262309 ymax: 71.090615
Geodetic CRS: WGS 84
# A tibble: 474,154 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
* <int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
2 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
3 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
4 3.e9 5f06e39d-… USFS - New … Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
5 1 e8 8354ecee-… 111313949 Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
6 1 e8 8354ecee-… 111313889 Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
7 1 e8 8354ecee-… 111313202 Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
8 2 e8 837381f4-… EMMA:EMMA:1… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
9 2 e8 837381f4-… EMMA:EMMA:7… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
10 2 e9 83fdfd3d-… INFOFLORA-T… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
# ℹ 474,144 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …Il y a 474 154 occurrences dans le jeu de données. On les affiche sur la carte du monde :
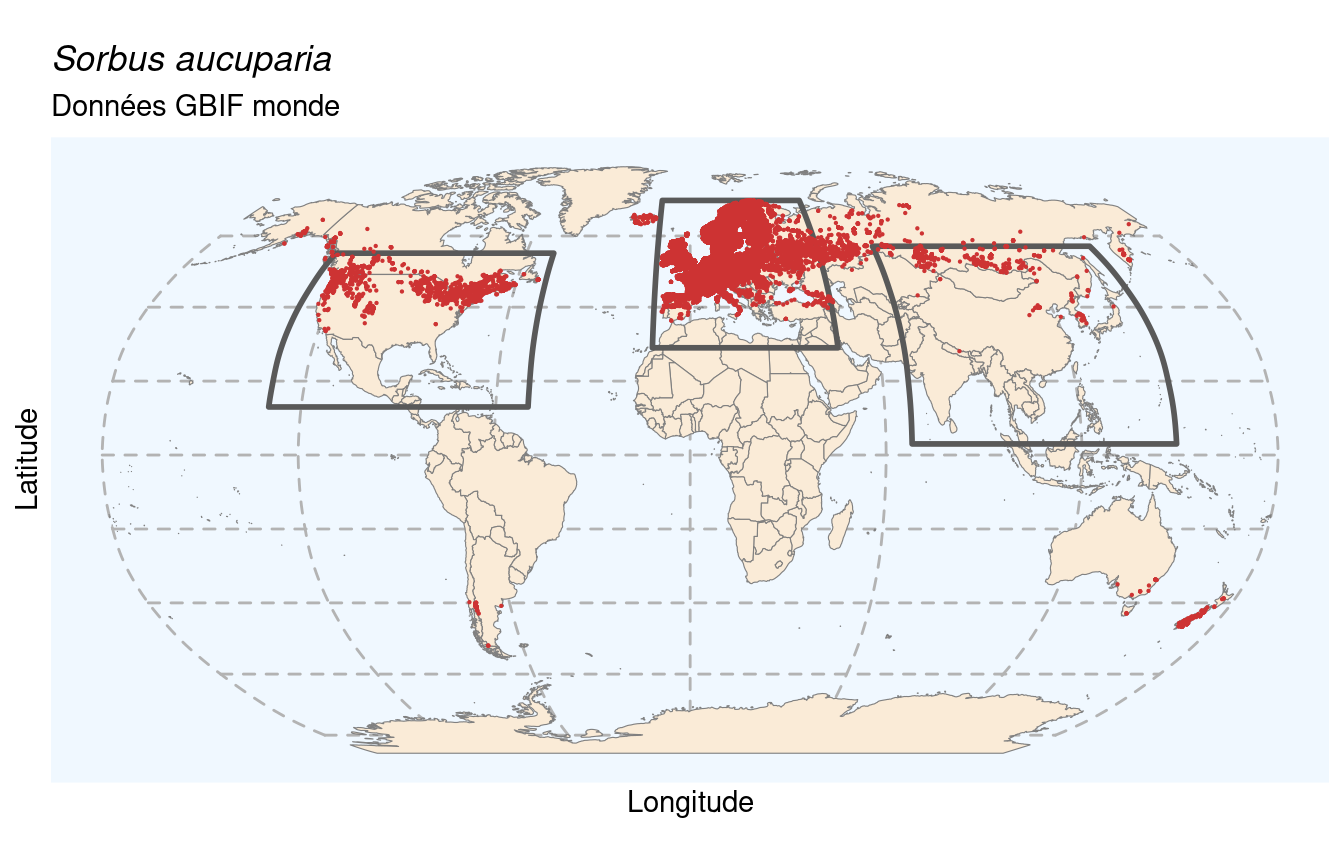
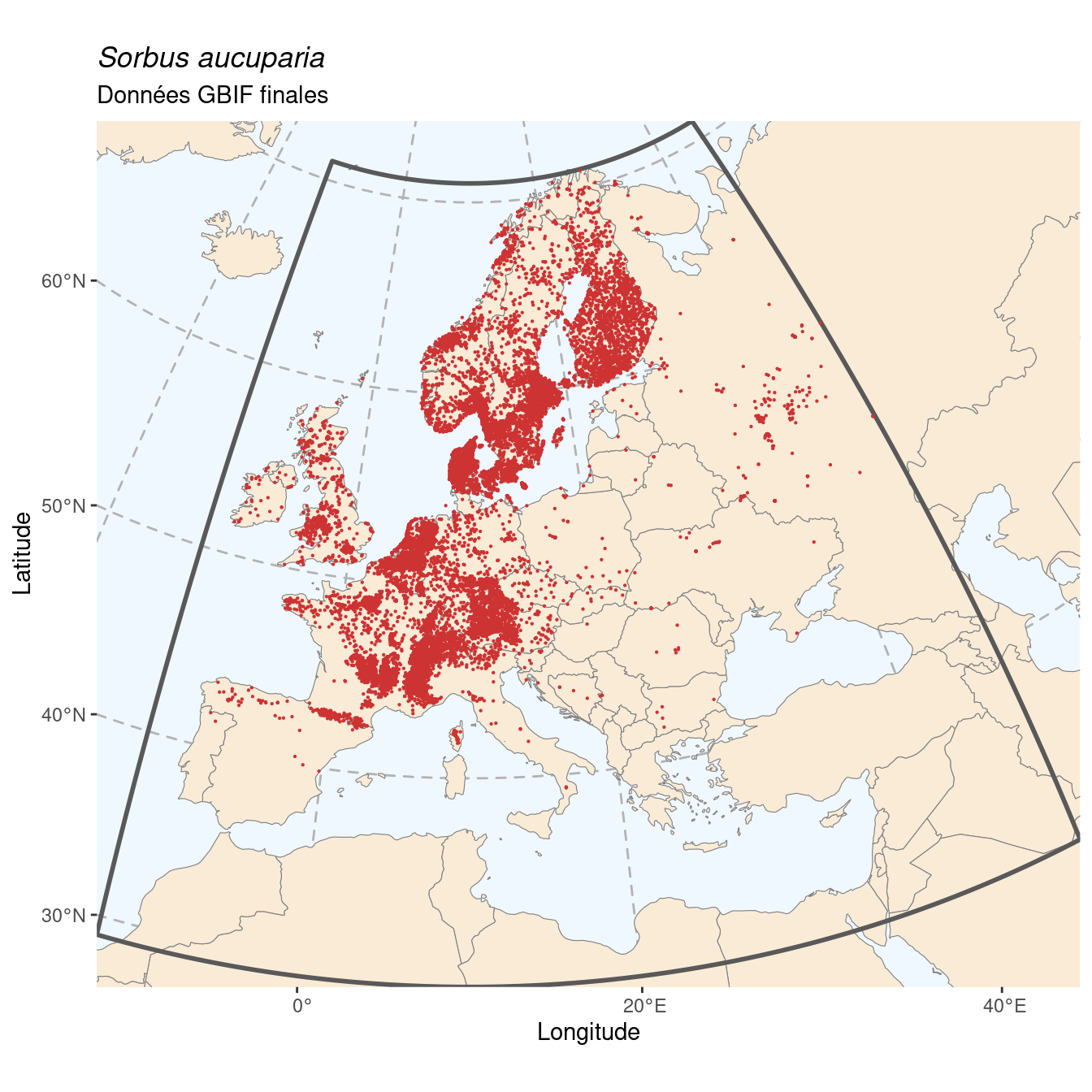
Figure 180.1: Occurrences de Sorbus aucuparia dans le monde.
180.1.1 Données par région
On évalue la proportion d’occurrences dans chaque région, en utilisant les 3 masques créés précédemment. Si on a au moins 50 % des occurrences dans une région, on peut continuer en travaillant uniquement sur ces données. Sinon, on vérifie qu’au moins 67 % des données tombent dans l’ensemble des régions, et on prend la région la plus représentée. Au final, s’il s’agit d’une espèce endémique d’Europe, on n’a pas besoin de manipulation supplémentaire.
[1] 0.9832965661[1] 0.006660283368[1] 0.004281309448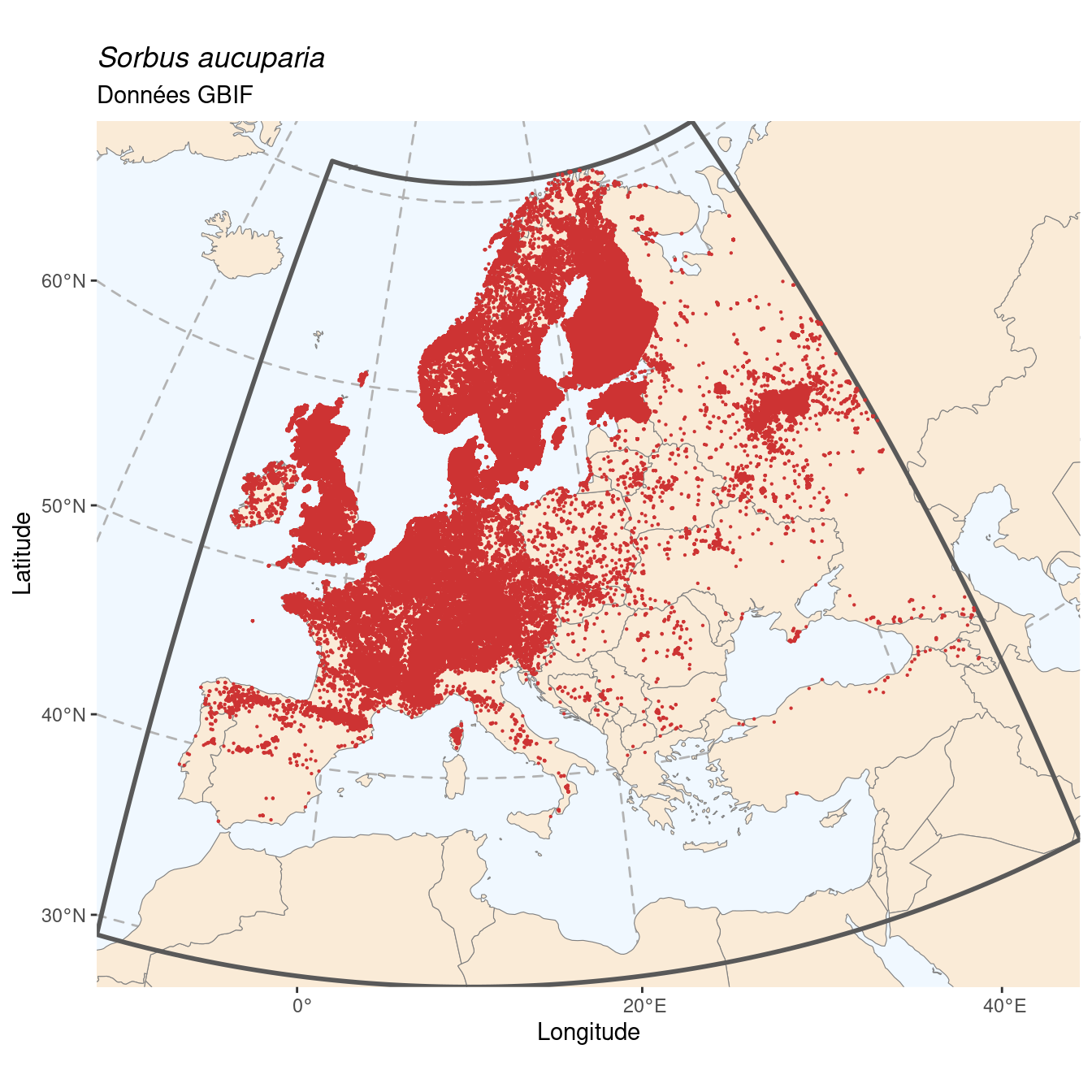
Figure 180.2: Occurrence de Sorbus aucuparia dans la région d’endémisme.
180.1.2 Licences
On s’intéresse ici au problème particulier des licences :
CC_BY_4_0 CC_BY_NC_4_0 CC0_1_0
317293 78325 70616 [1] 83.20049589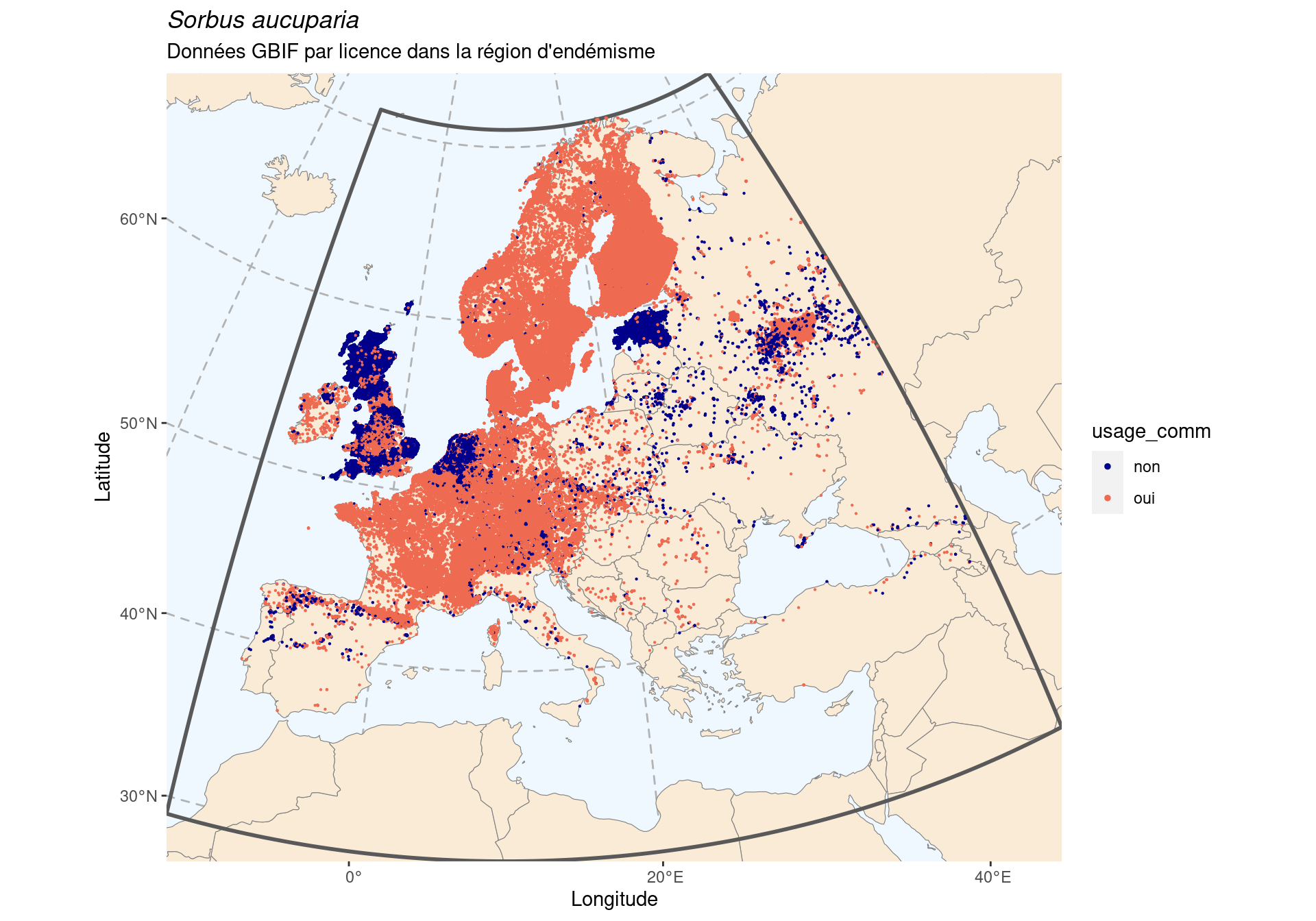
Figure 180.3: Occurrence de Sorbus aucuparia dans la région d’endémisme, selon la licence : CC BY-NC en bleu ; CC0 et CC BY en orange.
Idéalement, on aimerait avoir au moins 50 % de données vraiment libres
d’utilisation (c’est-à-dire sans la clause -NC). On continue en supprimant les
données en CC BY-NC.
[1] 387909180.1.3 Sous-échantillonnage
Si le jeu de données contient 25 000 occurrences ou plus, on procède au sous-échantillonnage aléatoire pour en conserver un maximum de 25 000.
Simple feature collection with 25000 features and 50 fields
Attribute-geometry relationship: 50 constant, 0 aggregate, 0 identity
Geometry type: POINT
Dimension: XY
Bounding box: xmin: -9.944698 ymin: 39.28806 xmax: 46.94 ymax: 70.938
Geodetic CRS: WGS 84
# A tibble: 25,000 × 51
gbifID datasetKey occurrenceID kingdom phylum class order family genus species infraspecificEpithet
<int64> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 1 e9 38b4c89f-… urn:lsid:ar… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
2 3.e9 688a357d-… ca051d65-69… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
3 2 e9 dd238f50-… 2c4672c8-f3… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
4 2 e9 38b4c89f-… urn:lsid:ar… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
5 3.e9 64dabd3c-… http://id.s… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
6 1 e9 75956ee6-… http://cbnm… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
7 1.e9 f2e389da-… http://tun.… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… "aucuparia"
8 3.e9 79d8c57d-… e06002b4-23… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
9 3.e9 c8916e4d-… urn:lsid:bi… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
10 3.e9 38b4c89f-… urn:lsid:ar… Plantae Trach… Magn… Rosa… Rosac… Sorb… Sorbus… ""
# ℹ 24,990 more rows
# ℹ 40 more variables: taxonRank <chr>, scientificName <chr>, verbatimScientificName <chr>,
# verbatimScientificNameAuthorship <chr>, countryCode <chr>, locality <chr>, stateProvince <chr>,
# occurrenceStatus <chr>, individualCount <int>, publishingOrgKey <chr>, decimalLatitude <dbl>,
# decimalLongitude <dbl>, coordinateUncertaintyInMeters <dbl>, coordinatePrecision <dbl>,
# elevation <dbl>, elevationAccuracy <dbl>, depth <dbl>, depthAccuracy <dbl>, eventDate <dttm>,
# day <int>, month <int>, year <int>, taxonKey <int>, speciesKey <int>, basisOfRecord <chr>, …180.2 Modélisation de la niche climatique
180.2.1 Préparer les données
Organisation des données d’entrées (occurrences et données environnementales) et des données produites de pseudo-absences :
class : SpatVector
geometry : points
dimensions : 25000, 0 (geometries, attributes)
extent : -9.944698, 46.94, 39.28806, 70.938 (xmin, xmax, ymin, ymax)
coord. ref. : lon/lat WGS 84 (EPSG:4326)
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= soau Data Formating -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
! Response variable is considered as only presences... Is it really what you want?
! No data has been set aside for modeling evaluation
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
Checking Pseudo-absence selection arguments...
> random pseudo absences selection
> Pseudo absences are selected in explanatory variables
! Some NAs have been automatically removed from your data
!!! Some data are located in the same raster cell.
Please set `filter.raster = TRUE` if you want an automatic filtering.
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.formated.data -=-=-=-=-=-=-=-=-=-=-=-=-=-=
dir.name = Output
sp.name = soau
24938 presences, 0 true absences and 72906 undefined points in dataset
6 explanatory variables
temp_max_august temp_min temp_wet_quart temp_season
Min. : 0.224 Min. :-22.004 Min. :-14.857 Min. : 197.0
1st Qu.:19.532 1st Qu.: -9.716 1st Qu.: 8.083 1st Qu.: 612.1
Median :22.400 Median : -4.224 Median : 12.535 Median : 745.1
Mean :24.183 Mean : -4.811 Mean : 11.777 Mean : 761.4
3rd Qu.:27.900 3rd Qu.: 0.380 3rd Qu.: 15.900 3rd Qu.: 906.6
Max. :45.452 Max. : 12.300 Max. : 24.786 Max. :1393.7
prec_wet_quart prec_season
Min. : 3.0 Min. : 4.63
1st Qu.: 179.0 1st Qu.: 22.62
Median : 218.0 Median : 31.06
Mean : 225.9 Mean : 35.41
3rd Qu.: 257.0 3rd Qu.: 39.31
Max. :1261.0 Max. :123.65
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=On visualise les pseudo-absences :
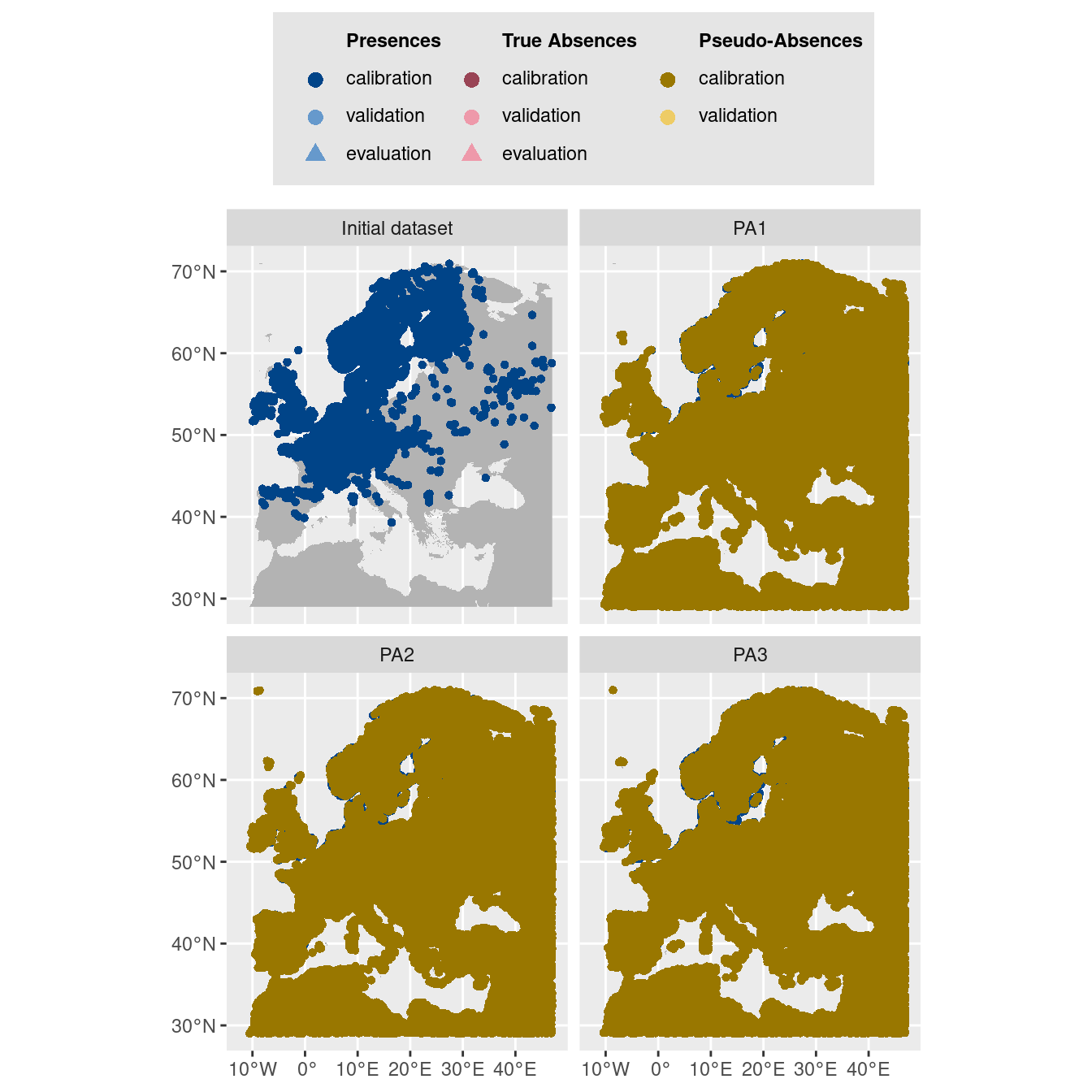
Figure 180.5: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
$data.vect
class : SpatVector
geometry : points
dimensions : 174752, 2 (geometries, attributes)
extent : -10.52083, 46.97917, 29.02083, 70.97917 (xmin, xmax, ymin, ymax)
coord. ref. :
names : resp dataset
type : <num> <chr>
values : 10 Initial dataset
10 Initial dataset
10 Initial dataset
$data.label
9 10
"**Presences**" "Presences (calibration)"
11 12
"Presences (validation)" "Presences (evaluation)"
19 20
"**True Absences**" "True Absences (calibration)"
21 22
"True Absences (validation)" "True Absences (evaluation)"
29 30
"**Pseudo-Absences**" "Pseudo-Absences (calibration)"
31 1
"Pseudo-Absences (validation)" "Background"
$data.plot
Figure 180.6: Les données d’occurrences (en haut à gauche) et les trois jeux de données aléatoires de pseudo-absences.
180.2.2 Modèles individuels de niche
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Build Single Models -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
Checking Models arguments...
Creating suitable Workdir...
! Weights where automatically defined for soau_PA1 to rise a 0.5 prevalence !
! Weights where automatically defined for soau_PA2 to rise a 0.5 prevalence !
! Weights where automatically defined for soau_PA3 to rise a 0.5 prevalence !
-=-=-=-=-=-=-=-=-=-=-=-=-=-= soau Modeling Summary -=-=-=-=-=-=-=-=-=-=-=-=-=-=
6 environmental variables ( temp_max_august temp_min temp_wet_quart temp_season prec_wet_quart prec_season )
Number of evaluation repetitions : 1
Models selected : GAM MARS MAXNET GBM ANN RF
Total number of model runs: 18
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=
-=-=-=- Run : soau_PA1
-=-=-=--=-=-=- soau_PA1_RUN1
-=-=-=- Run : soau_PA2
-=-=-=--=-=-=- soau_PA2_RUN1
-=-=-=- Run : soau_PA3
-=-=-=--=-=-=- soau_PA3_RUN1
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-= Done -=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=180.2.2.1 Évaluation de la performance
full.name PA run algo metric.eval cutoff sensitivity
1 soau_PA1_RUN1_GAM PA1 RUN1 GAM TSS 545.0 89.439
2 soau_PA1_RUN1_GAM PA1 RUN1 GAM ROC 542.5 89.524
3 soau_PA1_RUN1_MARS PA1 RUN1 MARS TSS 470.0 88.496
4 soau_PA1_RUN1_MARS PA1 RUN1 MARS ROC 439.5 90.040
5 soau_PA1_RUN1_MAXNET PA1 RUN1 MAXNET TSS 372.0 89.574
6 soau_PA1_RUN1_MAXNET PA1 RUN1 MAXNET ROC 370.5 89.749
specificity calibration validation evaluation
1 67.295 0.567 0.584 NA
2 67.215 0.815 0.817 NA
3 82.295 0.708 0.738 NA
4 80.785 0.927 0.937 NA
5 80.860 0.704 0.728 NA
6 80.735 0.926 0.936 NA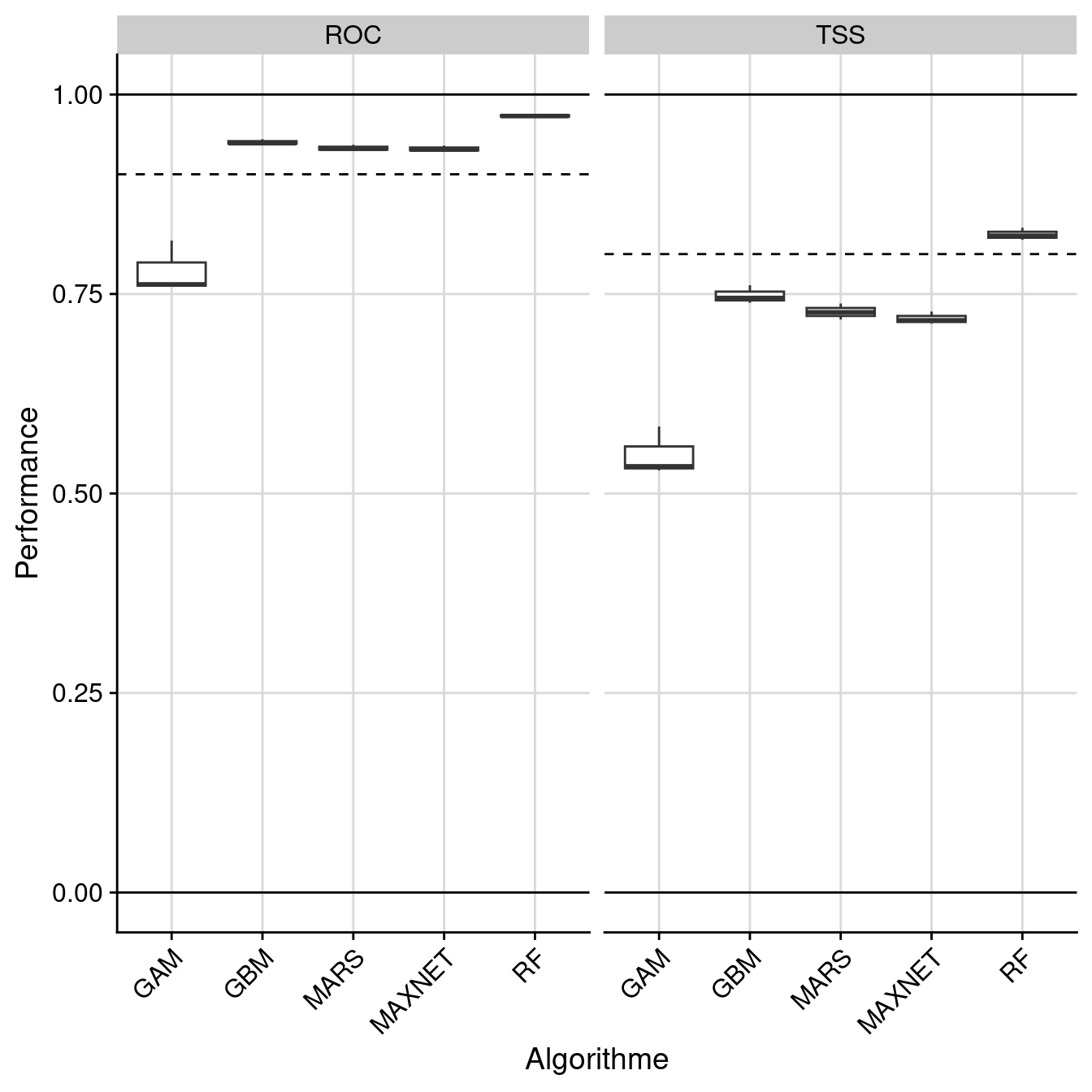
Figure 180.7: Évaluation de la performance des modèles individuels : True Skill Statistic & aire sous la courbe de ROC.
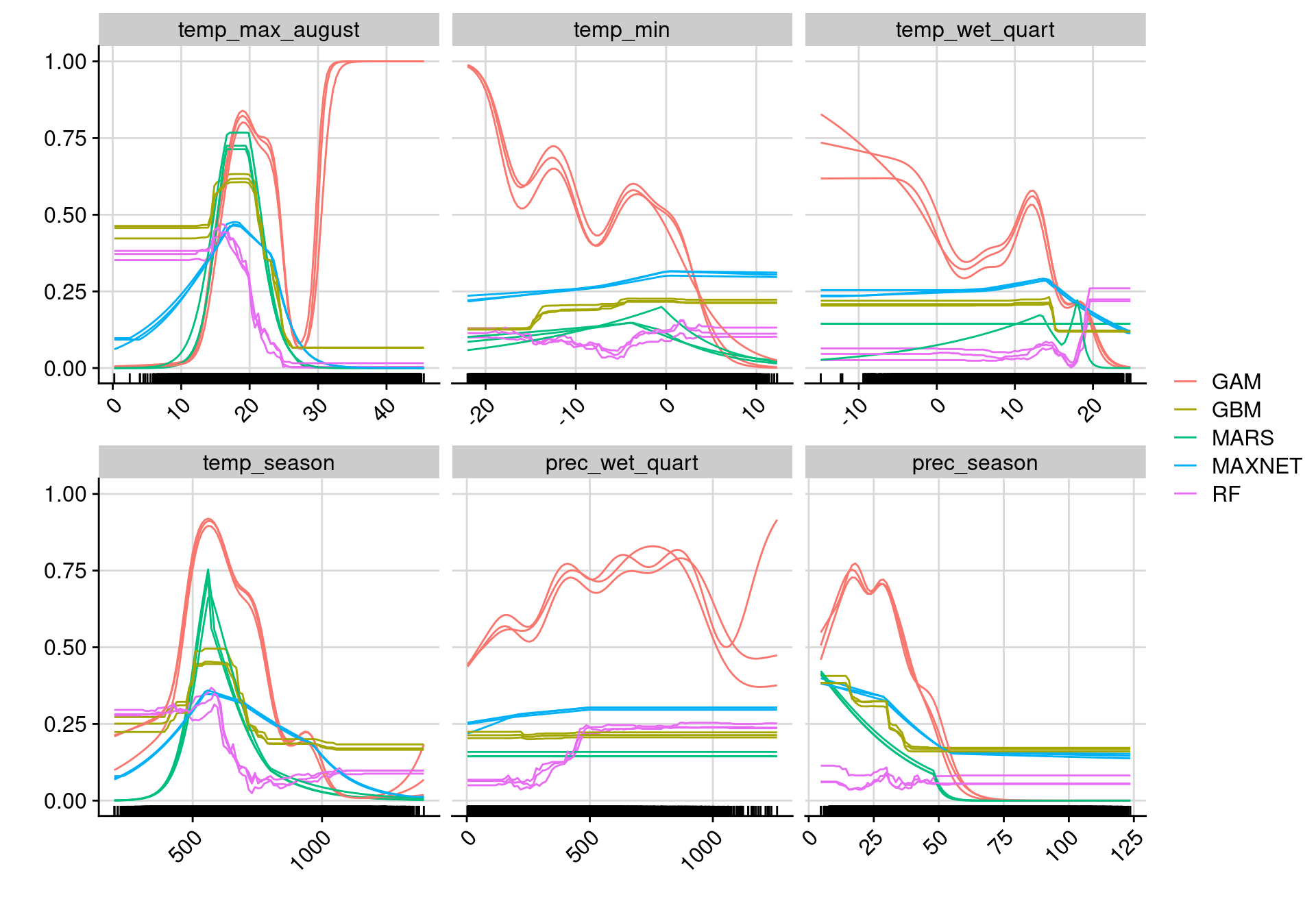
180.2.2.2 Importance des variables environnementales
full.name PA run algo expl.var rand var.imp
1 soau_PA1_RUN1_GAM PA1 RUN1 GAM temp_max_august 1 0.436777
2 soau_PA1_RUN1_GAM PA1 RUN1 GAM temp_min 1 0.050946
3 soau_PA1_RUN1_GAM PA1 RUN1 GAM temp_wet_quart 1 0.039165
4 soau_PA1_RUN1_GAM PA1 RUN1 GAM temp_season 1 0.436570
5 soau_PA1_RUN1_GAM PA1 RUN1 GAM prec_wet_quart 1 0.012728
6 soau_PA1_RUN1_GAM PA1 RUN1 GAM prec_season 1 0.255369
Figure 180.8: Pourcentage de variance expliquée pour chaque variable, décomposé par algorithme.
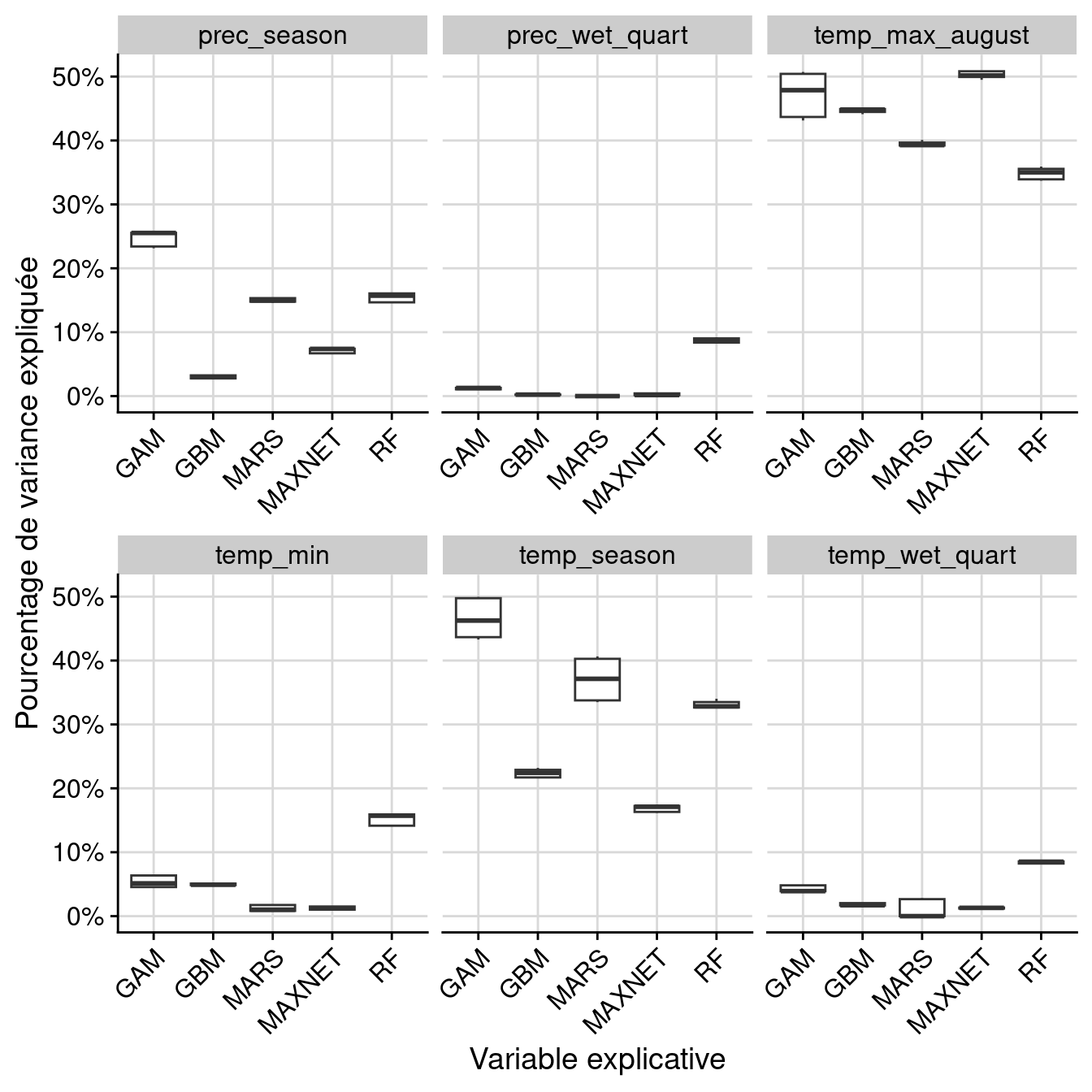
Figure 180.9: Pourcentage de variance expliquée pour chaque variable, décomposé par variable.
180.2.3 Modèles d’ensemble de niche
Seulement modèles avec TSS > 0.8
-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.ensemble.models.out -=-=-=-=-=-=-=-=-=-=-=-=-=
sp.name : soau
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
models computed:
soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo, soau_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
models failed: none
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=180.2.3.1 Évaluation de la qualité
full.name merged.by.PA merged.by.run
1 soau_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
2 soau_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
merged.by.algo filtered.by algo metric.eval cutoff sensitivity specificity
1 mergedAlgo TSS EMwmean TSS 606.0 96.271 94.386
2 mergedAlgo TSS EMwmean ROC 612.5 96.114 94.545
calibration validation evaluation
1 0.906 NA NA
2 0.992 NA NA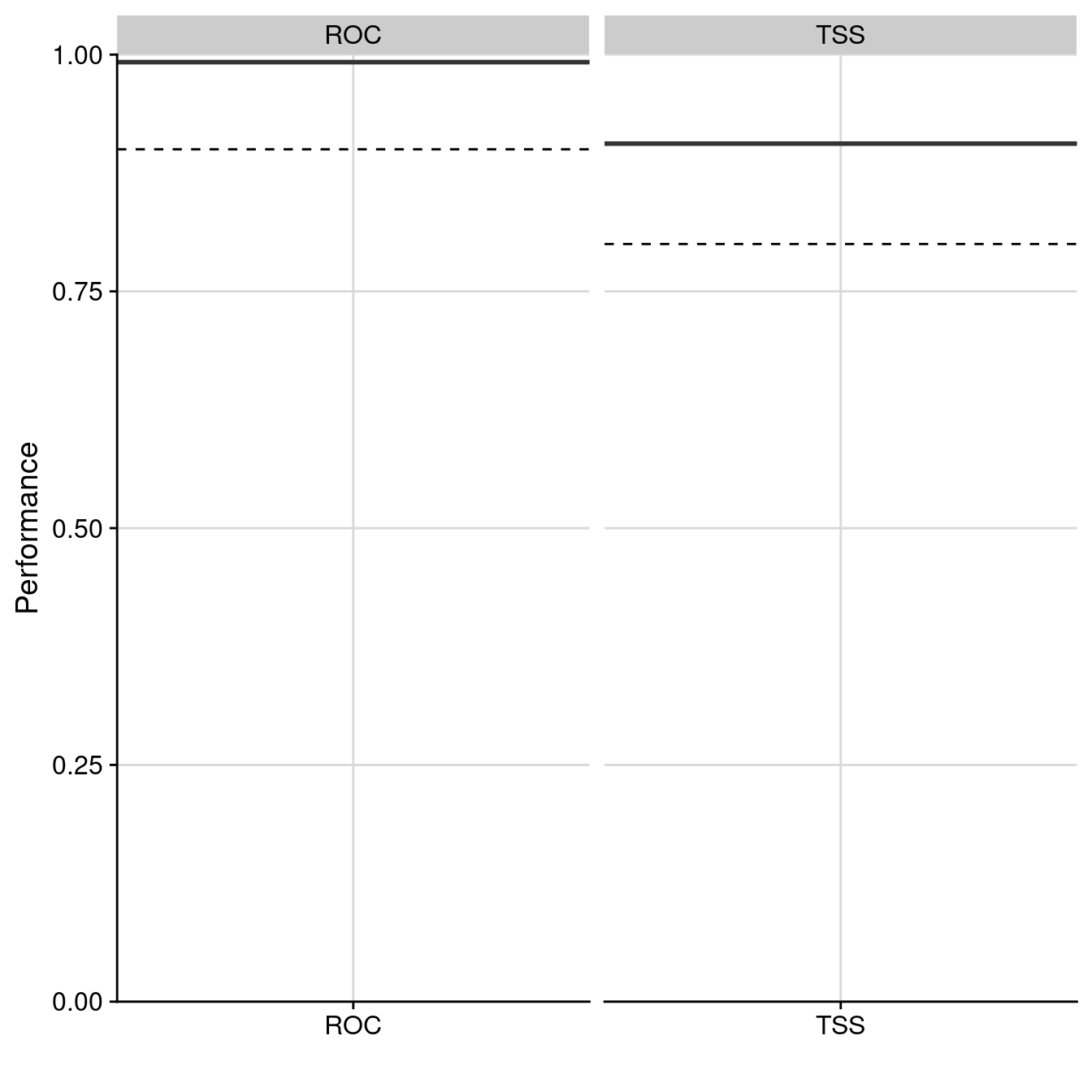
Figure 180.11: Évaluation de la qualité du modèle d’ensemble : Aire sous la courbe de ROC & True Skill Statistic.
full.name merged.by.PA merged.by.run
1 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
2 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
3 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
4 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
5 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
6 soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo mergedData mergedRun
merged.by.algo filtered.by algo expl.var rand var.imp
1 mergedAlgo TSS EMcv temp_max_august 1 0.595835
2 mergedAlgo TSS EMcv temp_min 1 0.476368
3 mergedAlgo TSS EMcv temp_wet_quart 1 0.404059
4 mergedAlgo TSS EMcv temp_season 1 0.502399
5 mergedAlgo TSS EMcv prec_wet_quart 1 0.379918
6 mergedAlgo TSS EMcv prec_season 1 0.443656Par variable :
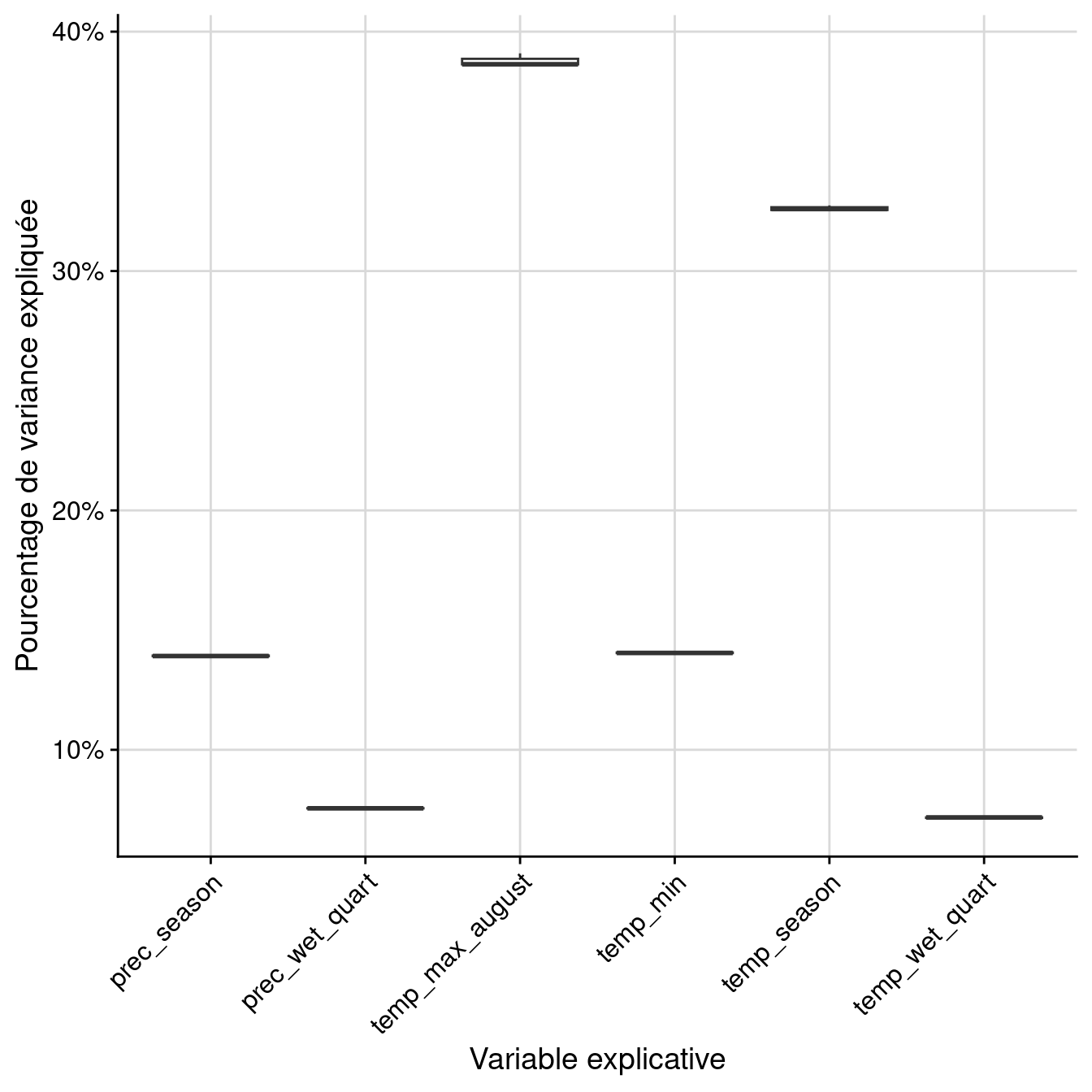
Figure 180.12: Pourcentage de variance expliquée par variable dans le modèle d’ensemble.
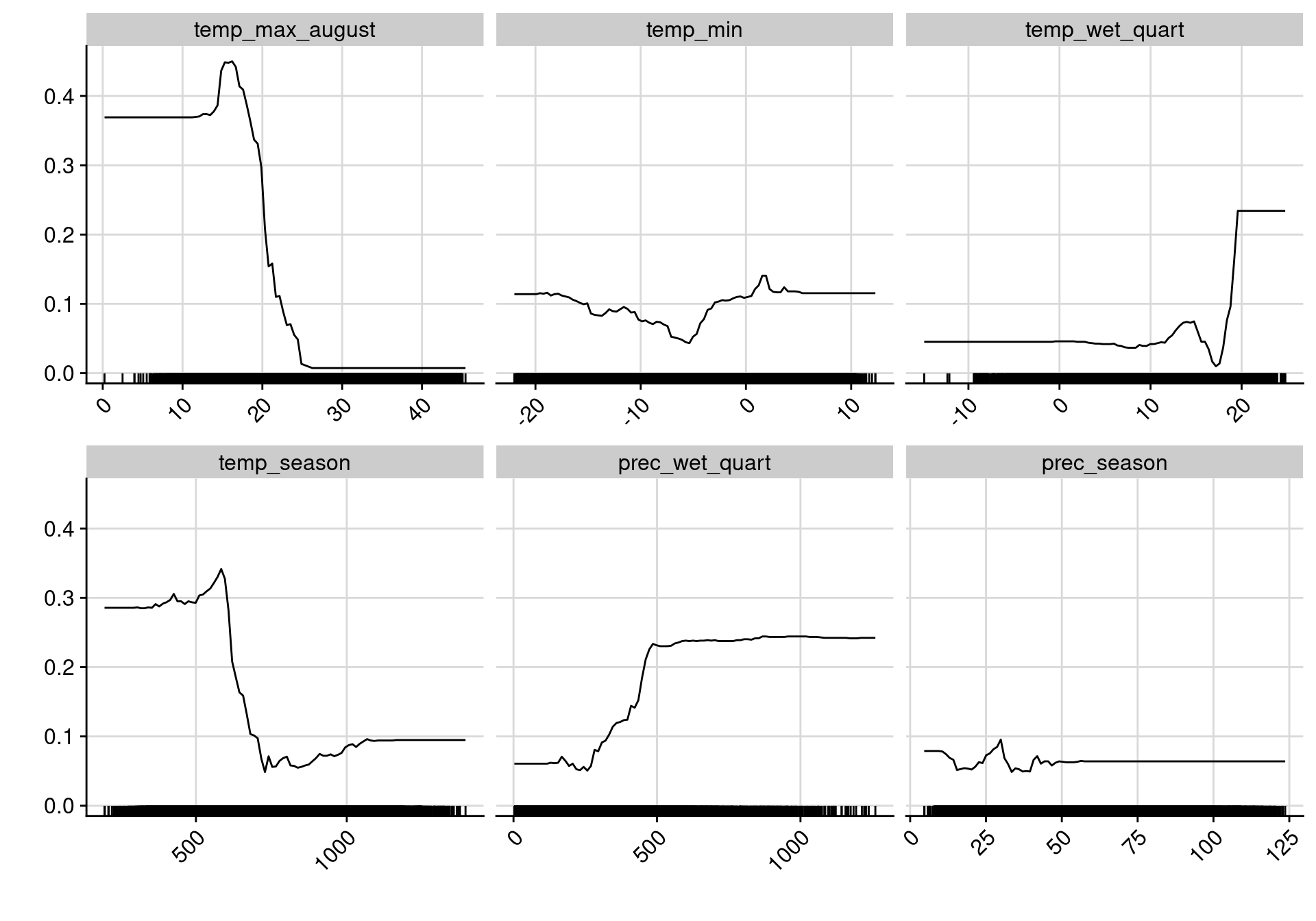
Figure 180.13: Courbes de réponse de chaque variable climatique dans le modèle d’ensemble.
180.3 Projections
180.3.1 Distribution potentielle contemporaine
Contemporaine : Cartographie des observations sur couches climatiques Extraction des conditions climatiques favorables
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/soau/current
sp.name : soau
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/soau/soau.AllModels.models.out )
models.projected :
soau_PA1_RUN1_GAM, soau_PA1_RUN1_MARS, soau_PA1_RUN1_MAXNET, soau_PA1_RUN1_GBM, soau_PA1_RUN1_RF, soau_PA2_RUN1_GAM, soau_PA2_RUN1_MARS, soau_PA2_RUN1_MAXNET, soau_PA2_RUN1_GBM, soau_PA2_RUN1_RF, soau_PA3_RUN1_GAM, soau_PA3_RUN1_MARS, soau_PA3_RUN1_MAXNET, soau_PA3_RUN1_GBM, soau_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=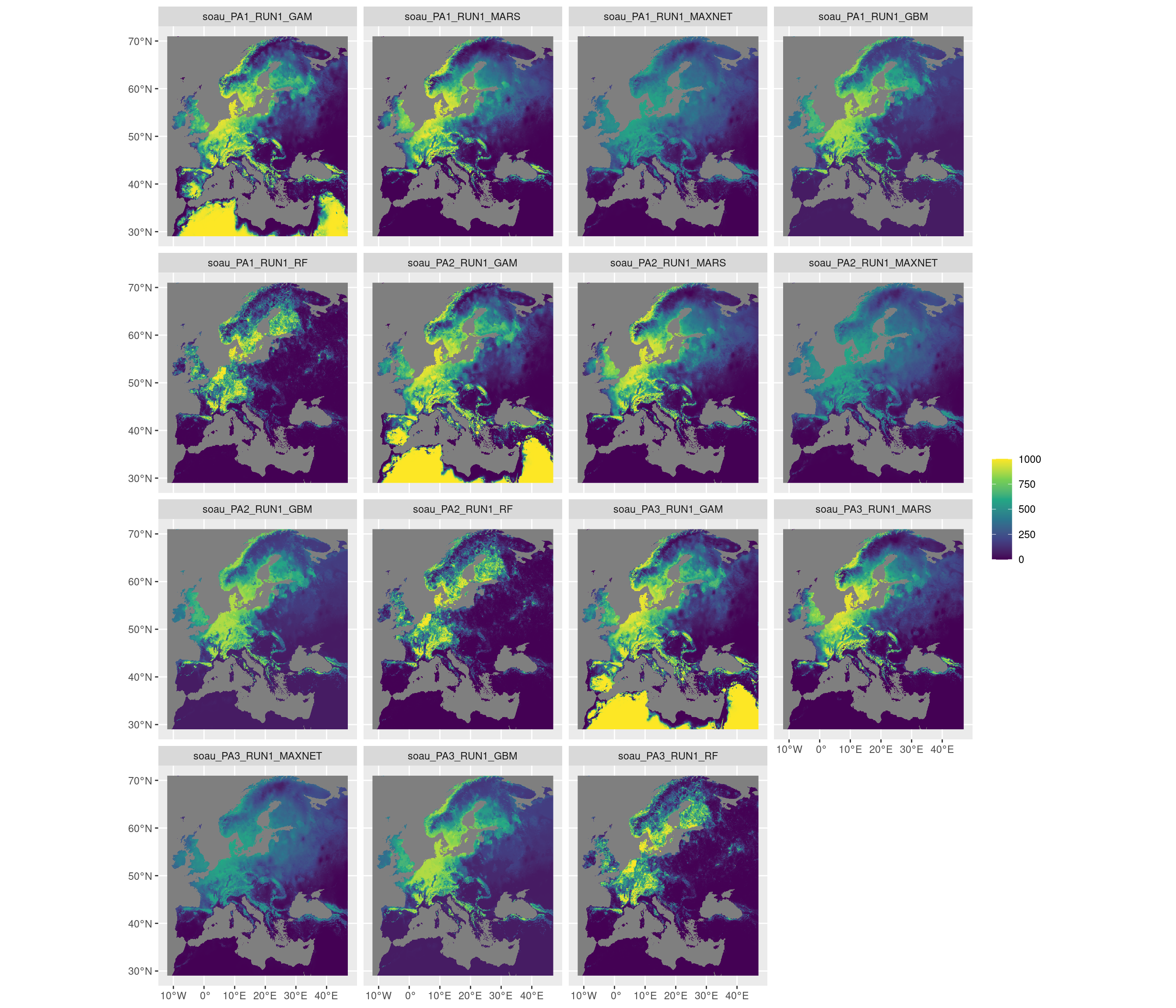
Figure 180.14: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
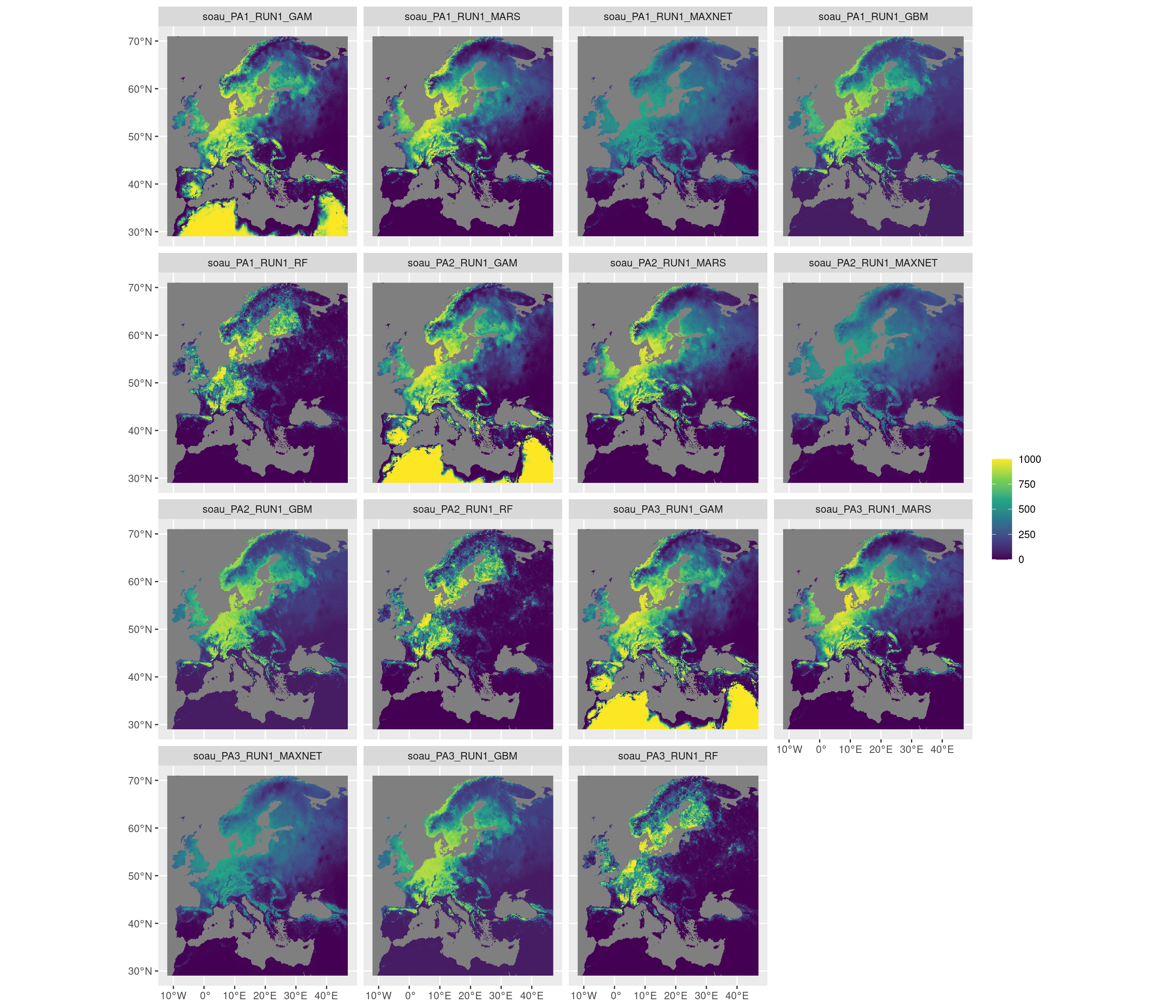
Figure 180.15: Projection de la distribution potentielle contemporaine sur l’Europe, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/soau/current
sp.name : soau
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/soau/soau.AllModels.ensemble.models.out )
models.projected :
soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo, soau_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=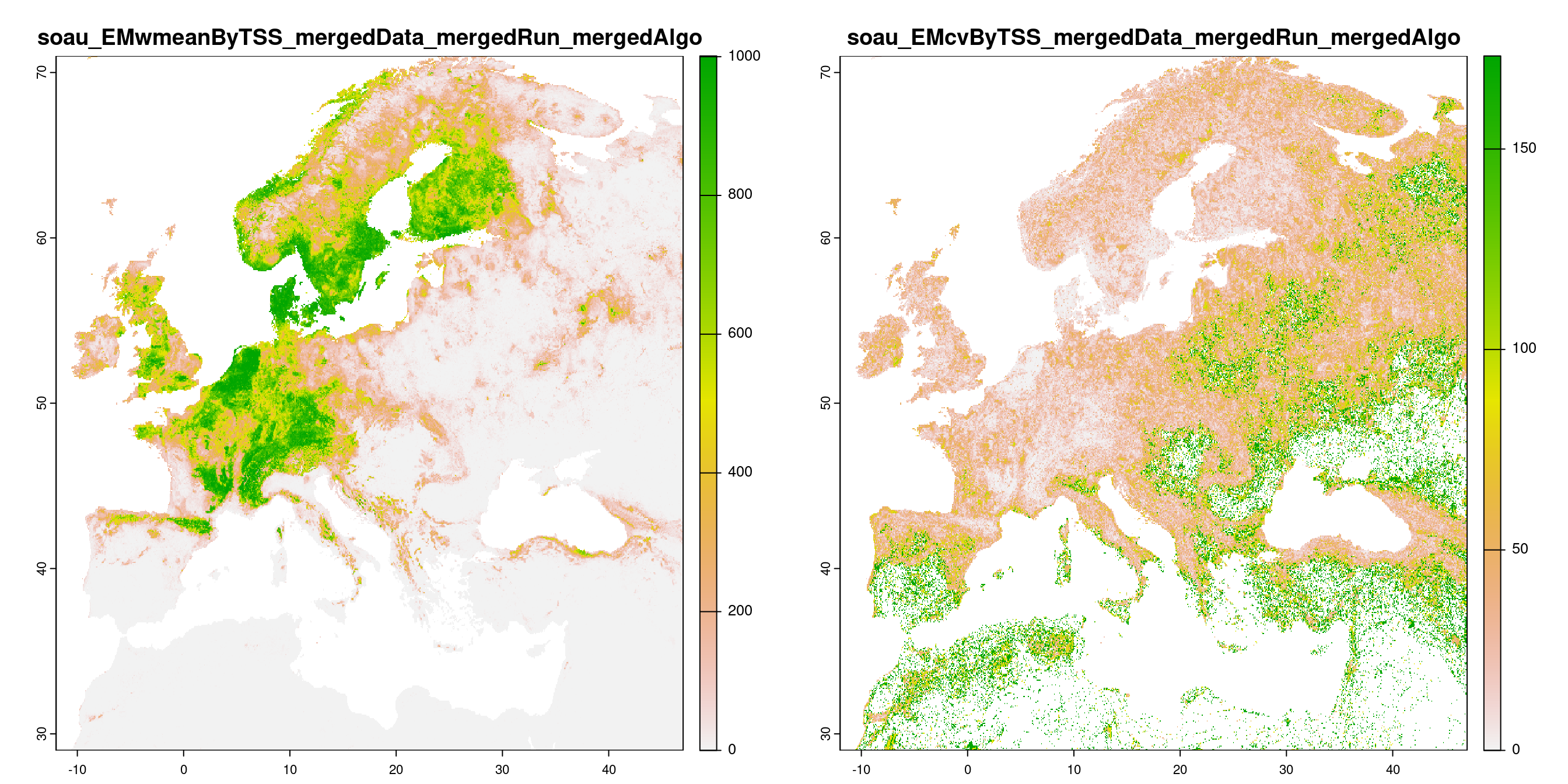
Figure 180.16: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) ; B) Incertitude associée à la projection.
180.3.2 Interpolation à l’échelle de la métropole
- → conditions climatiques favorables à fine échelle
On projette la niche climatique sur ce raster à fine échelle.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/soau/cont_gre
sp.name : soau
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/soau/soau.AllModels.models.out )
models.projected :
soau_PA1_RUN1_GAM, soau_PA1_RUN1_MARS, soau_PA1_RUN1_MAXNET, soau_PA1_RUN1_GBM, soau_PA1_RUN1_RF, soau_PA2_RUN1_GAM, soau_PA2_RUN1_MARS, soau_PA2_RUN1_MAXNET, soau_PA2_RUN1_GBM, soau_PA2_RUN1_RF, soau_PA3_RUN1_GAM, soau_PA3_RUN1_MARS, soau_PA3_RUN1_MAXNET, soau_PA3_RUN1_GBM, soau_PA3_RUN1_RF
available binary projection : TSS, ROC
available filtered projection : TSS, ROC
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=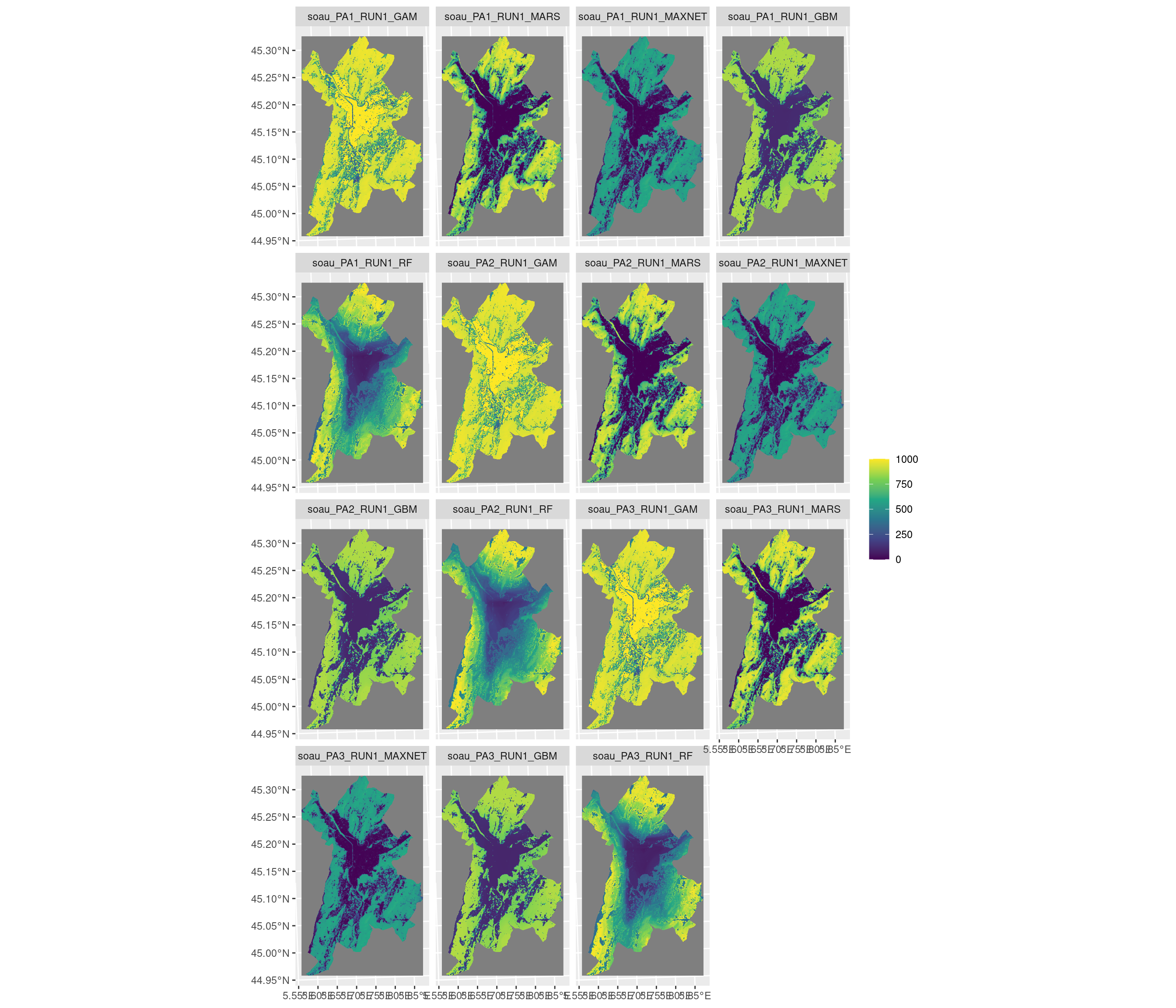
Figure 180.17: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.

Figure 180.18: Projection de la distribution potentielle contemporaine sur la métropole de Grenoble, selon les 6 algorithmes et les 3 runs.
-=-=-=-=-=-=-=-=-=-=-=-=-=-= BIOMOD.projection.out -=-=-=-=-=-=-=-=-=-=-=-=-=-=
Projection directory : Output/soau/cont_gre
sp.name : soau
expl.var.names : temp_max_august temp_min temp_wet_quart temp_season
prec_wet_quart prec_season
modeling.id : AllModels ( Output/soau/soau.AllModels.ensemble.models.out )
models.projected :
soau_EMcvByTSS_mergedData_mergedRun_mergedAlgo, soau_EMwmeanByTSS_mergedData_mergedRun_mergedAlgo
-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=-=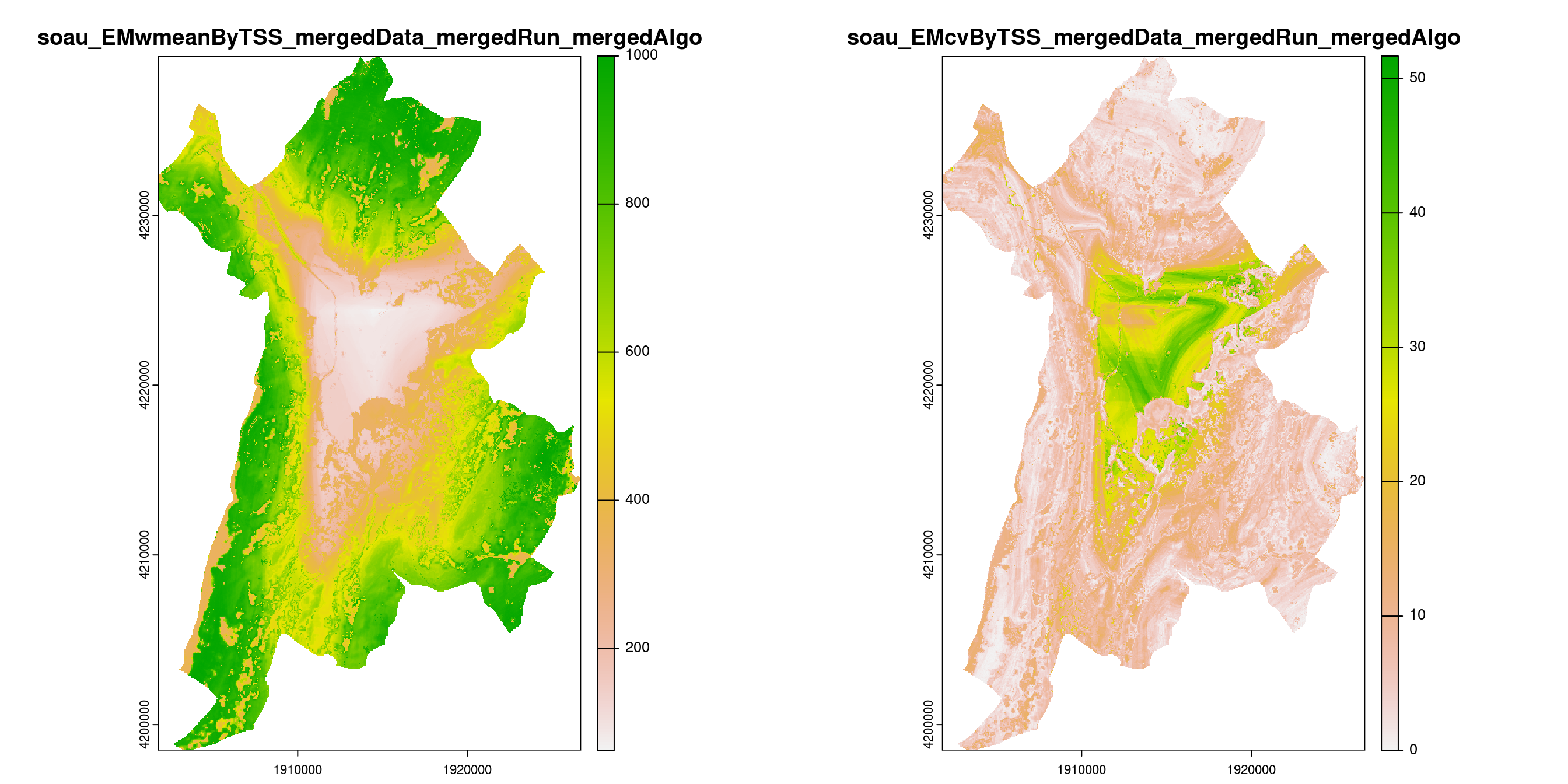
Figure 180.19: A) Projection de la distribution potentielle contemporaine (modèle d’ensemble) sur la métropole de Grenoble ; B) Incertitude associée à la projection.
180.3.3 Distributions potentielles futures
Futur : Projection à l’échelle de l’Europe jusqu’à l’horizon 2100
Toutes les fenêtres temporelles × scénarios pour le GCM « IPSL-CM6A-LR »
- SSP1-2.6
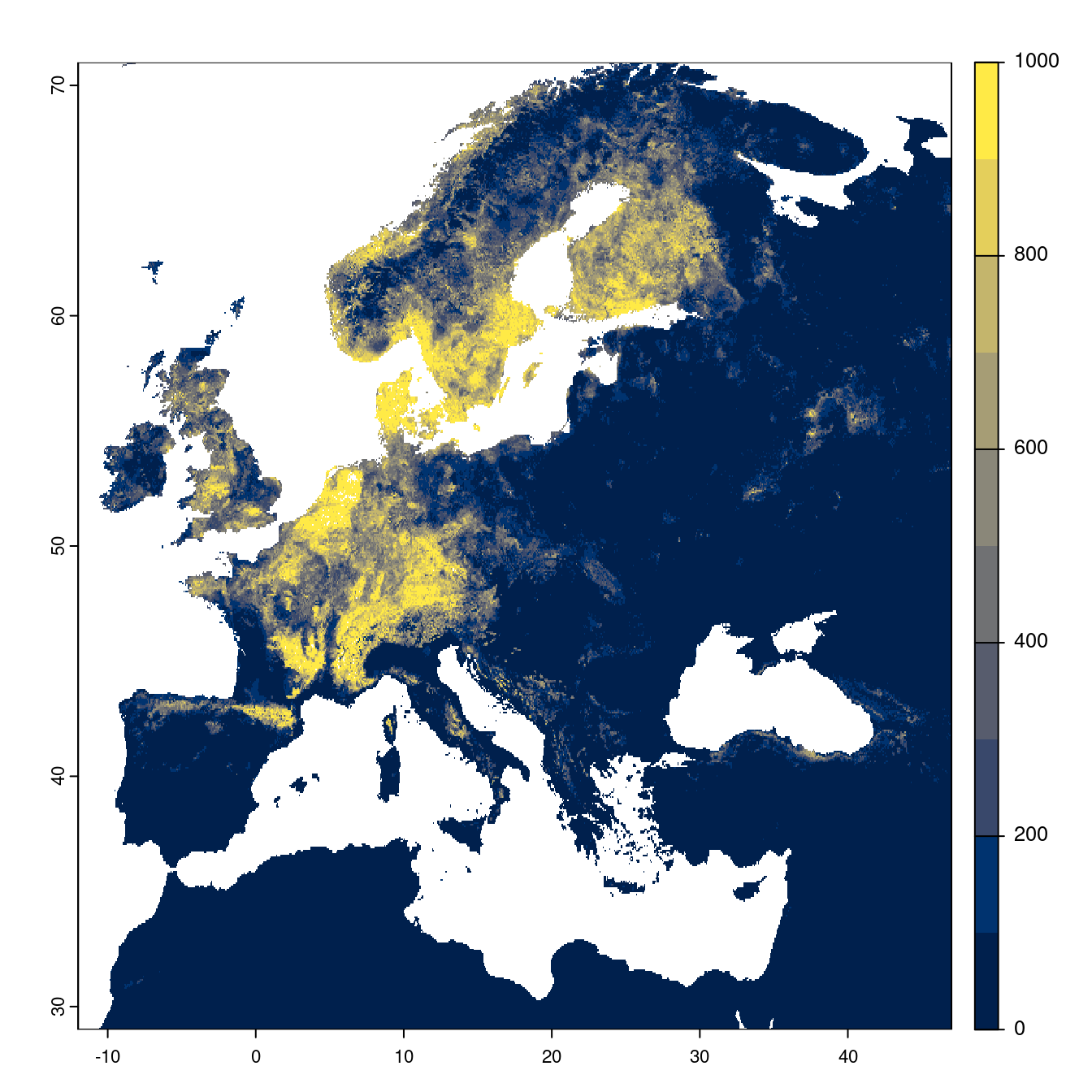
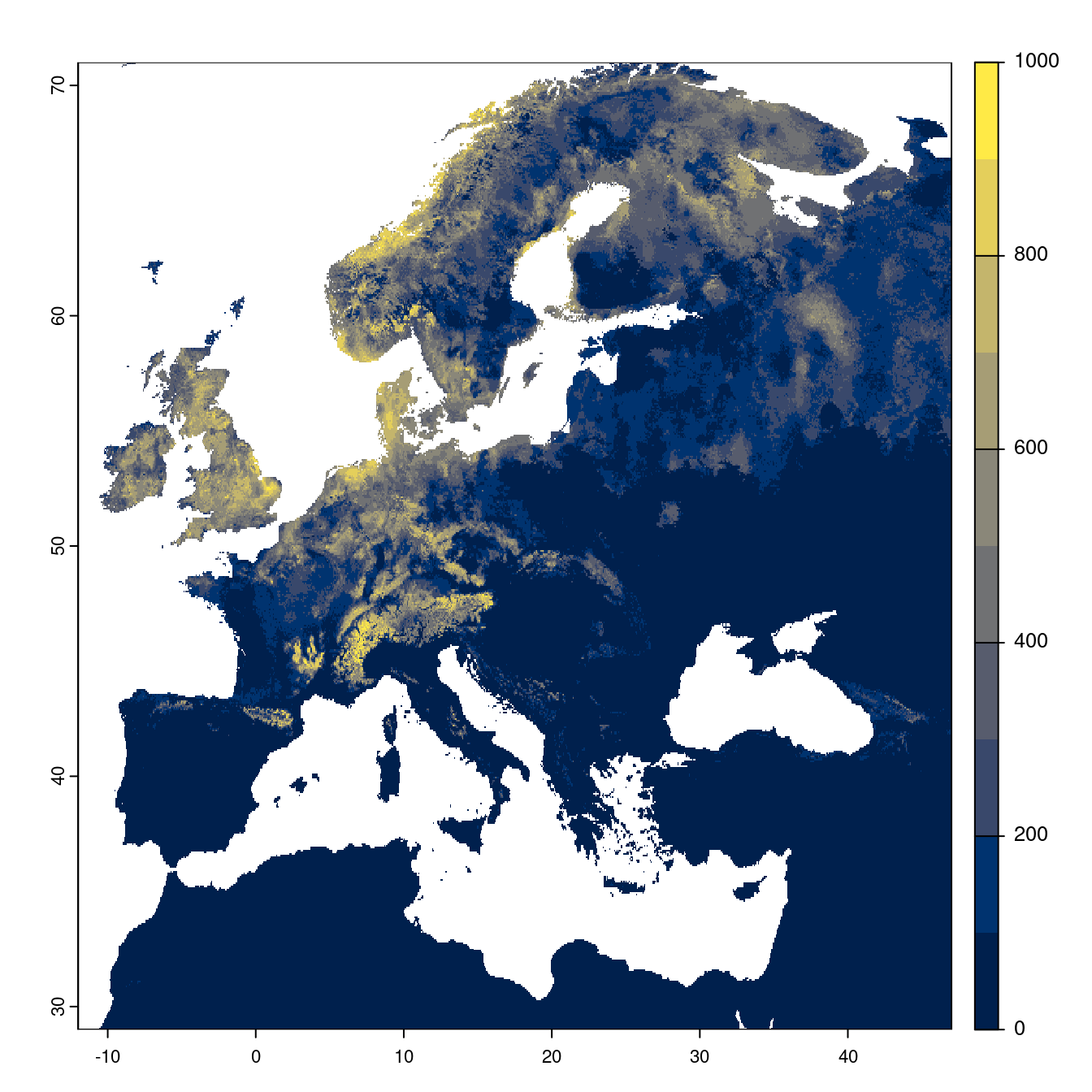
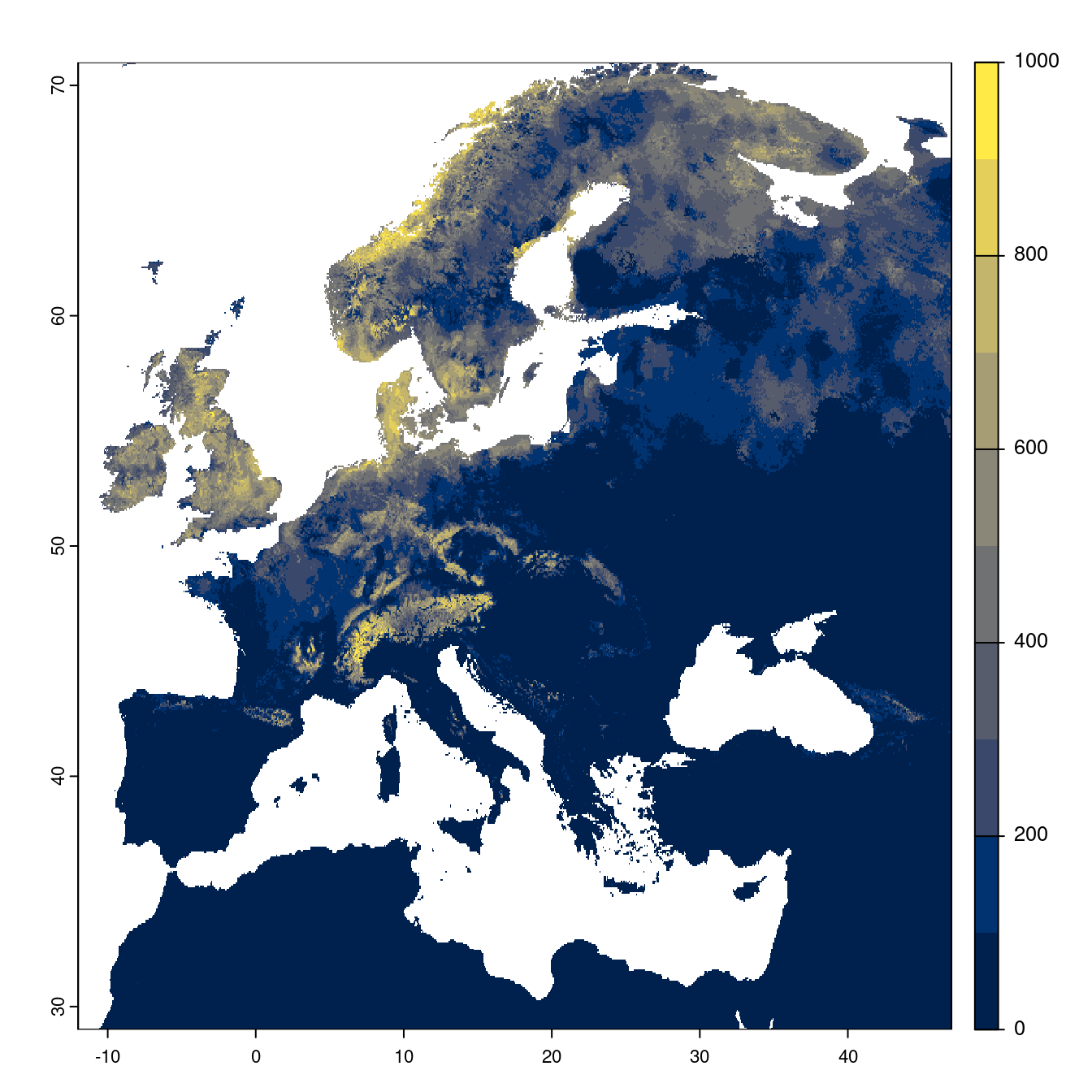
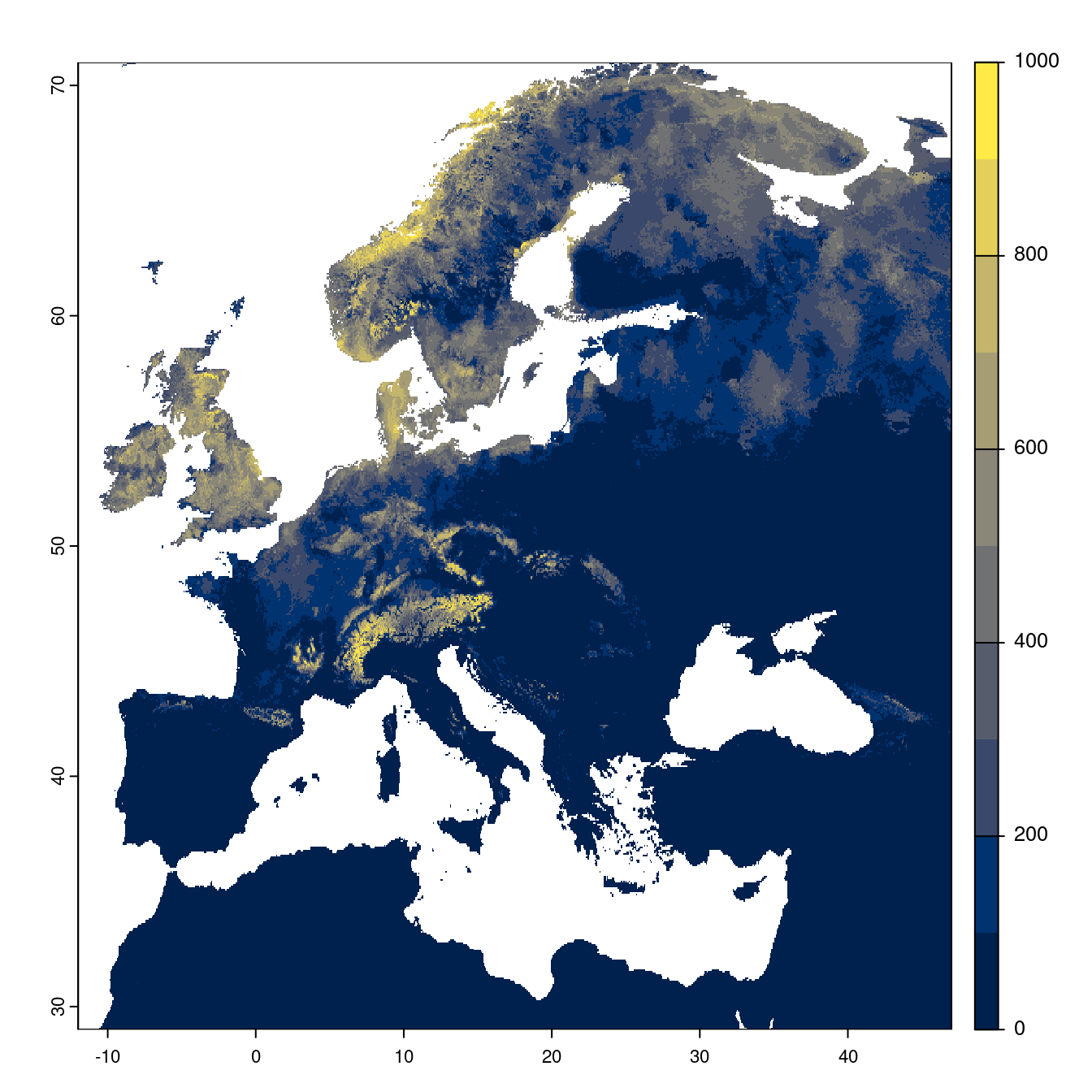
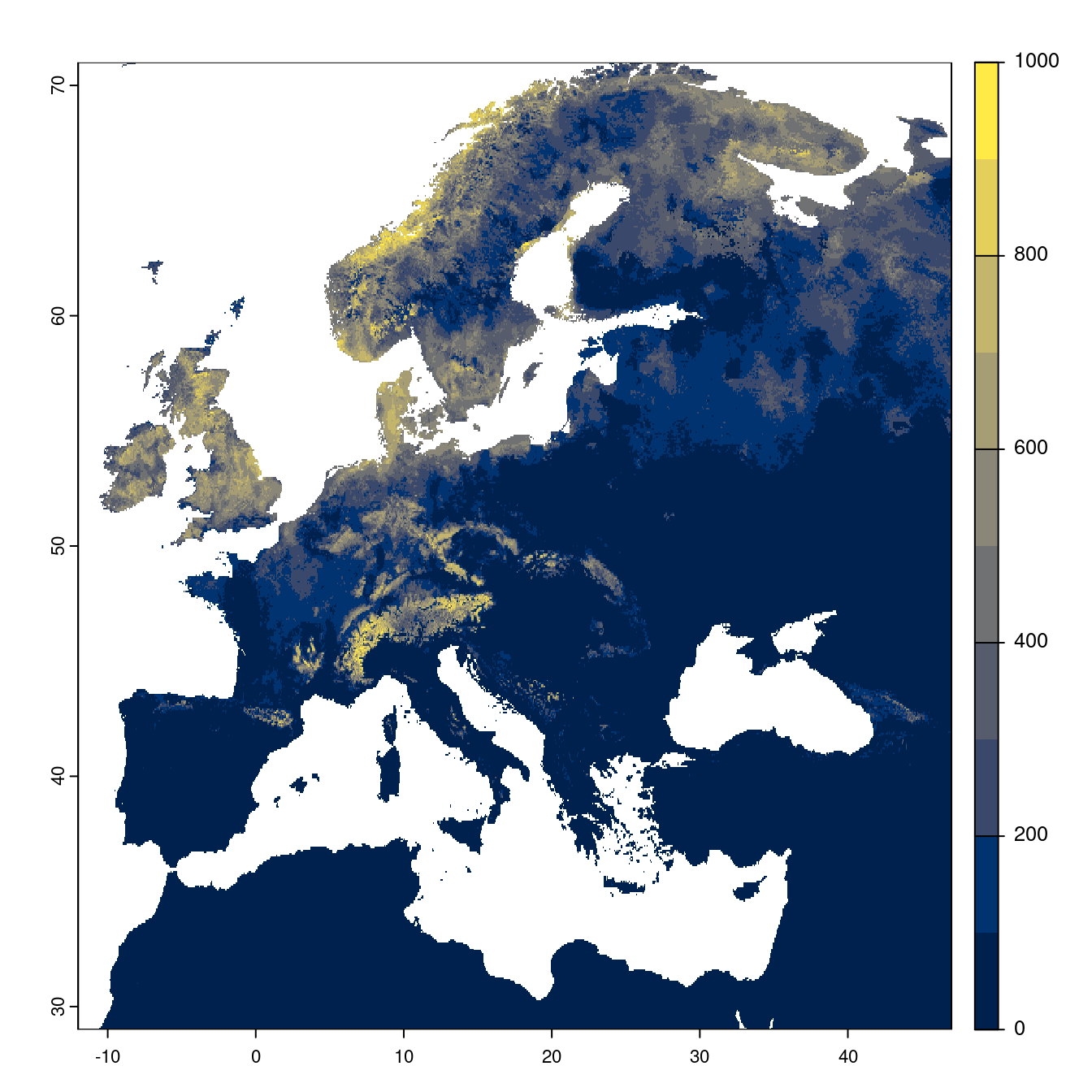
Figure 180.20: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP1-2.6 (modèle d’ensemble).
On obtient pour la métrique TSS une carte unique de moyenne pondérée sur les algorithmes et sur les jeux de pseudo-absences.
- SSP2-4.5
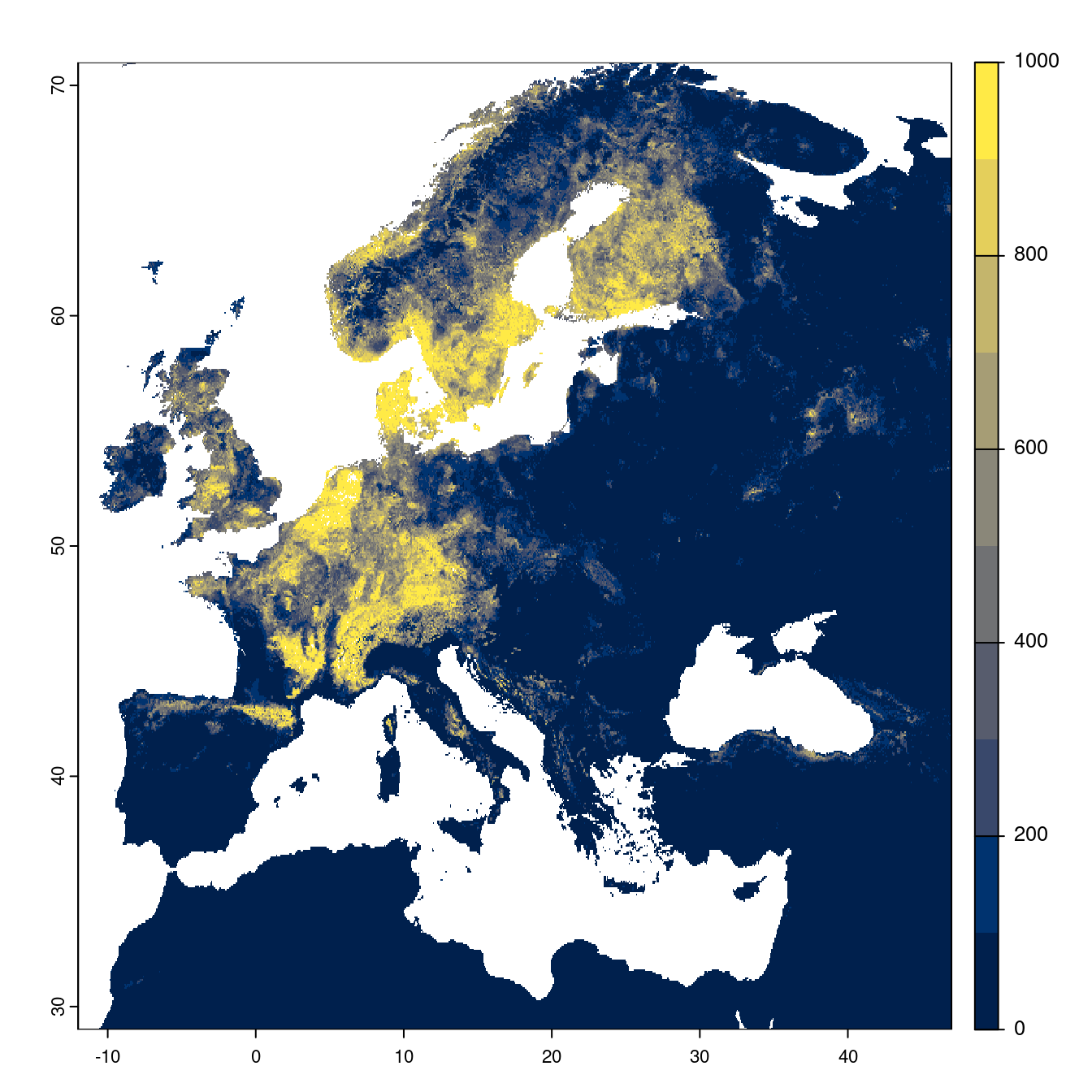
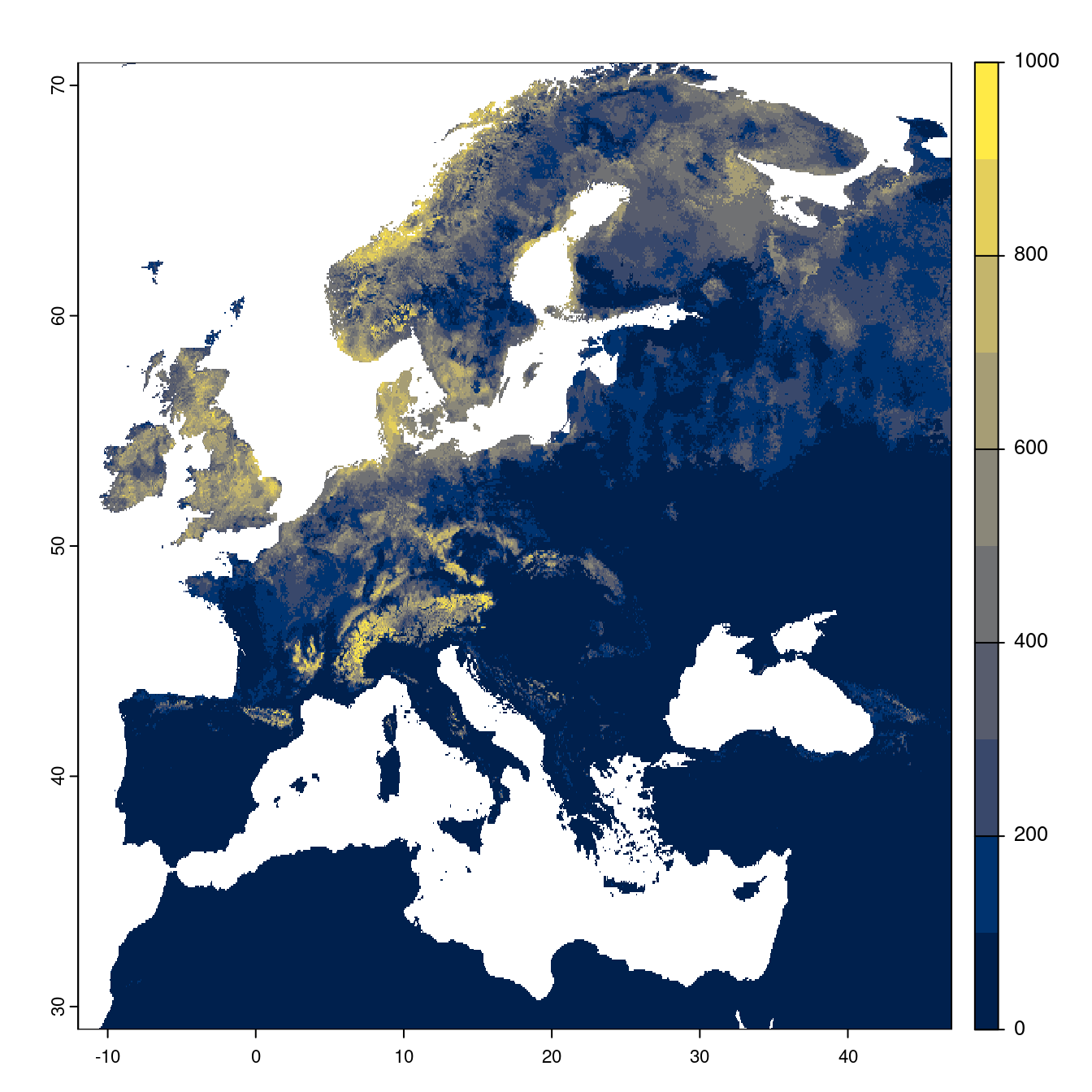
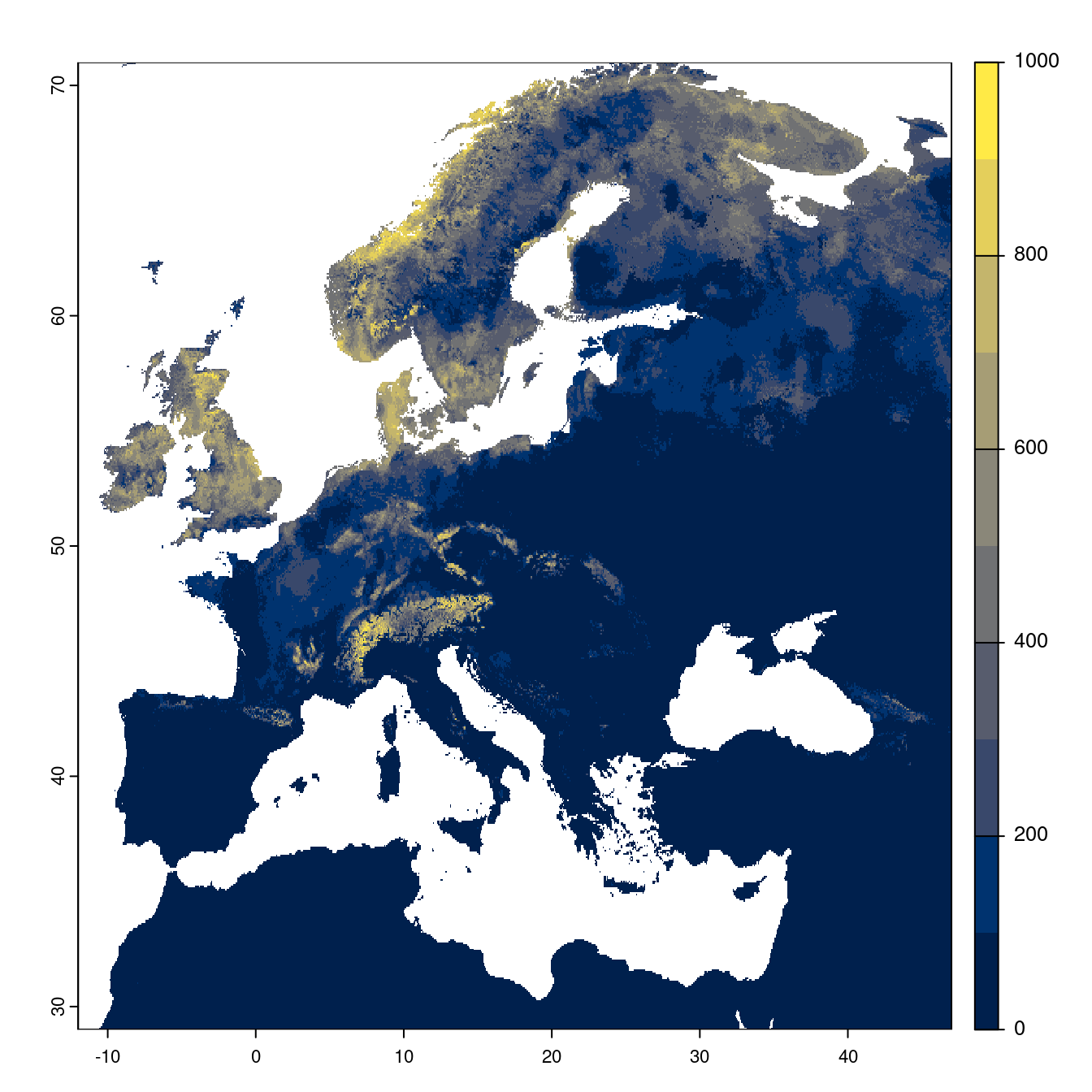
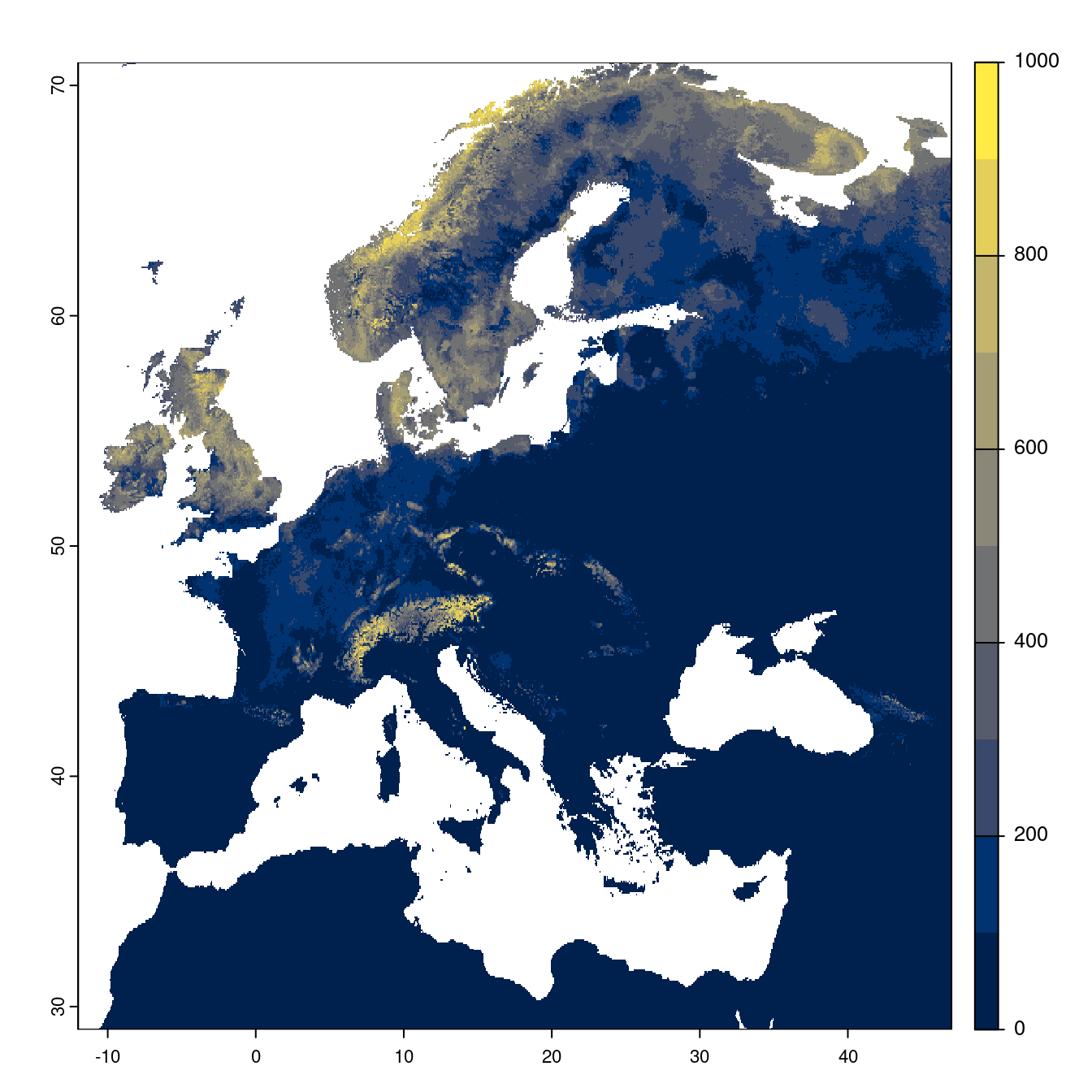
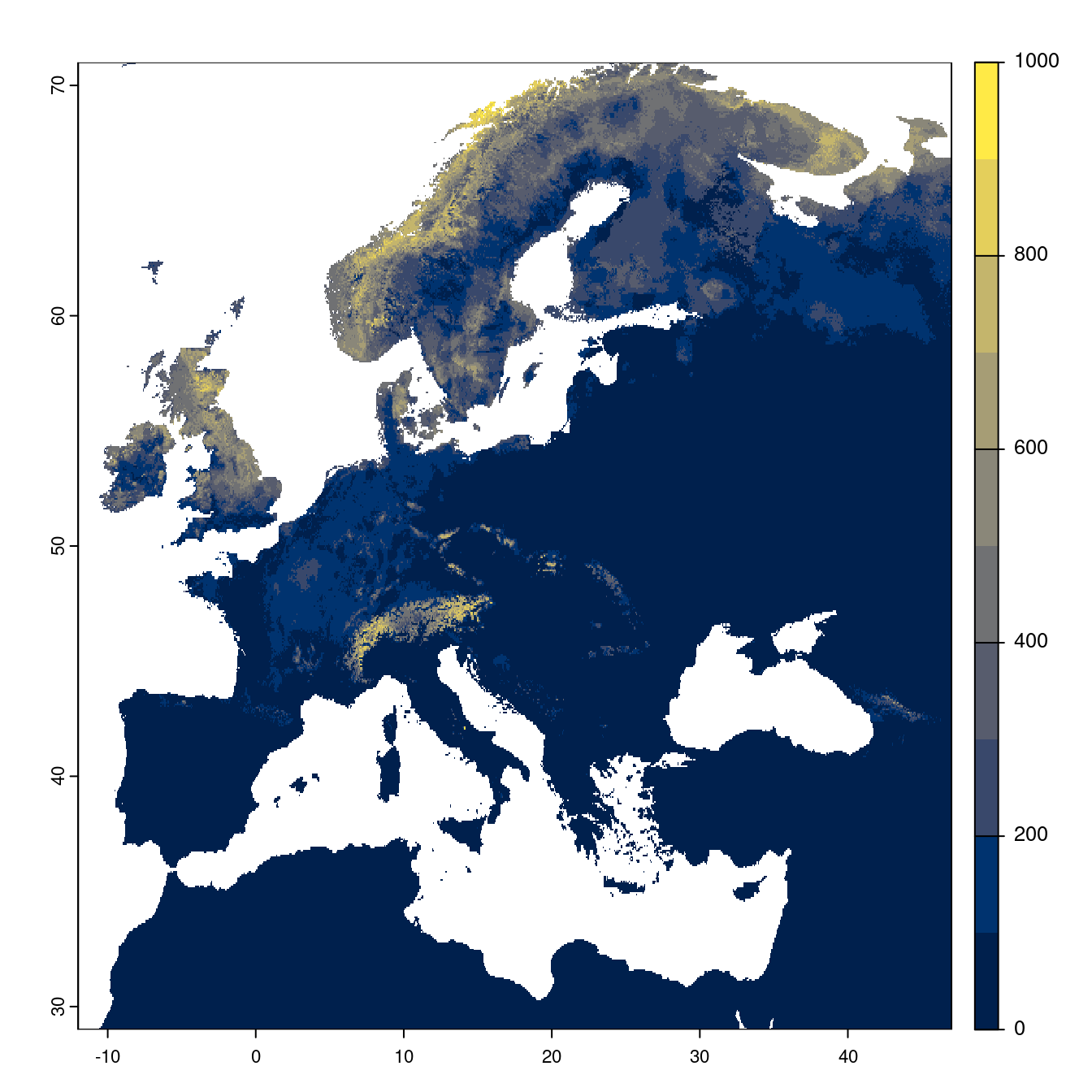
Figure 180.21: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP2-4.5 (modèle d’ensemble).
- SSP3-7.0
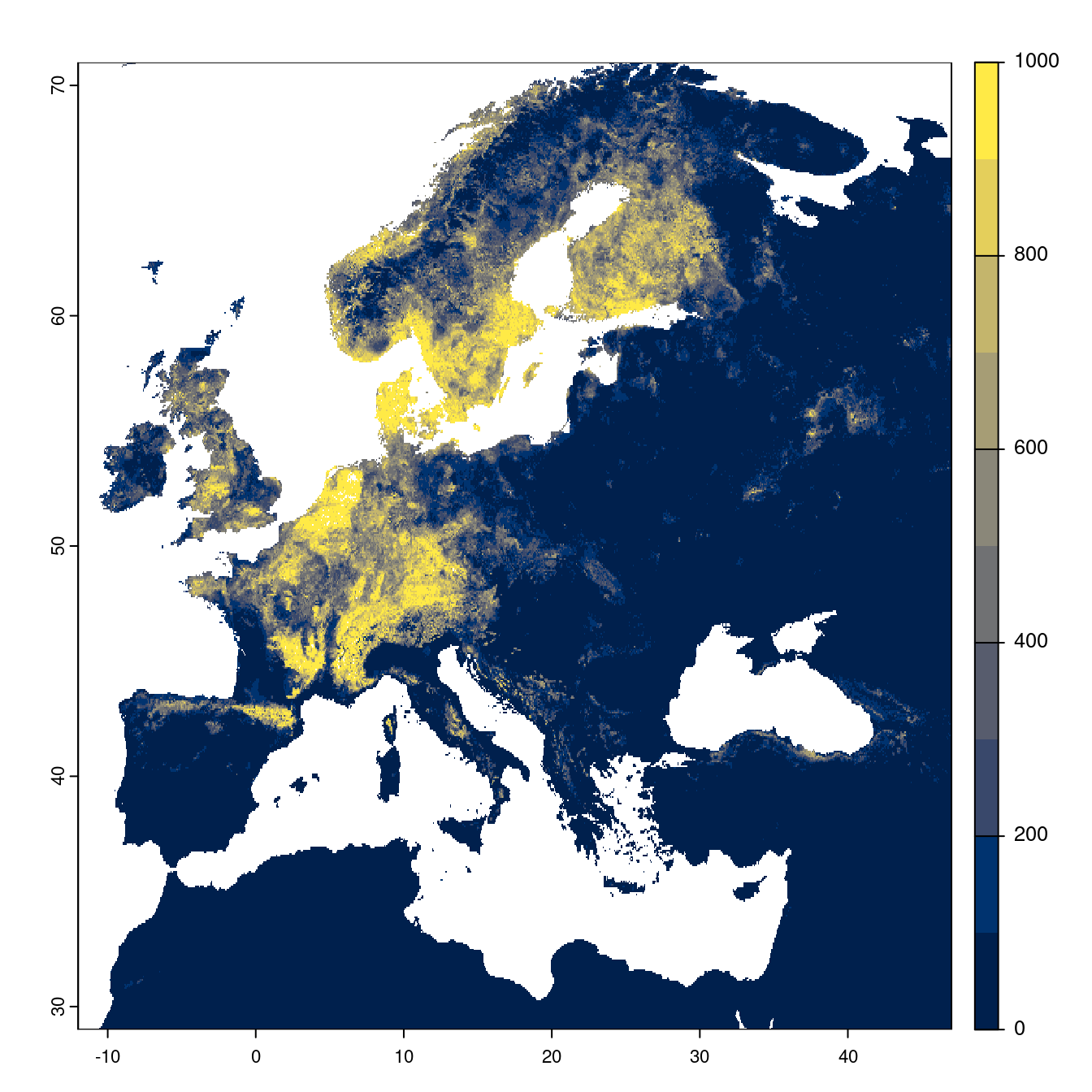
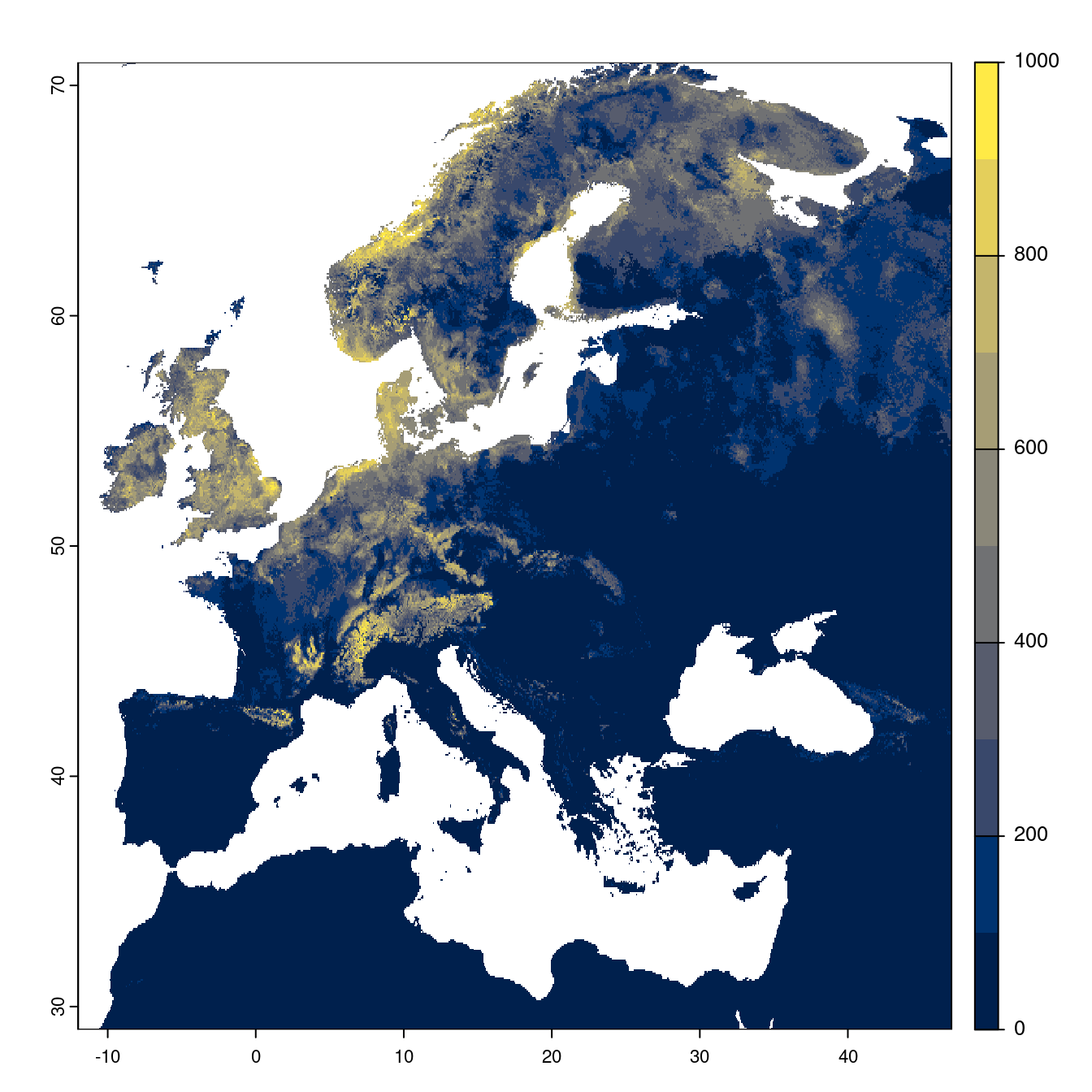
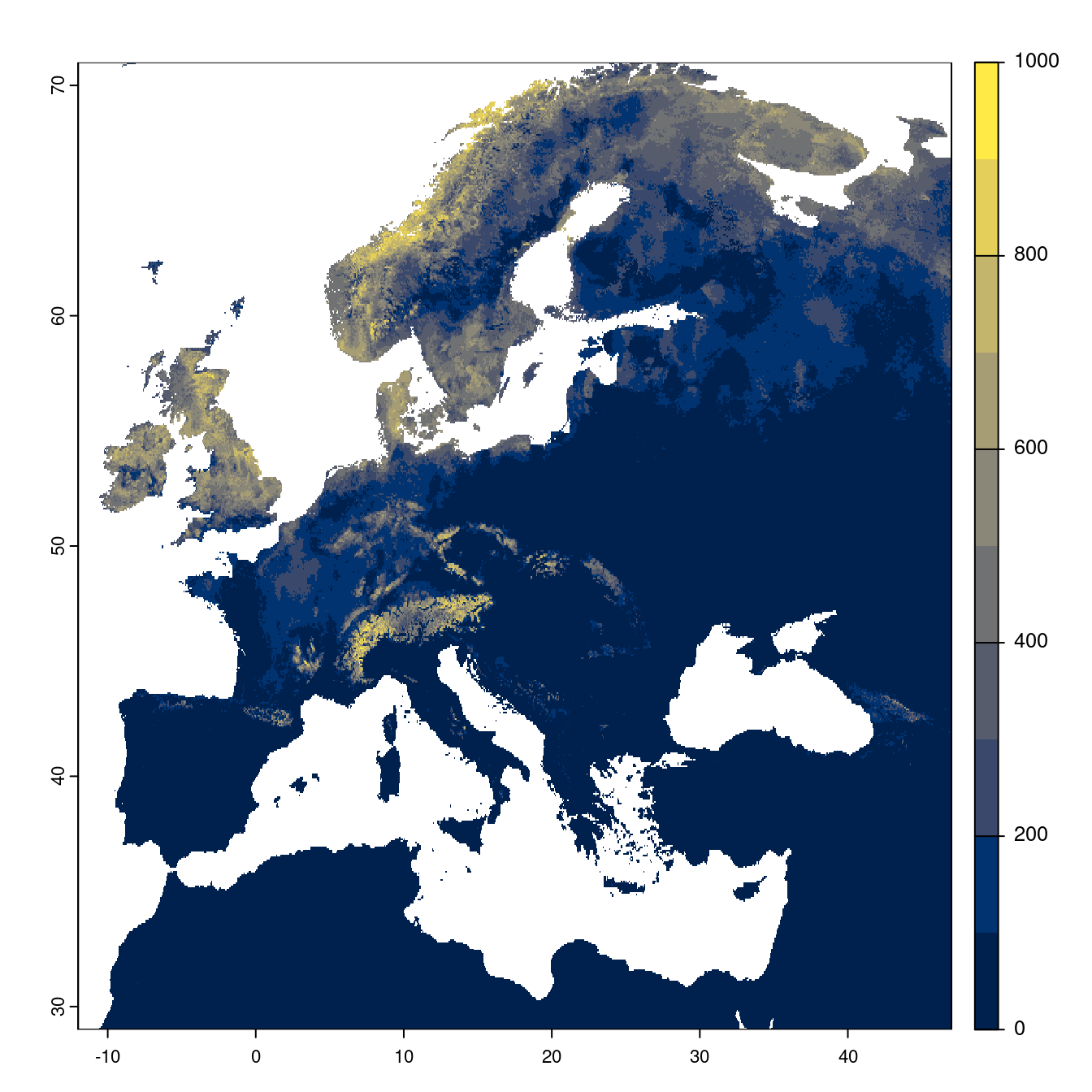
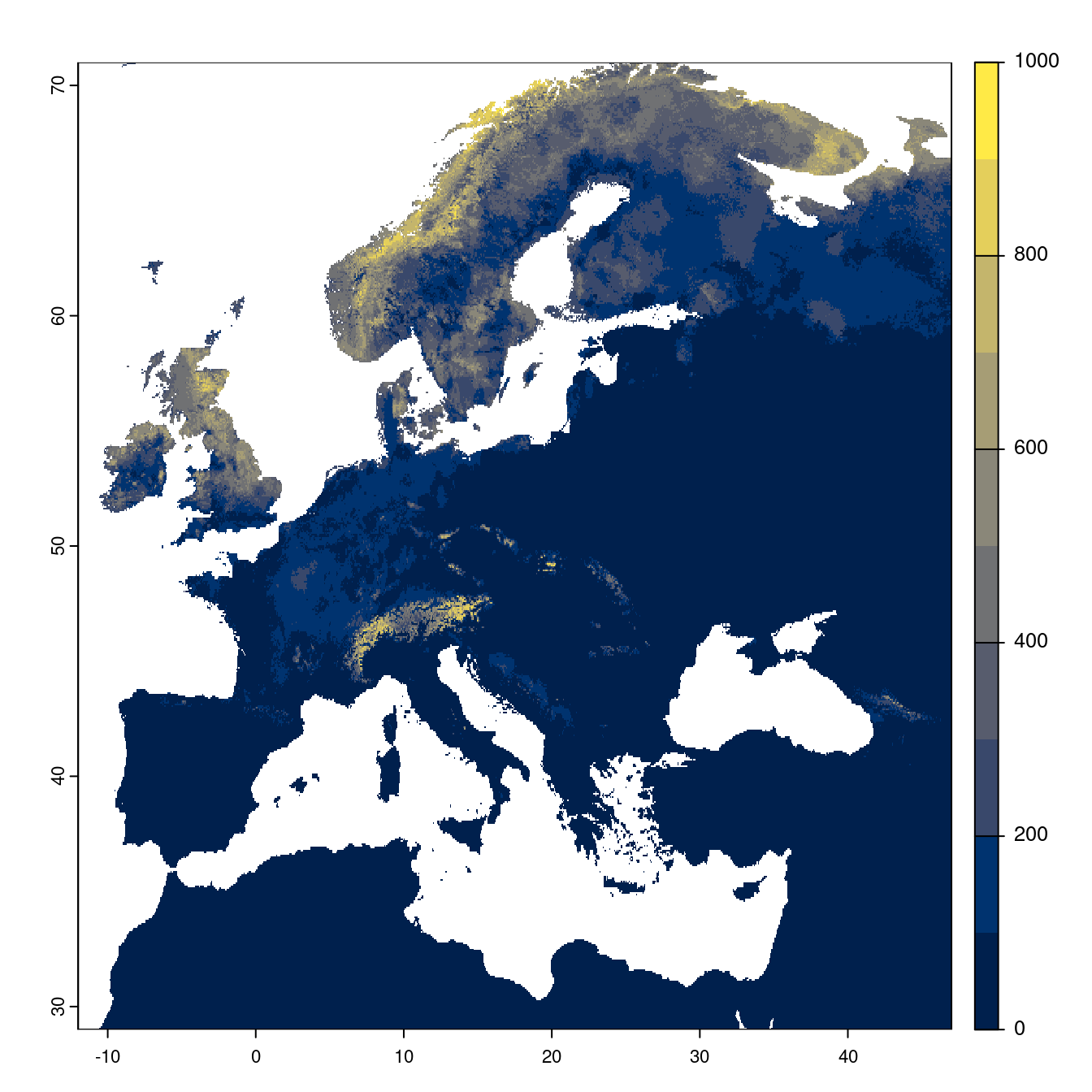
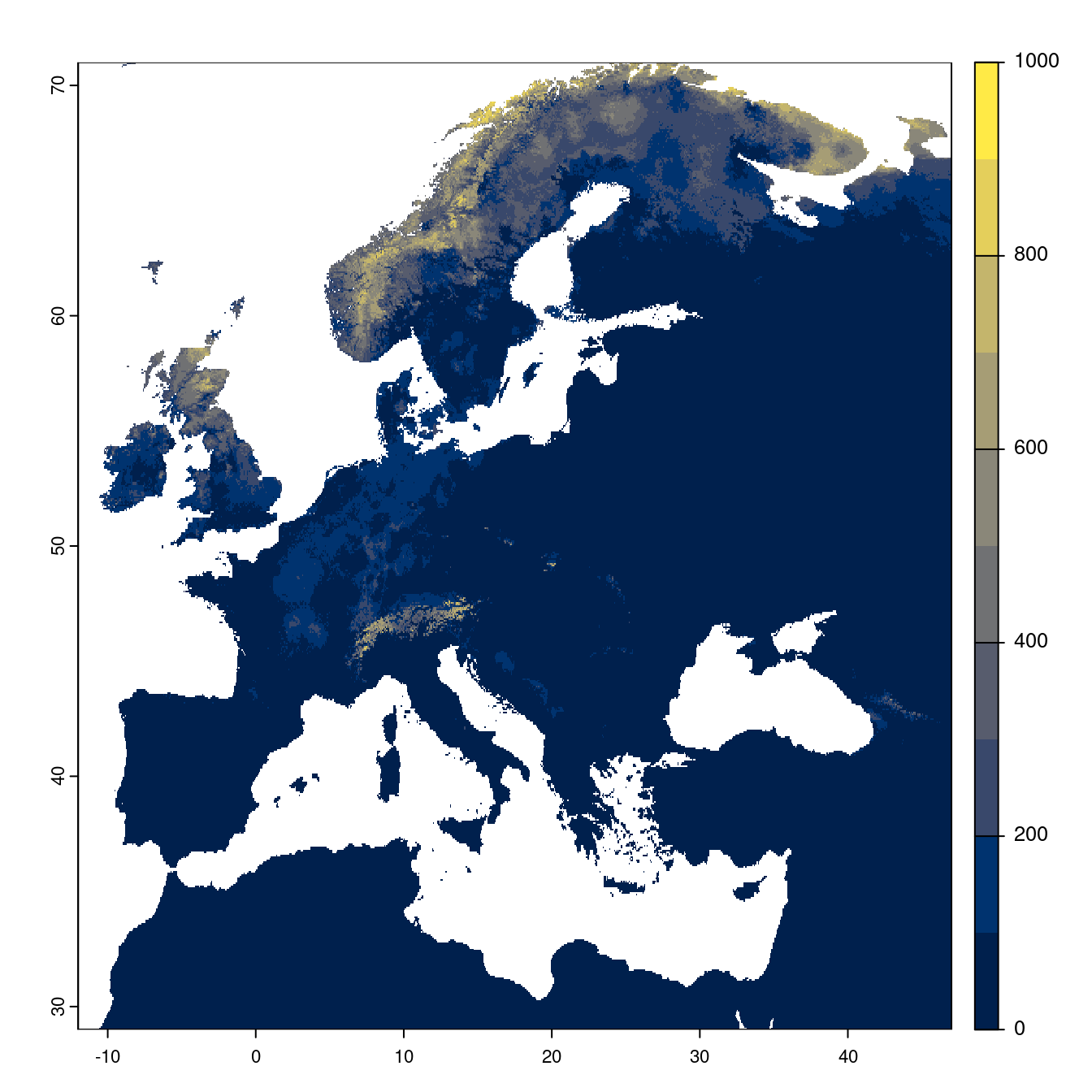
Figure 180.22: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP3-7.0 (modèle d’ensemble).
- SSP5-8.5

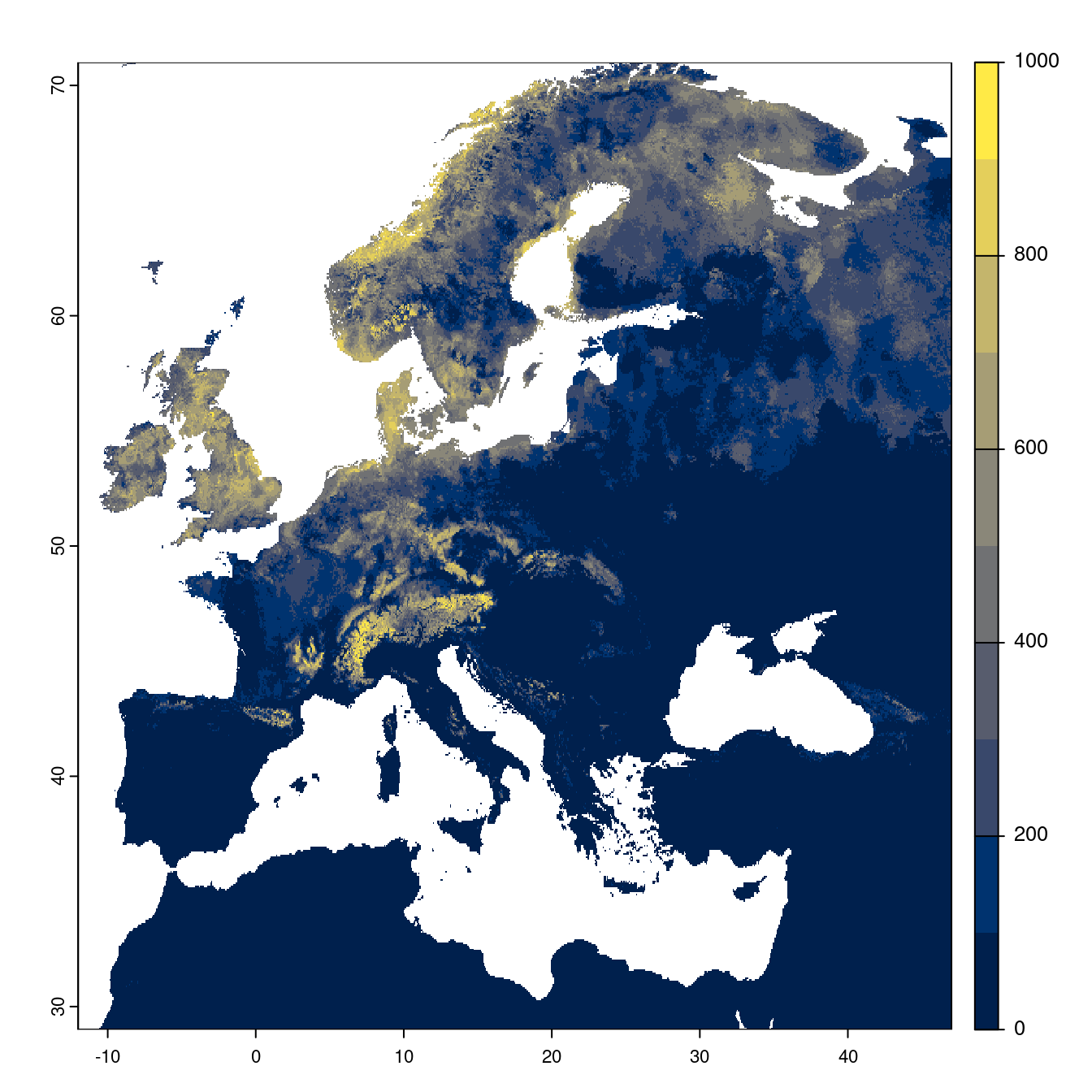

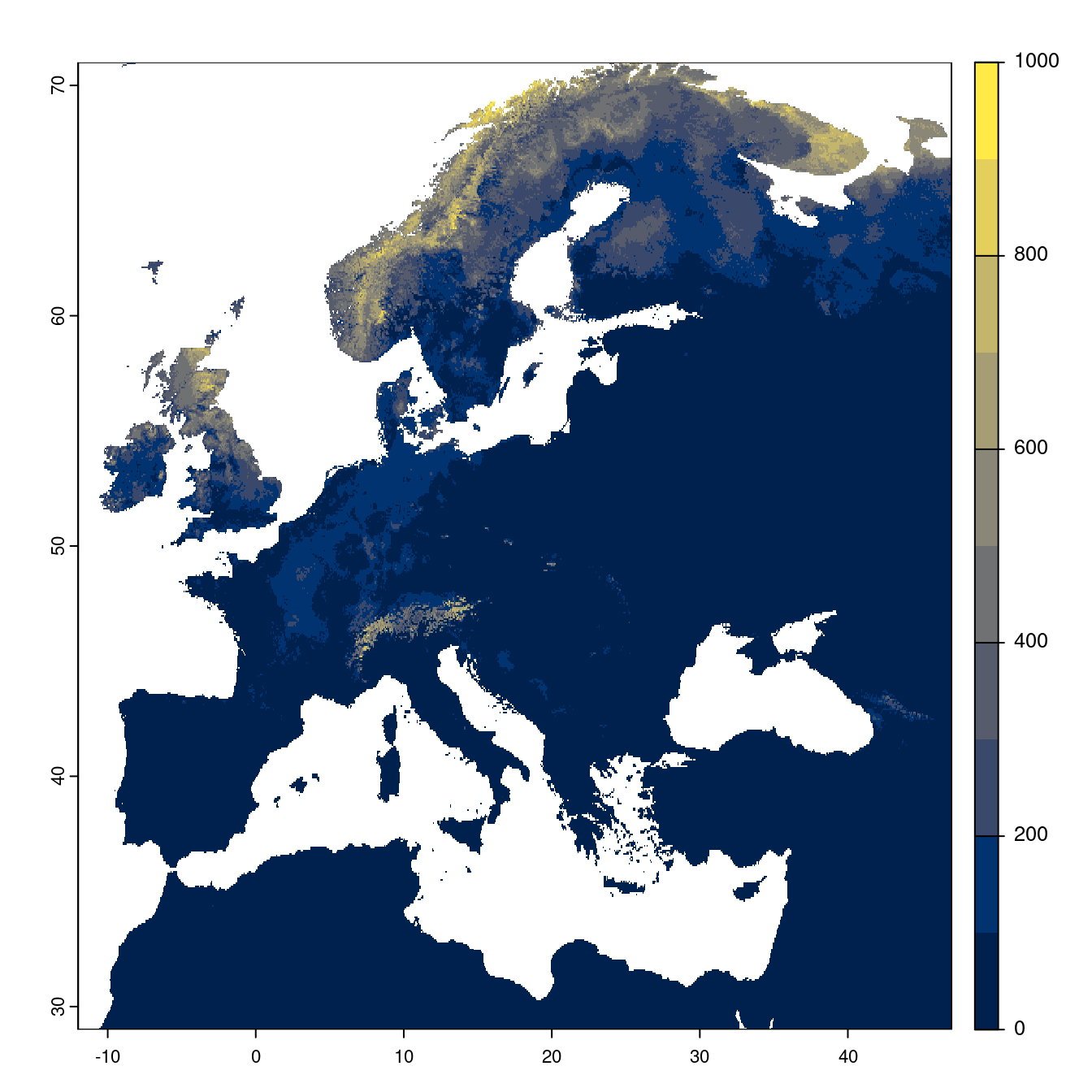
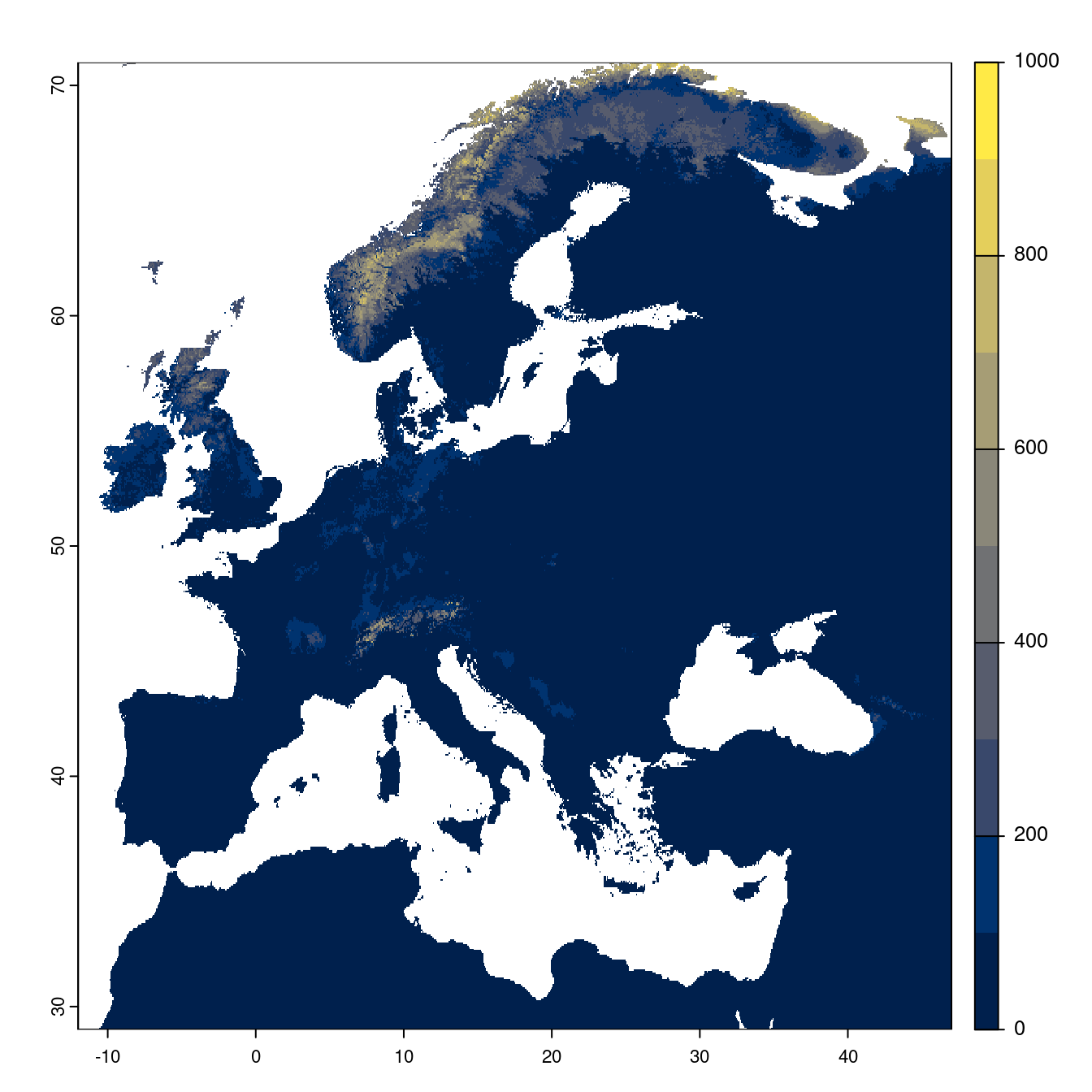
Figure 180.23: Projection des distributions potentielles futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble).
180.4 Inférence à l’échelle de la métropole
- Croisement avec températures de surface 2019 (fournie par la métropole)
- → conditions climatiques favorables à fine échelle
- à peu près 7 pixels × 9 (WorldClim)
180.4.1 Distributions potentielles futures
Même chose que précédemment mais sur les rasters de Grenoble
- SSP1-2.6

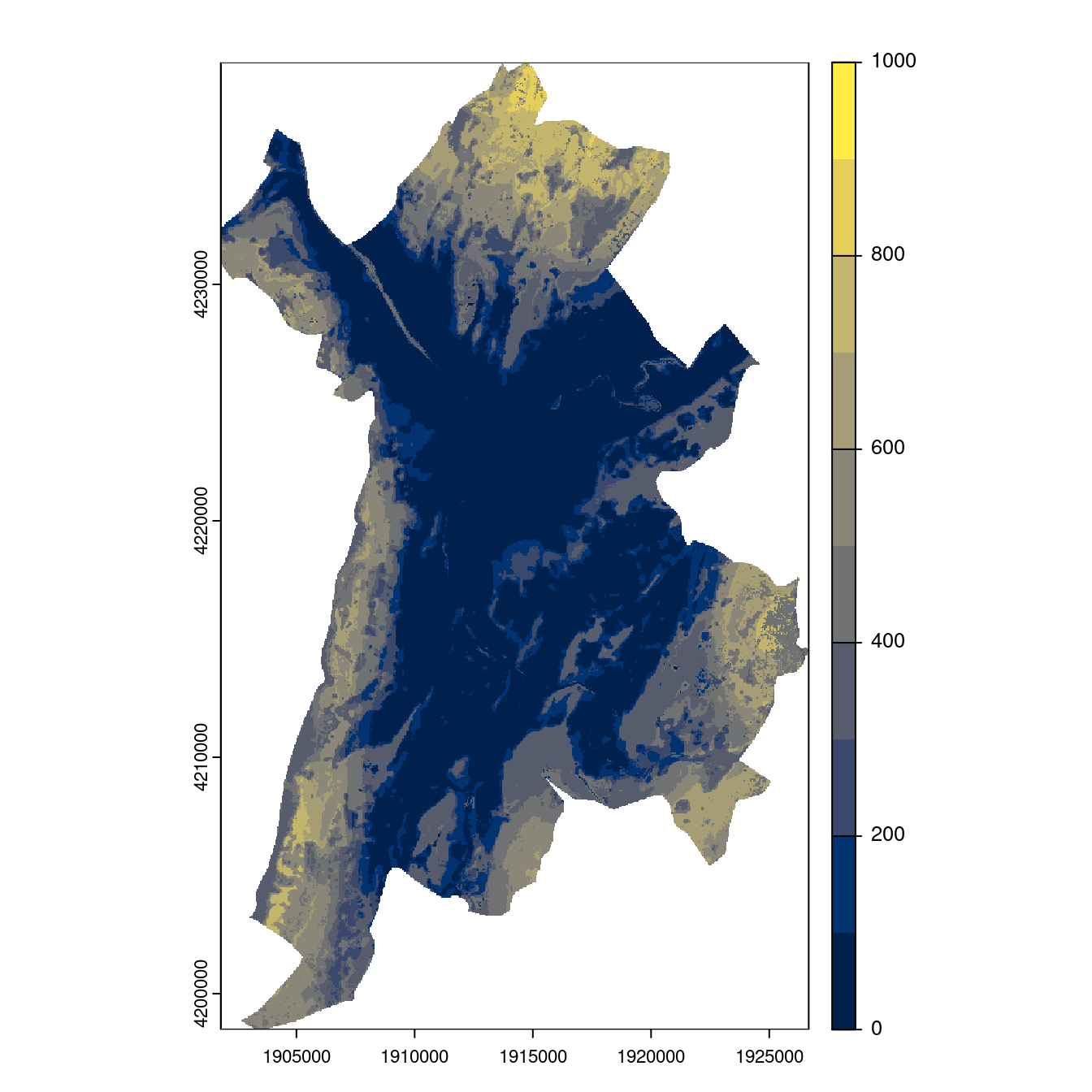
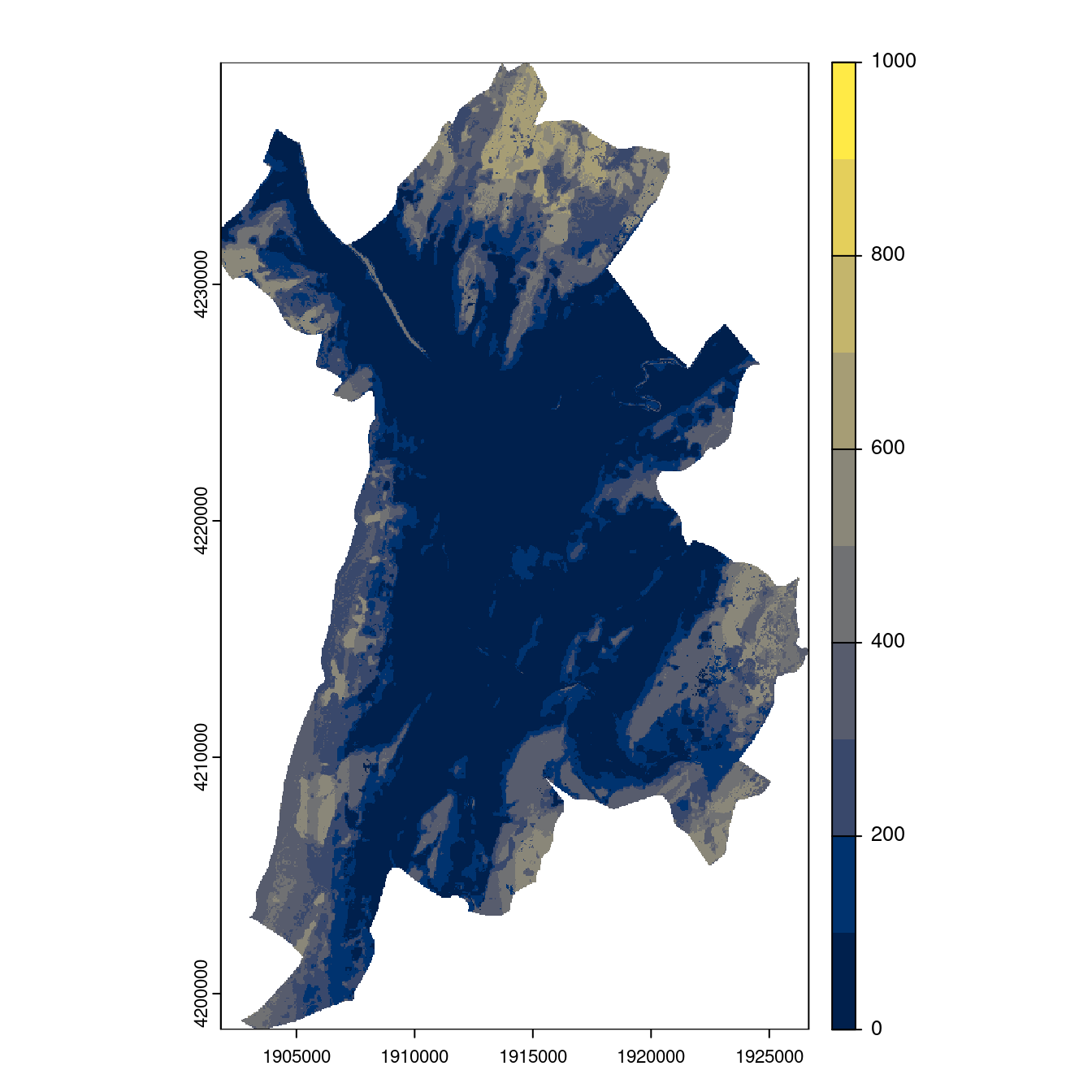
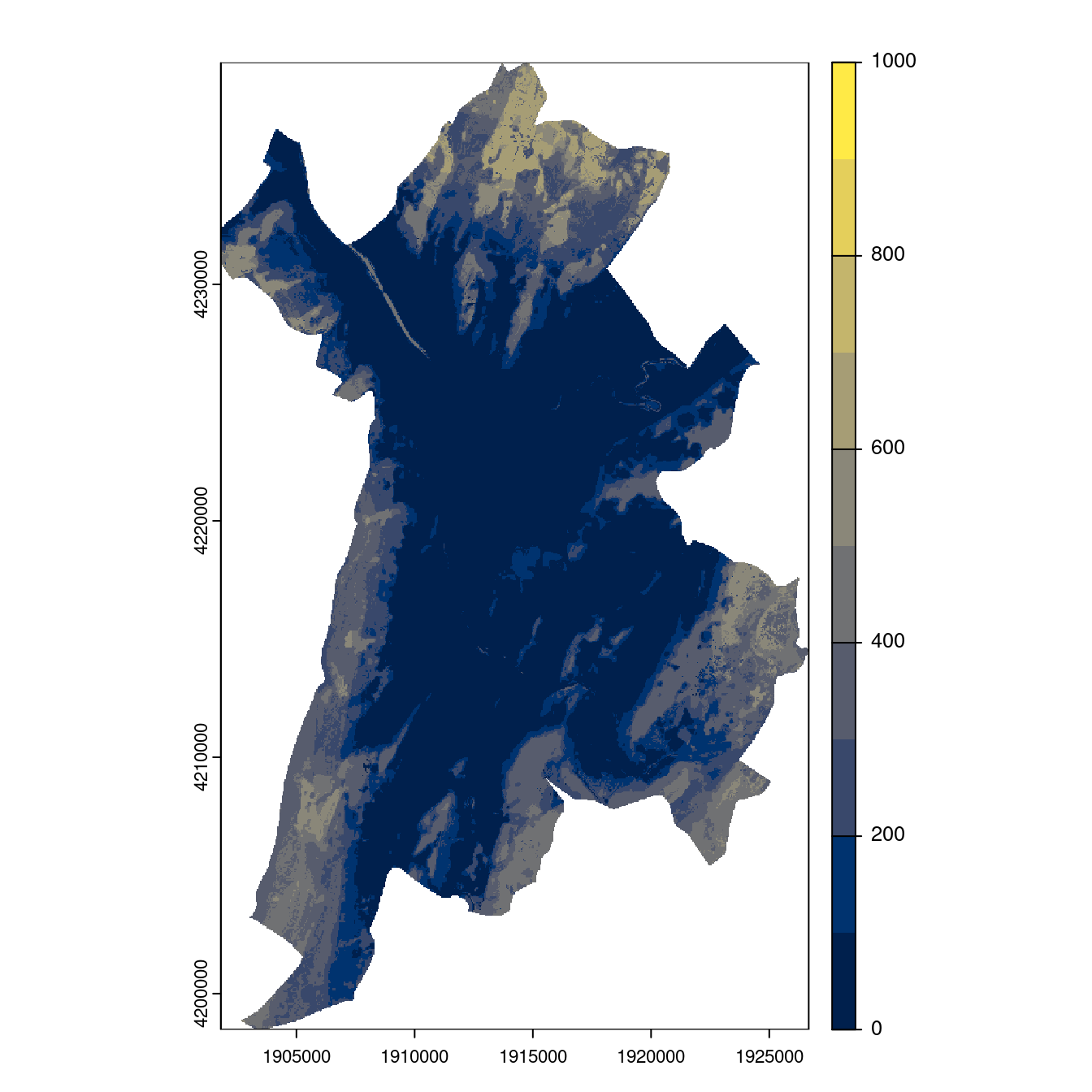
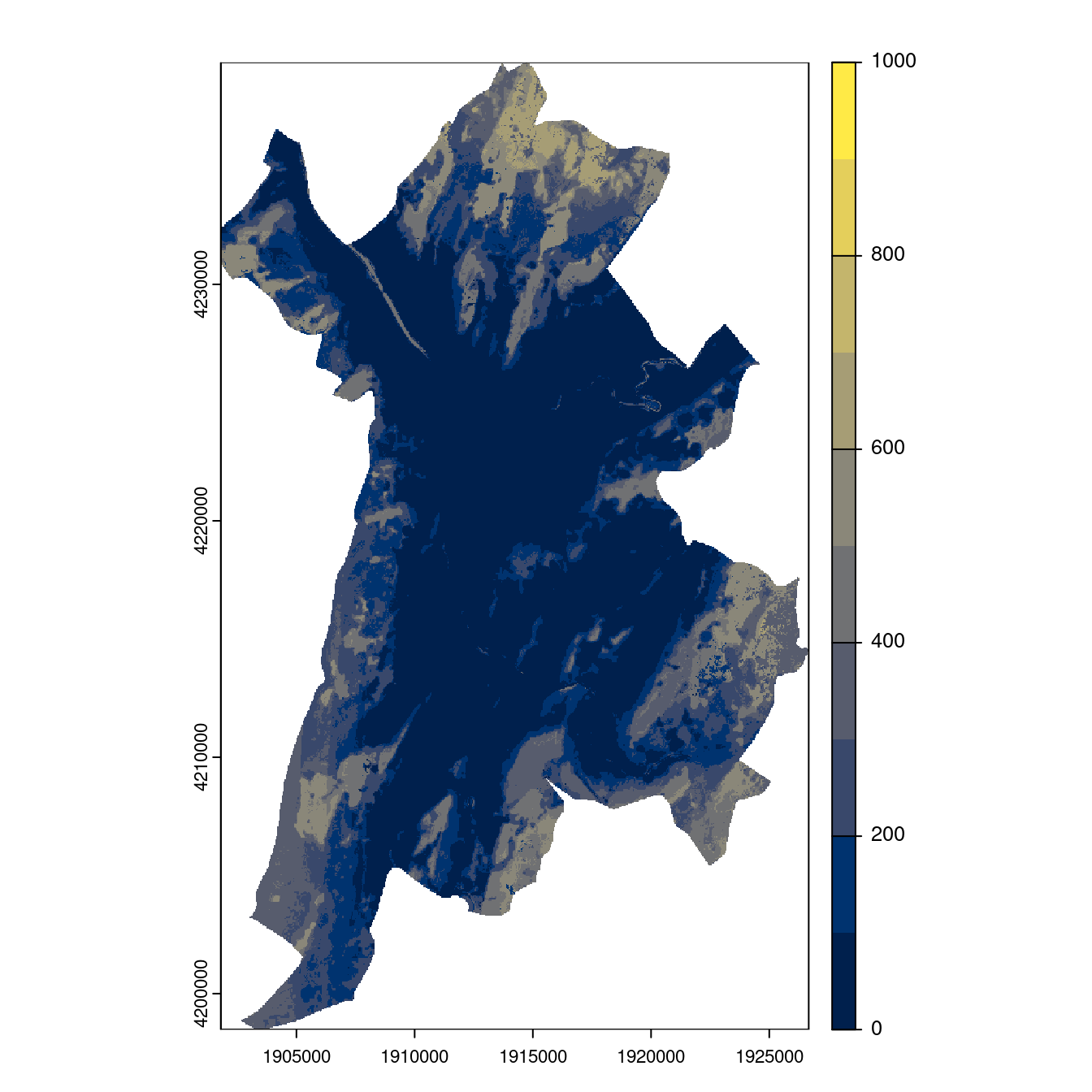
Figure 180.24: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP2-4.5
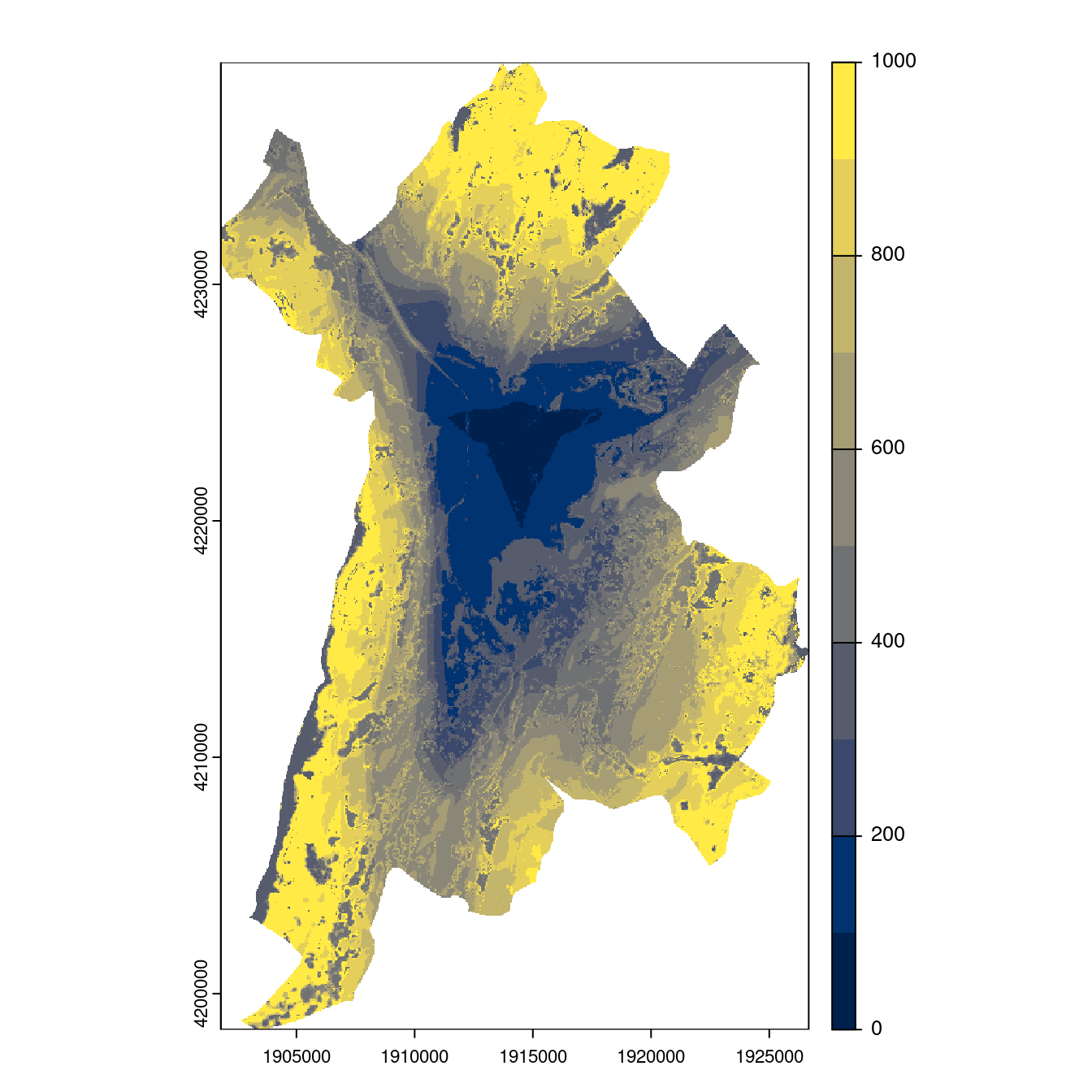
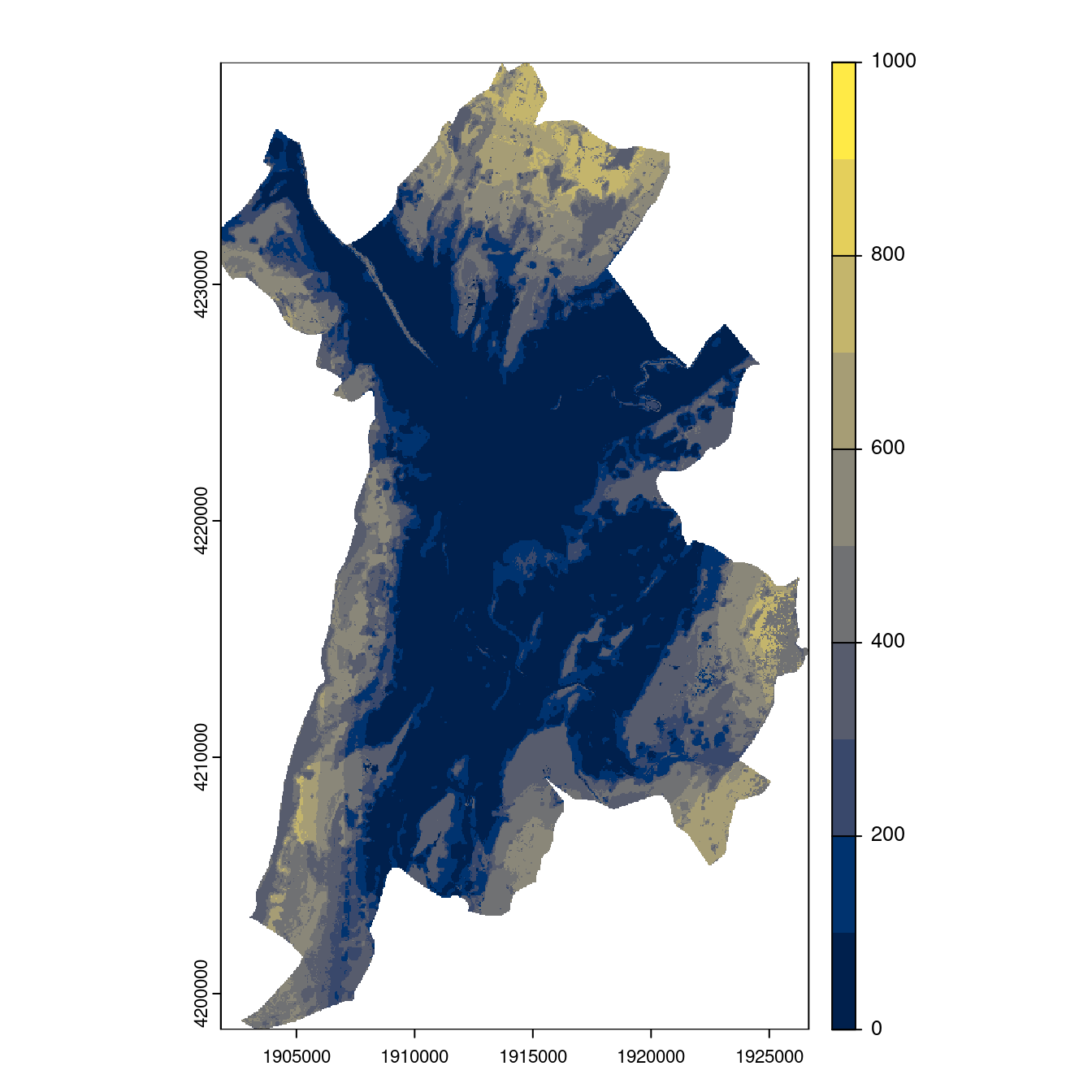
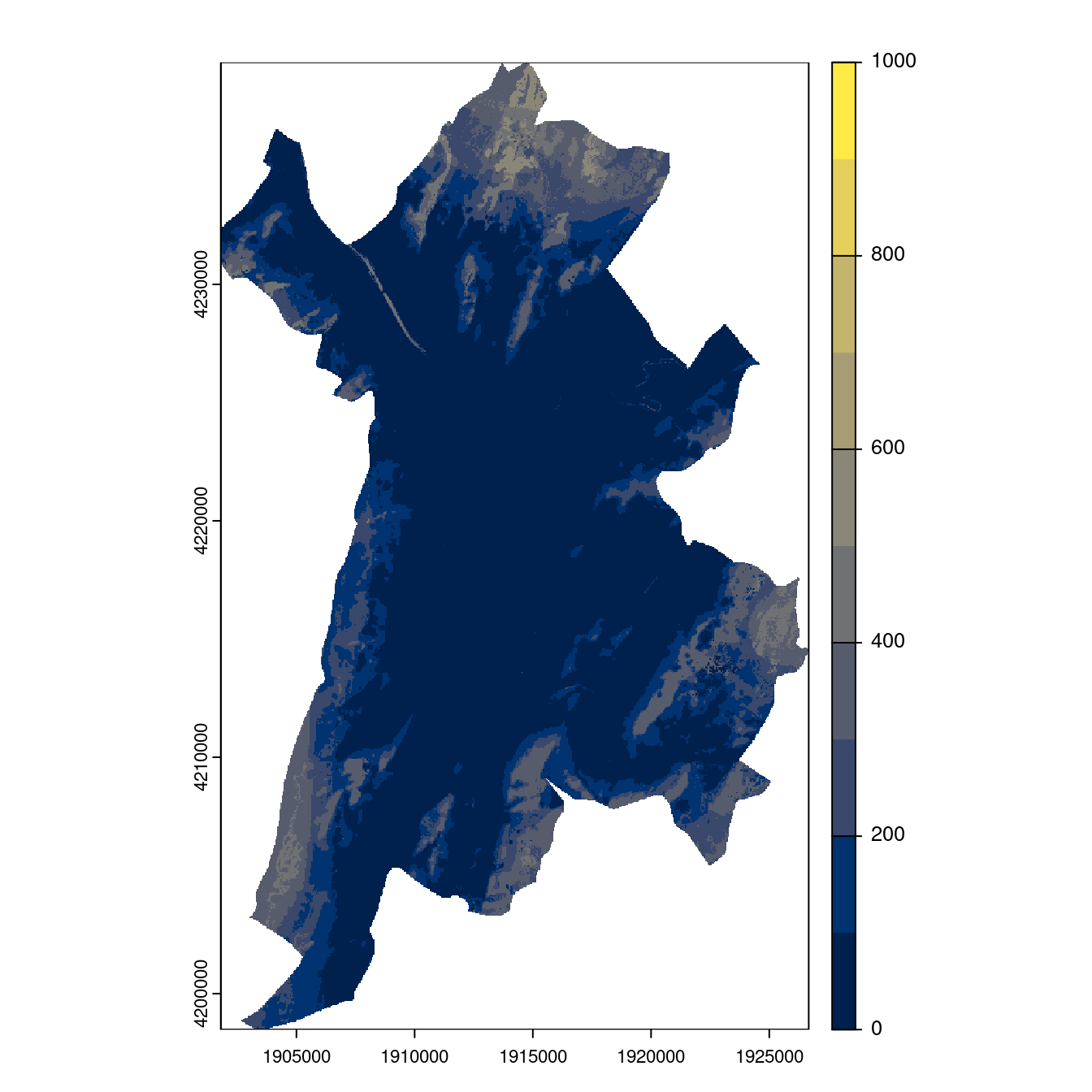
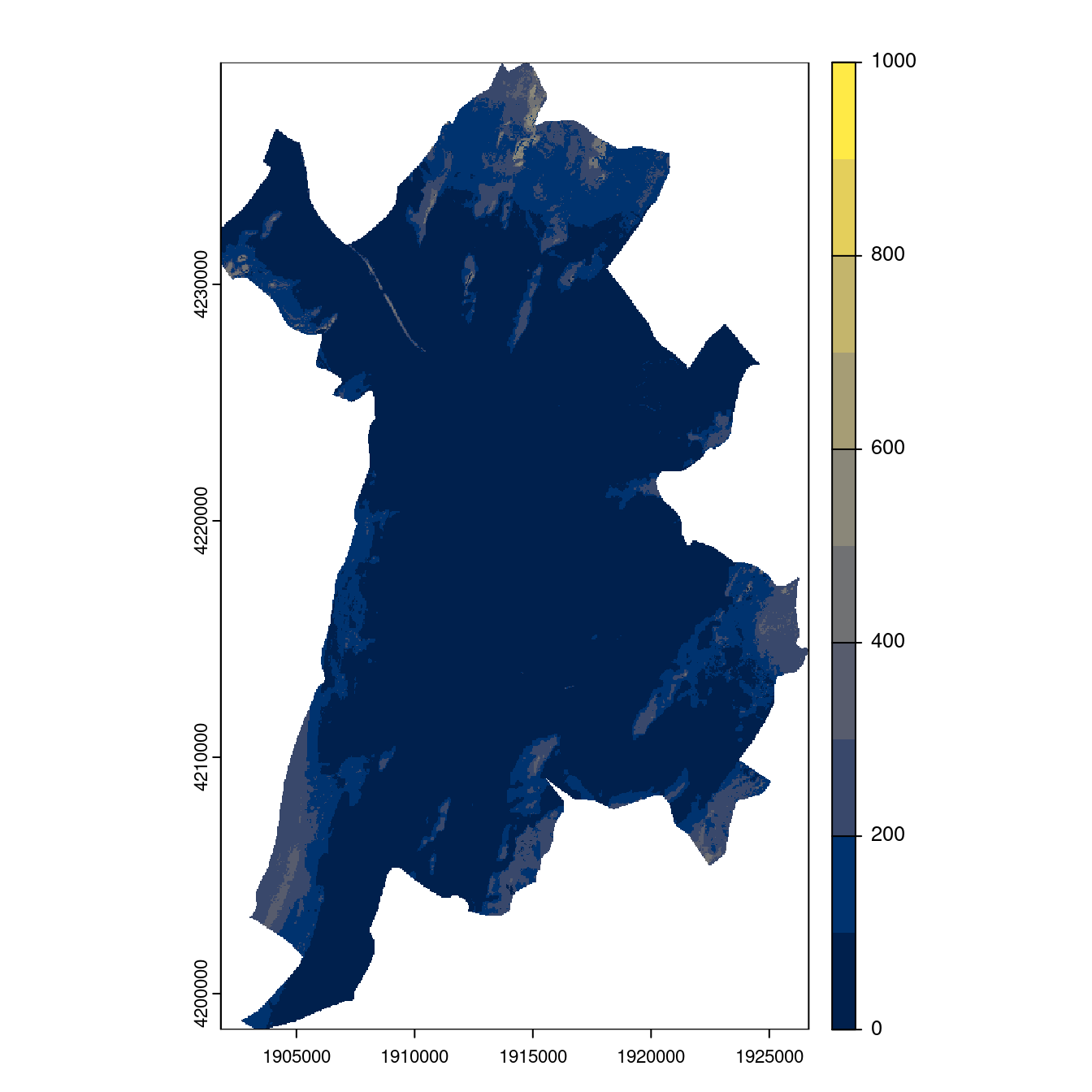
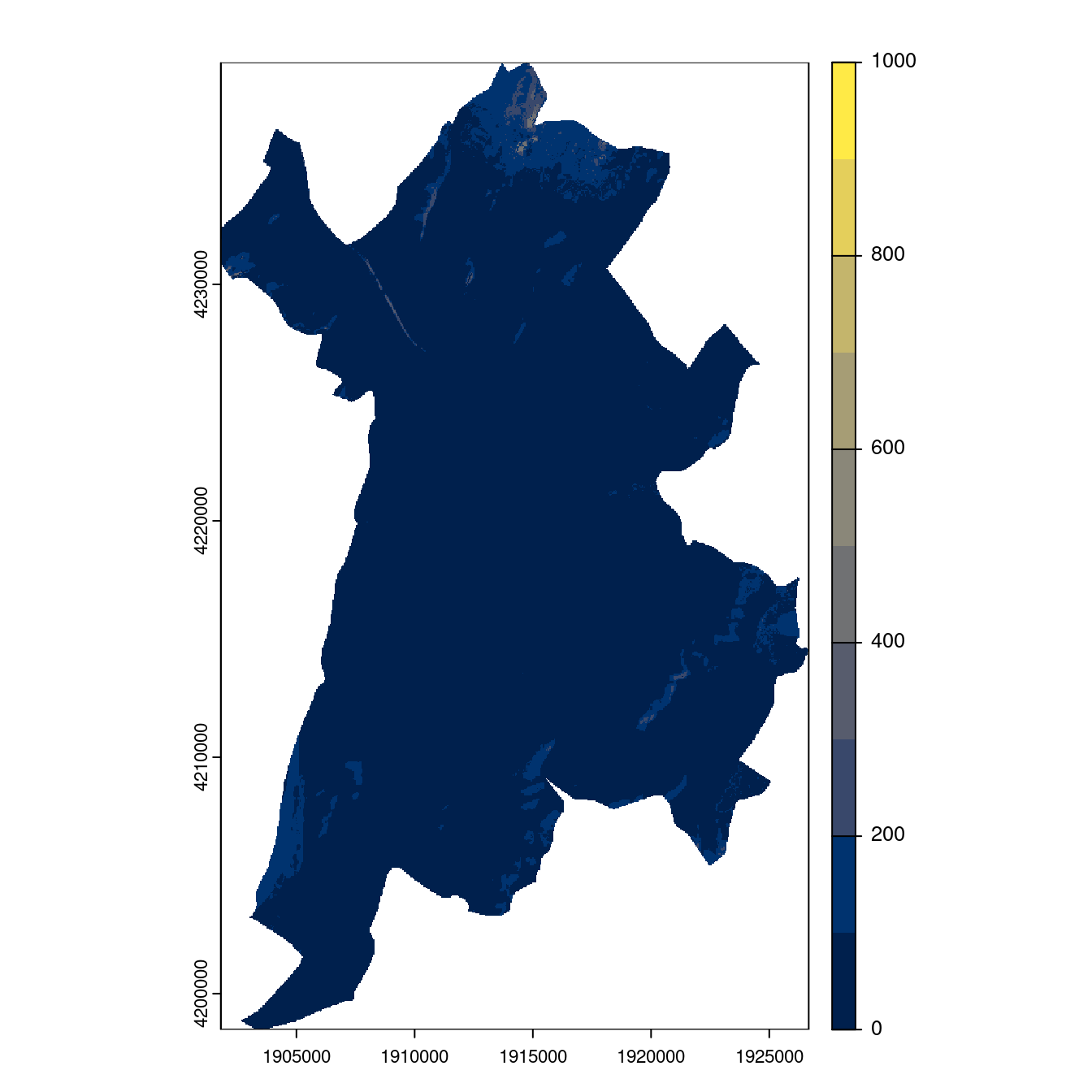
Figure 180.25: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP3-7.0

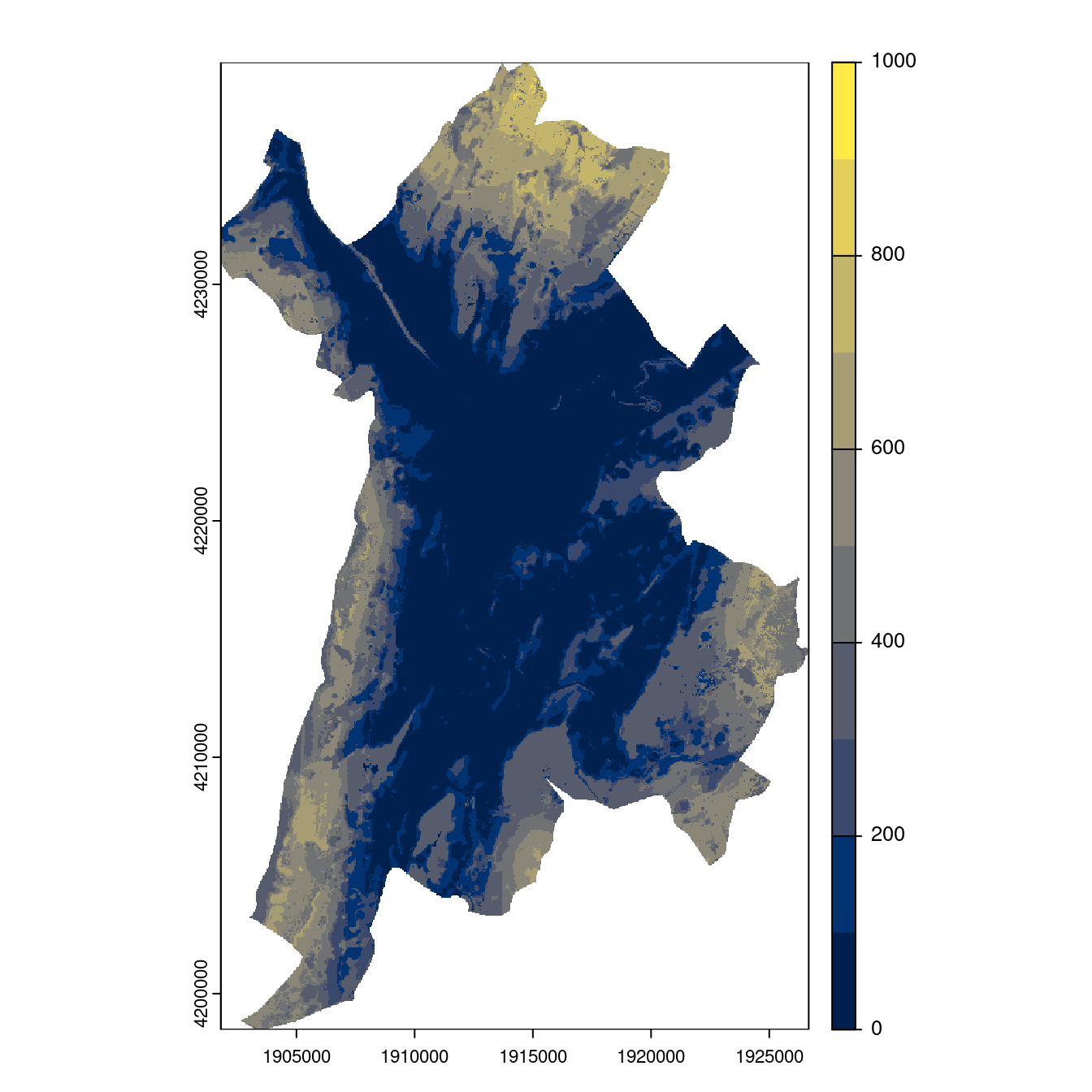

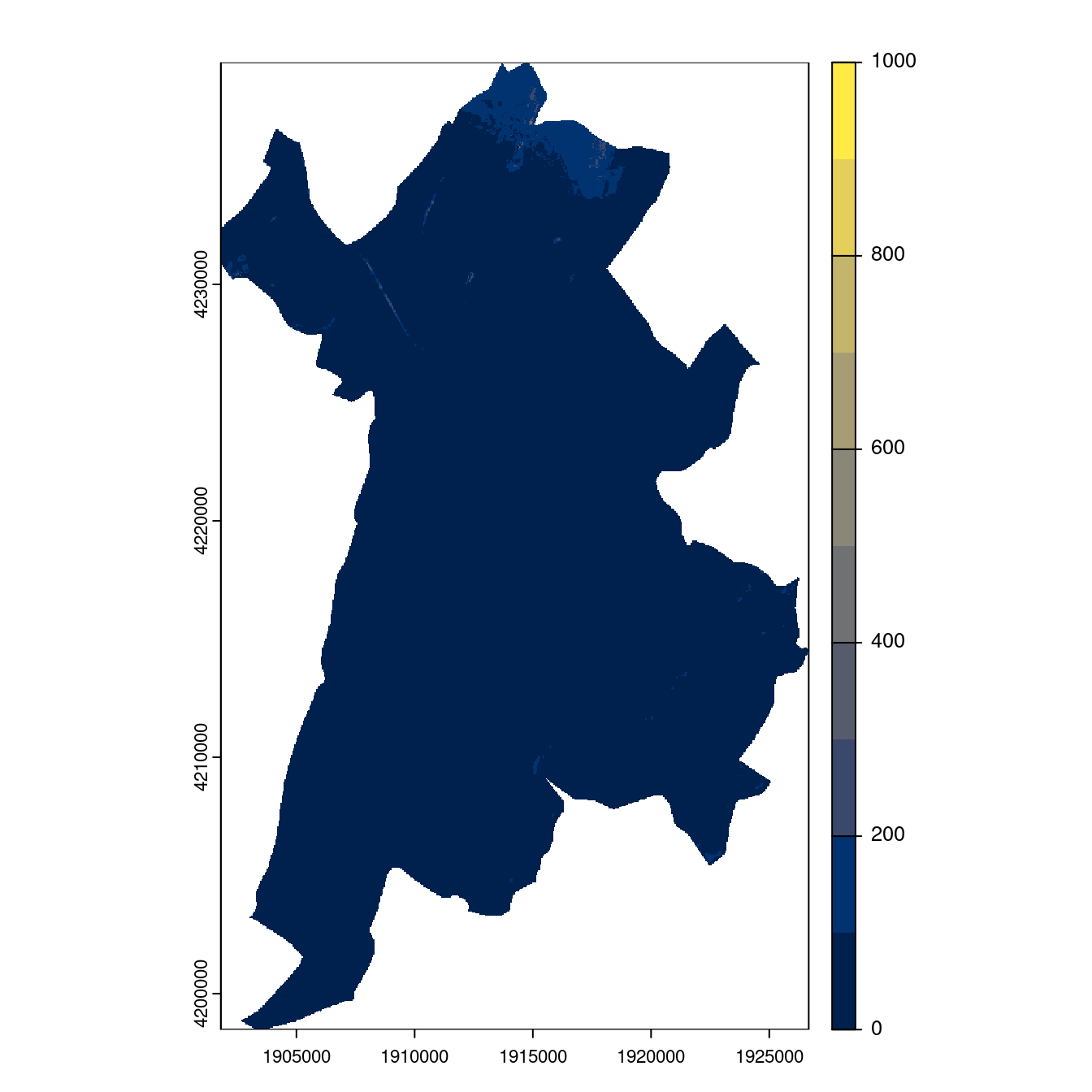
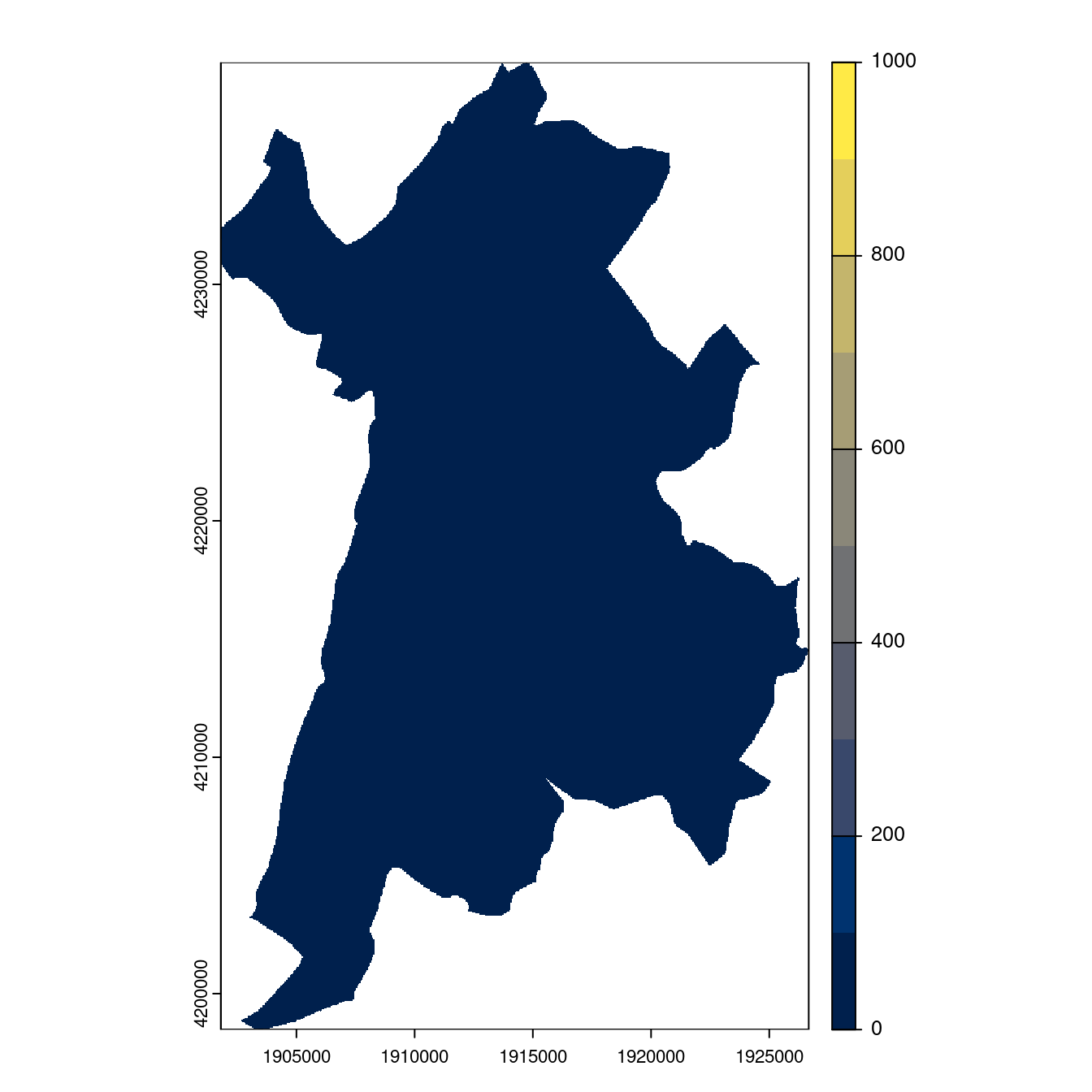
Figure 180.26: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
- SSP5-8.5
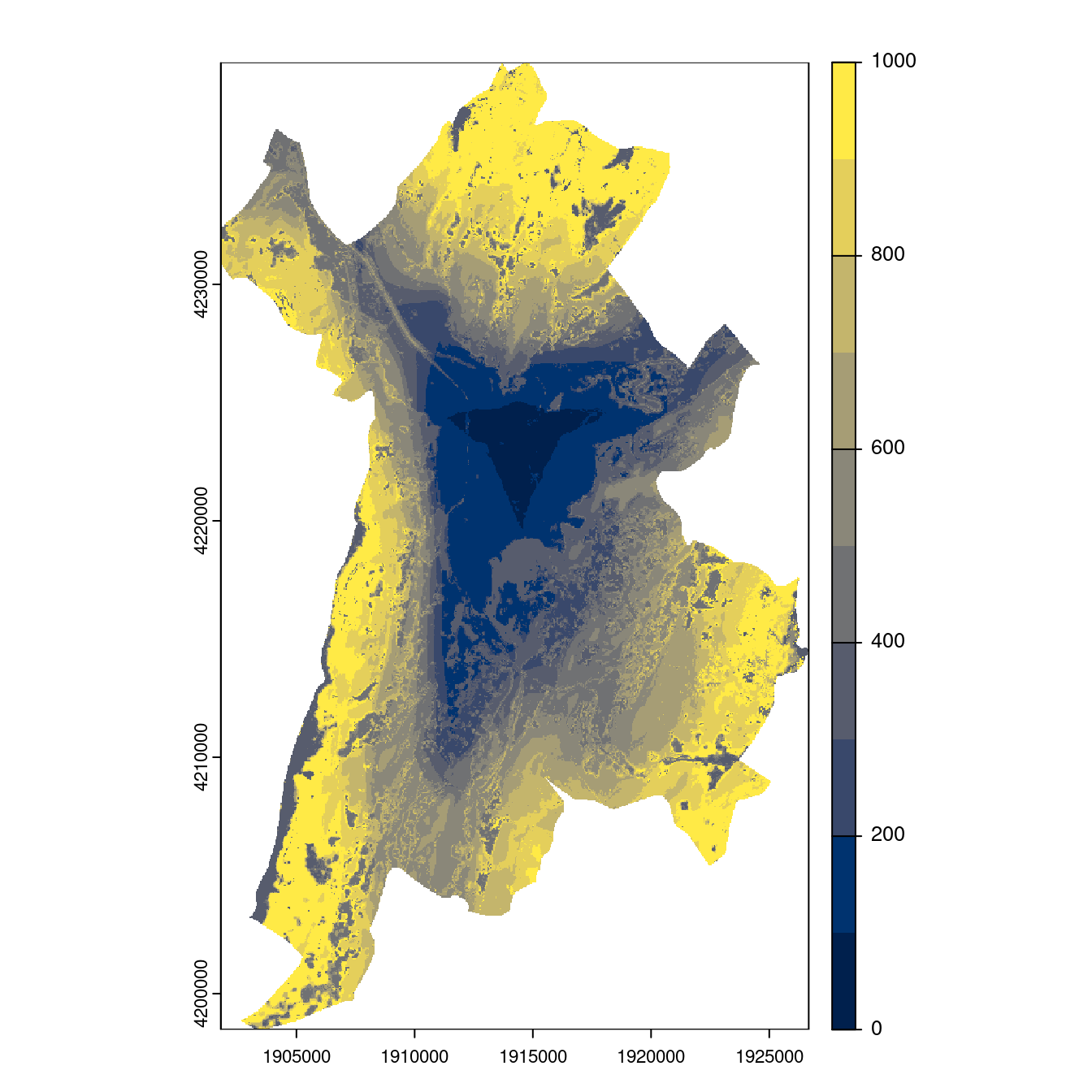
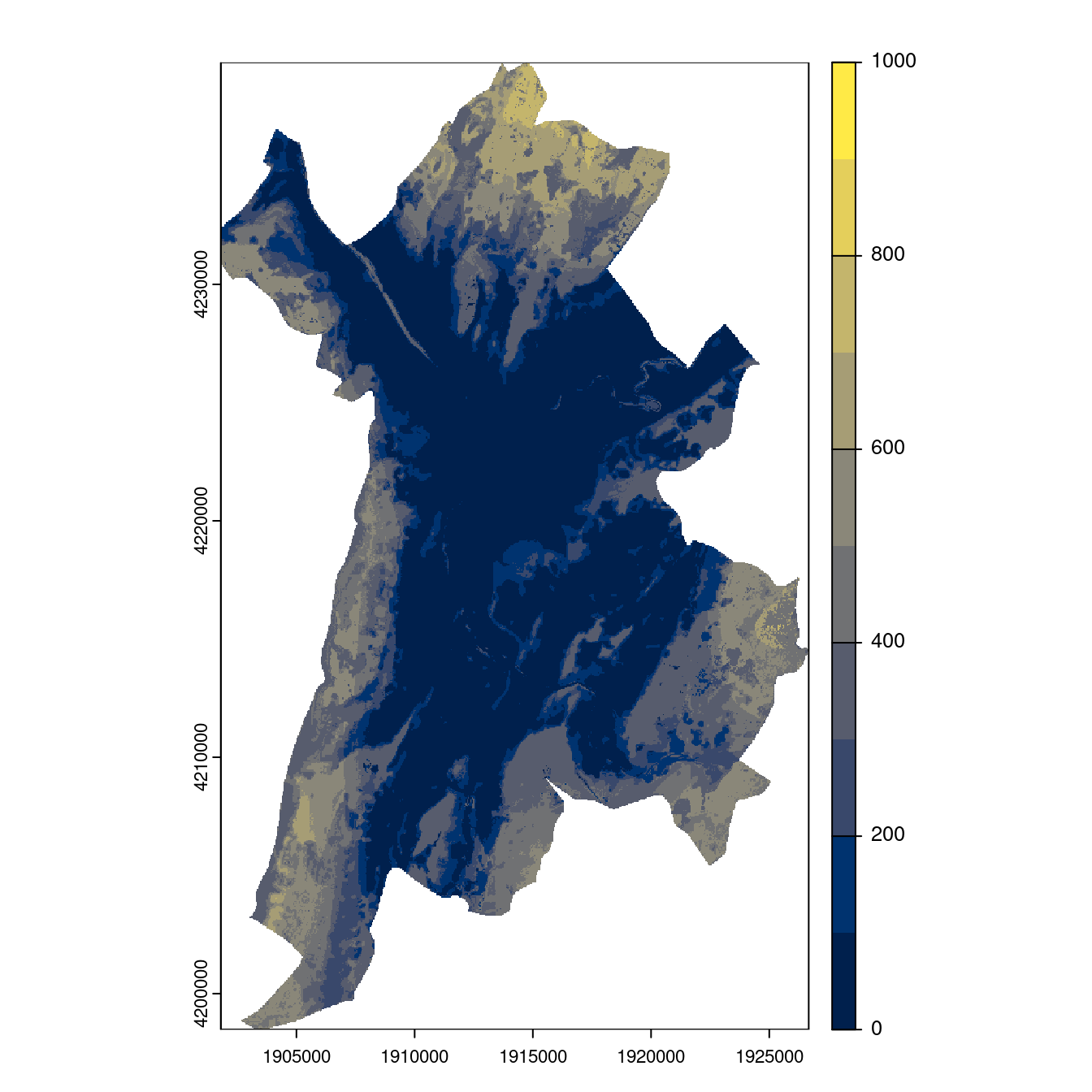
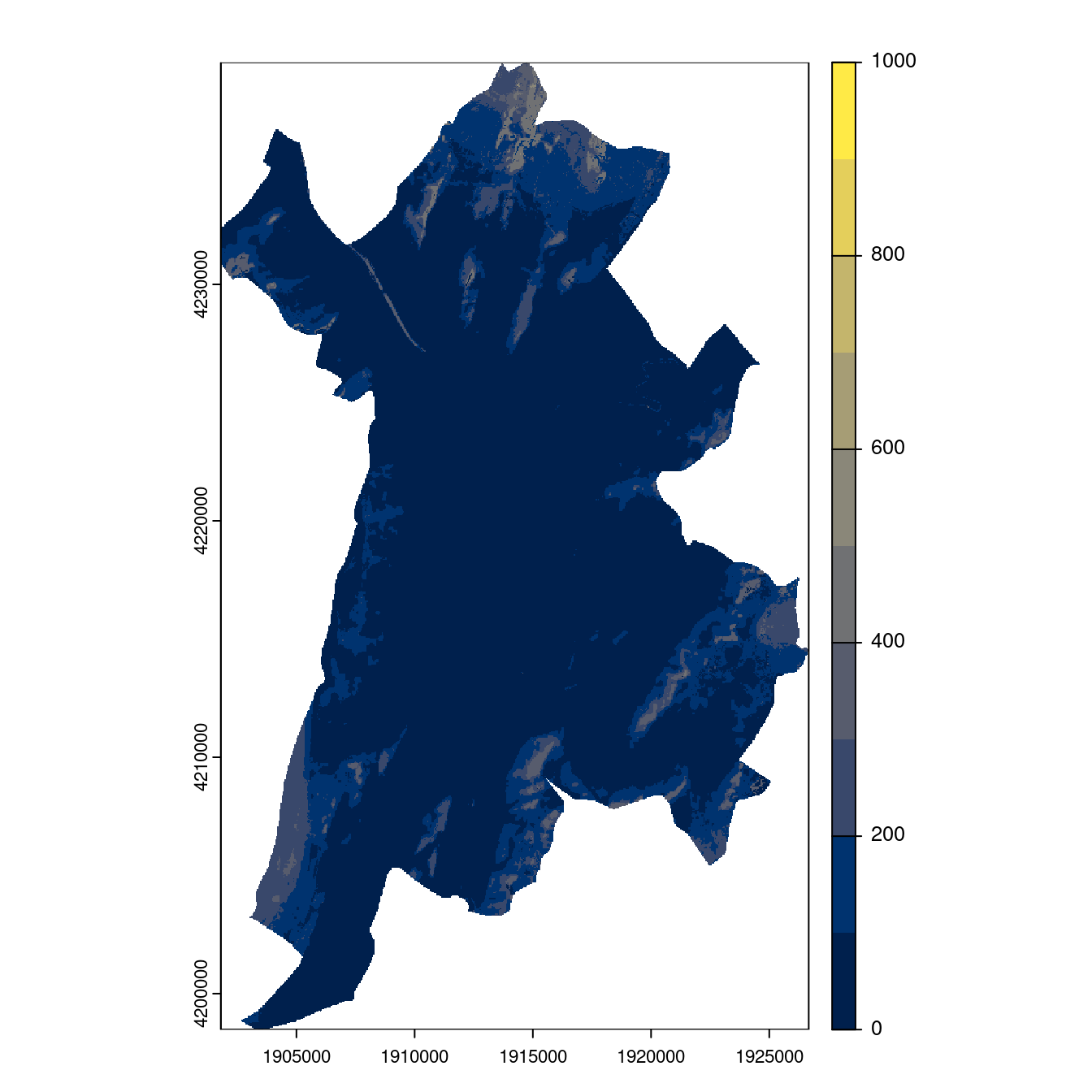
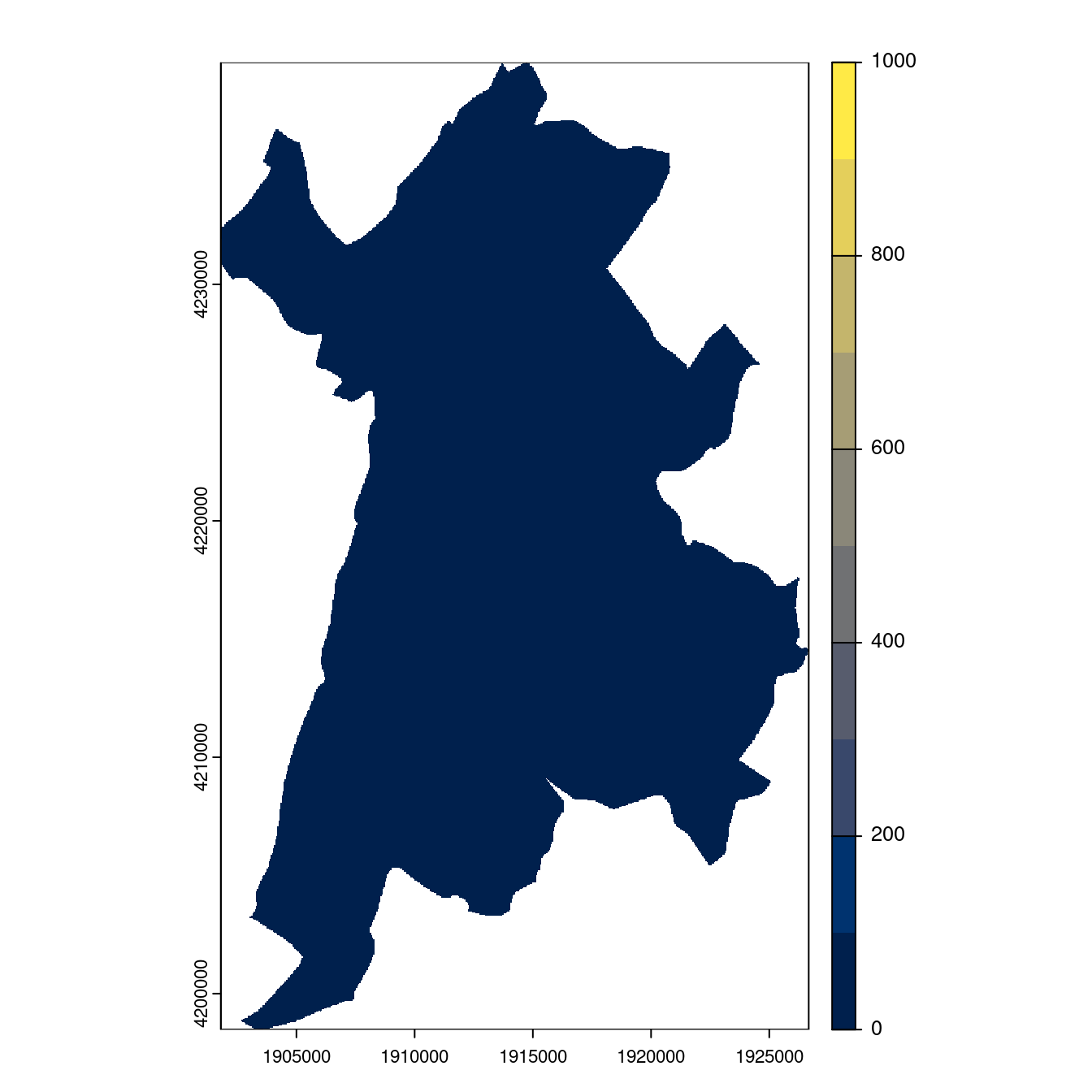
Figure 180.27: Projection des distributions potentielles contemporaine et futures jusqu’à l’horizon 2100 sous le scénario SSP5-8.5 (modèle d’ensemble, GCM IPSL).
180.4.2 Évolution temporelle
# A tibble: 17 × 8
min q1 median mean q3 max ssp year
<table[1d]> <table[1d]> <table[1d]> <table[1d]> <table[1d]> <tab> <fct> <dbl>
1 62 381 575 581.32679 833 1000 0 2000
2 6 29 202 258.94876 437 852 126 2040
3 5 21 91 171.60931 305 684 126 2060
4 6 23 88 172.60422 314 700 126 2080
5 8 21 107 171.93897 279 684 126 2100
6 3 27 168 234.01824 407 795 245 2040
7 5 18 39 112.78799 184 561 245 2060
8 8 21 28 69.06615 91 548 245 2080
9 3 25 34 43.20723 47 437 245 2100
10 3 30 160 235.42489 403 811 370 2040
11 7 22 42 111.75877 182 614 370 2060
12 6 29 40 42.27561 49 493 370 2080
13 3 22 30 29.67585 39 73 370 2100
14 3 25 172 224.49796 387 794 585 2040
15 5 19 26 67.35760 91 513 585 2060
16 0 15 30 27.04842 38 84 585 2080
17 13 24 27 27.46554 31 79 585 2100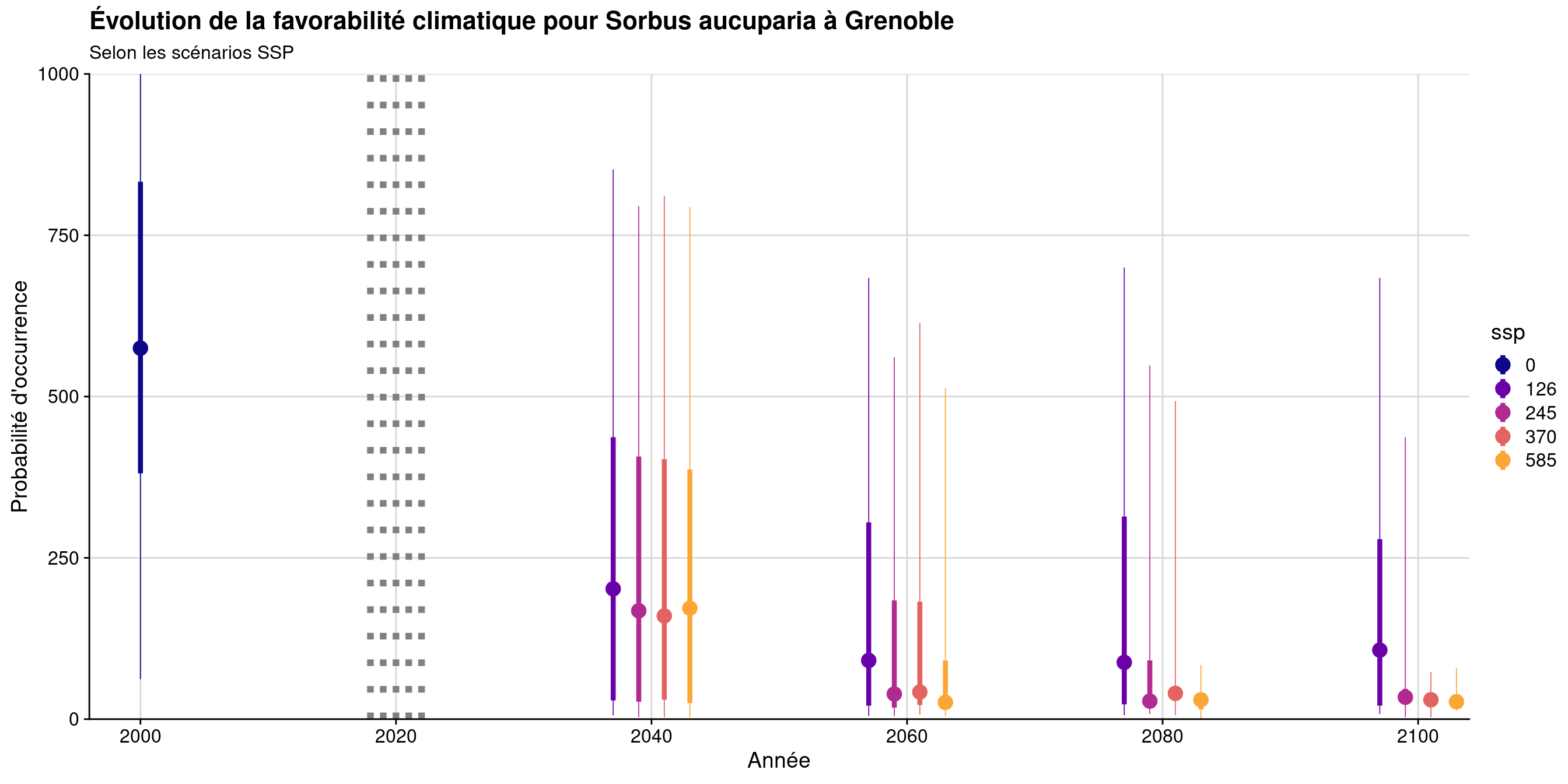
Figure 180.28: Évolution de la favorabilité climatique au cours du XXIème siècle. Le point représente la médiane sur l’étendue de la métropole (i.e. tous les pixels de la carte) ; le trait gras représente 50 % des données intermédiaires (espace inter-quartile) ; le trait fin représente le reste des données du minimum au maximum.